D2 |
Назад |
#Постройте совмещение структур вашего белка и четырех структурных гомологов
В PDBeFold было составлено выравнивание 4q7z с шестью структурами, различающимися по RMSD на 1-2 А: 1smf, 1ql9, 1s82, 4d8n.
Все белки — trypsin-fold протеазы разных млекопитающих (человек, крыса, бык, свинья).
Пространственное выравниевание
Выравнивание этих белков методом Muscle дало схожий результат с пространственным выравниванием. На рис.2 видно, что различие между двумя выравниваниями — сдвиг в одну АА посередине белка. Отличаются они всего в паре мест: в районе 50й, 115й и 175й колонок. Рассмотрим различие около 175й колонки: в пространственном выравнивании Arg-184 из 4q7z находится в "глициновой" колонке, а в Muscle — выделен в отдельную колонку. В то же время в пространственном выравнивании Asp-186 из 4q7z не выровнен и вынесен в отдельную колонку.
В 4q7z Arg-184 находится на самом краю бета-тяжа (как и прочие глицины), а невыровненный Asp-186 — в петле. Петли в белках менее консервативны, чем элементы вторичной структуры, а границы этих элементов определяются неточно. Так что здесь информация о структуре белков не помогает проверить, восстановило ли выравнивание по последовательности эволюционное соответствие разных остатков.
В принципе надо больше верить пространственному выравниванию, поскольку в нём сравниваются объекты, более близкие к реальным белкам, чем аминоксилотные последовательности. Но в данном случае различия двух выравниваний настолько малы, что нельзя наверняка сказать, какое из выравниваний правильное.
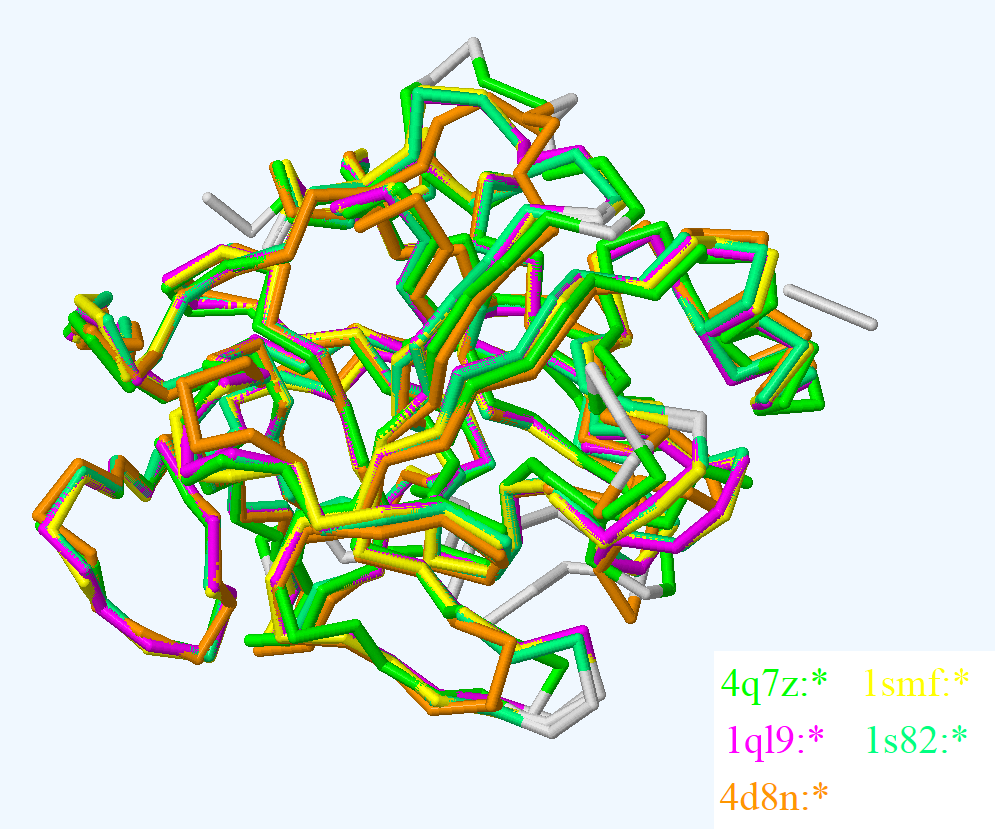
Рис.1 — совмещение четырёх структур с 4q7z
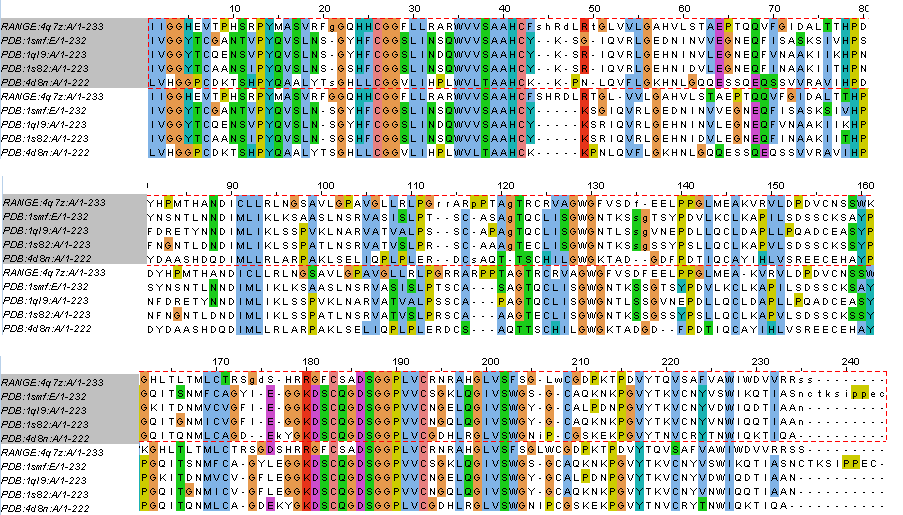
Рис.2 — структурное выравнивание 4q7z и гомологов (выделено серым) и выравнивание методом Muscle (ниже). Разница между двумя множественными выравниваниями — сдвиг на одну АА в средней части белка.
#Используйте поиск по сходству структур в PDBeFold
Без изменения исходных настроек поиск схожих с "1b0m A:203-315" структур не выдаёт полную структуру 1b0m. Если провести поиск, убрав галочку с поля "if no matches within limits of acceptability are found, show close ones", то выдаётся сообщение:
If you are certain that the structures should match, try the following:
decrease the lowest acceptible percent of matching SSEs
change the match criteria.
Если же искать названный участок не во всей PDB, а именно в 1b0m, то выдаётся схожее по духу сообщение:
Detail: ¤ Data on percentage #1 not found.
Видимо, проблема в том, как выставлены проценты сходства запроса и находок.
Так и оказалось: вся структура 1b0m состоит из 754 АА, а запрос — из 134 АА, то есть 15% 1b0m. Чтобы 1b0m выдался в результатах поиска надо поставить % совпадения для запроса — 100%, а для находки — 14%. Это и правда помогло (см.рис.3).
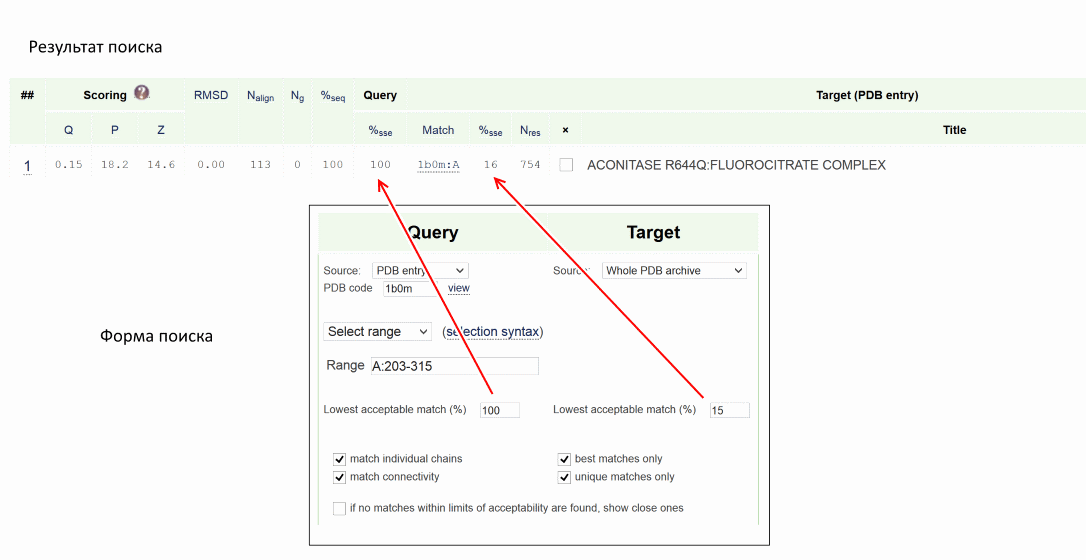
Рис.3 — при правильных настройках поиск фрагмента структуры "1b0m A:203-315" выдаёт саму структуру.
#Совмещение по заданному выравниванию
Для совмещения были выбраны следующие участки структуры 2bnq — E:114-242, D:115-204. (cм.рис.4)
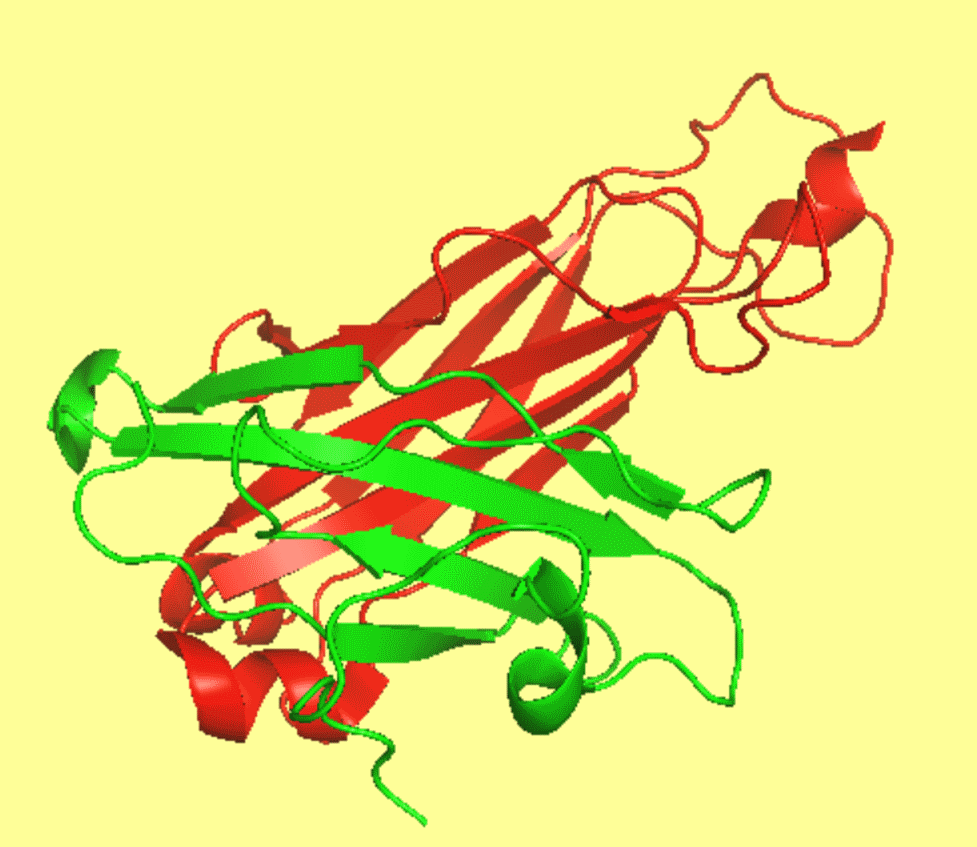
Рис.4 — Зелёным - D:115-204; Красным - Е:114-242
В SheeP были составлены карты бета-листов этих участков (см.рис.5).

Рис.5 — Карты накладываемых участков; в красную рамку выделены выравниваемые участки.
Результат выравнивания по заданным остаткам (скрипт) приведен на рис.6 Из карт SheeP видно, что тяжи практически не идентичны друг другу. В выравнивнивании эти листы тоже совмещаются плохо. В целом сходство топологий слабое.
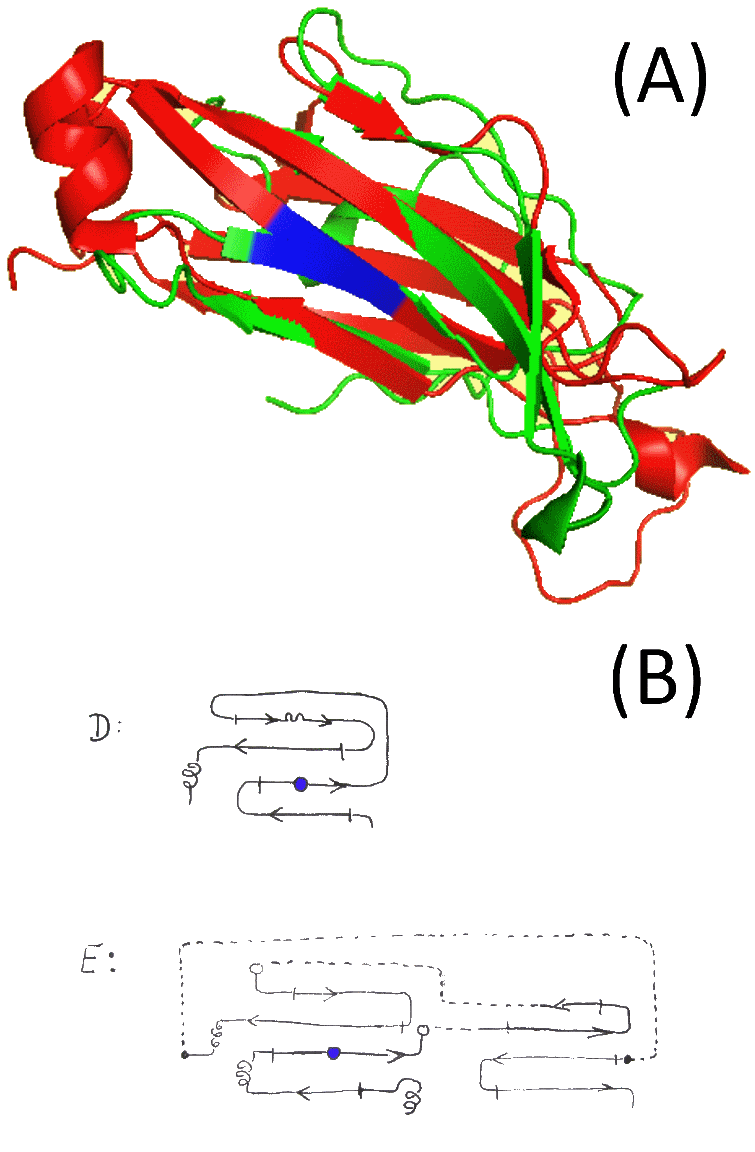
Рис.6 — (А) Совмещение вышеназванных участков; топология двух бета-листов отдалённо схожа; синим покрашены АА из красных рамочек на рис.5;
(B) Схематическое изображение участков вторичной структуры в D- и E-цепях