D6 |
Назад |
#Напишите программу, которая скачивает pdb файл по указанному ID
Прошу: pdbload.py
#Скачайте все последовательности белков, структуры которых определенны при помощи метода ЭМ
Последовательность действия при решении этого задания см.на рис.1
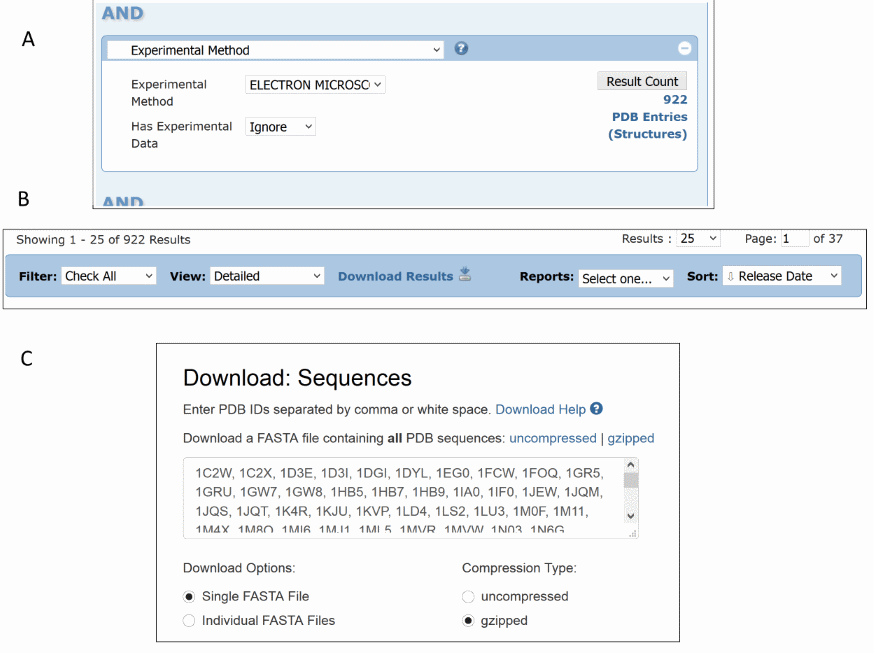
Рис.1 — как скачать FASTA всех структур, найденных при помощи ЭМ:
А) Перейти в "Advanced Search" меню поиска структур; выбрать критерии поиска ("Search criteria"). Альтернатива — провести обычный поиск, а потом ограничить результаты только како-либо из предложенных категорий.
В) В верху списка выданных результатов нажать на кнопку "Download results".
С) В появившемся окне выбрать, в каком виде скачать результаты.
#Сравните список структурных гомологов PDBeFold со списком jFATCAT на странице 1DGK в PDB
На cтранице PDB приведена только cсылка на структуру 1CZA, определённую тем же набором авторов в том же 1999 году. Рядом с ссылкой приписано, что собственно для 1DGK поиск структурных гомологов, но вот 1СZA на 95% идентичен по структуре и поэтому может использоваться для поиска гомологов 1DGK.
Все белки-гомологи 1CZA — гексокиназы разных организмов: как про-, так и эукариот. Всего 30-37 штук, в зависимости от того, какой из четырёх доменов 1CZA выбрать. Результатов для всего белка целиком не предложено.
Поиск в PDBeFold со стандартными настройками выдаёт 21 результат. В этом списке почти все белки — человеческие гексокиназы, с разными мутациями или лигандами. Провести поиск по отдельным доменам в PDB и сравнить результат у меня не хватило терпения — поиск идёт слишком долго.
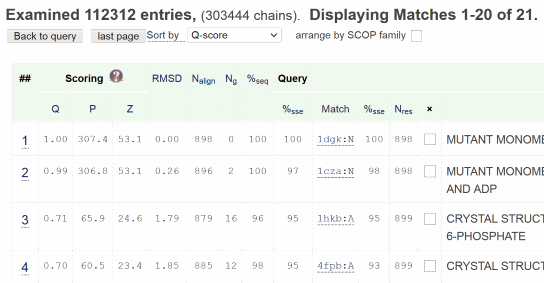
Рис.2 — часть выдачи PDBeFold