| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Создание репрезентативной выборки гомологов белка MRGA_BACSU
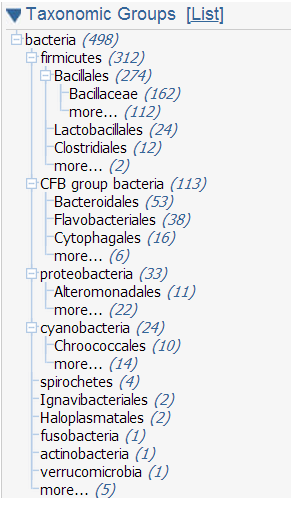
Сначала был определен филум организма, которому принадлежит белок MRGA_BACSU. Это FIRMINICUTES. Затем был осуществлен поиск гомологов с помощью BLAST. Были использованы следующие параметры: максимальное количество хитов - 5000, значение E_value = 0.00001. В поле Organism введем название филума - Firminicutes и исключаем его, ставя галочку в поле Exclude. Аналогичным образом исключаем Eukaryota. Таким образом, мы ищем гомологи белка в других филумах Bacteria. В качестве банка данных используем RefSeq. В секции Descriptions окна выдачи BLAST выберем все последовательности (Select: All) и получим о них выдачу GenBank. Щелкнем справа в окошке на "Tree" и посмотрим, к каким таксономическим группам организмов принадлежат хиты. Поскольку не удалось загрузить все полученные результаты, то я использовала максимальное количество хитов = 500. В таблице 1 приведено описание параметров BLAST, используемых для поиска гомологов. В таблице 2 приведены результаты этого поиска (филумы и названия организмов). Т.к. для выборки использовалось количество хитов меньшее, чем то, которое описывает все варианты, я приведу дополнение к таблице 2 (эти последовательности не вошли в fasta-file):
-филум Verrucomicrobia (Chlamydiae); организм Coraliomargarita akajimensis DSM 45221;
-филум Tenericutes; организм Acholeplasma laidlawii PG-8A;
-филум Planctomycetes; организм Blastopirellula marina DSM 3645;
-филум Euryarchaeota; организм Natrinema sp. J7-2;
-филум Thaumarchaeota; организм Candidatus Nitrososphaera gargensis Ga9.2;
-филум Deinococcus-Thermus; организм Deinococcus proteolyticus MRP;
Само дерево представлено на рисунке ниже:
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | blastp (protein-protein BLAST) | Reference proteins (refseq_protein) | Exclude Firmicutes; exclude Eukaryota | 0.00001 | 2169 (ПОРОГ 5000) |
| По эукариотам | blastp (protein-protein BLAST) | Reference proteins (refseq_protein) | Eukaryota | 0.00001 | 3 |
Таблица 1. Описание параметров BLAST, используемых для поиска гомологов белка MRGA_BACSU.
| Домен | Филум/Царство | Название организма | Количество белков (в конкретной выборке) |
| Archaea | Thaumarchaeota | Candidatus Nitrososphaera gargensis Ga9.21 | 1 |
| Bacteria | Actinobacteria | Slackia sp. CM382Б Atopobium vaginae DSM 15829 Atopobium vaginae PB189-T1-4 |
1 1 1 |
| CFB group bacteria | Cesiribacter andamanensis AMV16 Formosa sp. AK20 |
1 1 |
|
| Cyanobacteria | Xenococcus sp. PCC 7305 Gloeocapsa sp. PCC 73106 |
1 1 |
|
| Fusobacteria | Streptobacillus moniliformis DSM 12112 Sebaldella termitidis ATCC 33386 Leptotrichia buccalis C-1013-b |
1 1 1 |
|
| Ignavibacteria | Melioribacter roseus P3M Ignavibacterium album JCM 16511 |
1 1 |
|
| Proteobacteria | Marinobacter santoriniensis NKSG1 Edwardsiella tarda C07-087 Serratia marcescens WW4 |
1 1 1 |
|
| Spirochaetes | Spirochaeta thermophila DSM 6578 Spirochaeta africana DSM 8902 Spirochaeta caldaria DSM 7334 |
1 1 1 |
|
| unclassified Bacteria | Haloplasma contractile SSD-17B | 2 | |
| Eukaryota | Green plants | Oryza sativa Indica Group Ricinus communis |
1 1 |
| Metazoa | Bos taurus | 1 |
Таблица 2. Встречаемость белков, гомологичных MRGA_BACSU в различных таксонах. Т.к. сервер не смог обработать все результаты, было поставлено ограничение на 500.
Множественное выравнивание гомологов белка MRGA_BACSU
В результате выборки гомологичных последовательностей был получен файл в fasta-формате, который их содержит: файл. Ниже, на рис.1. представлено множественное выравнивае полученных гомологичных последовательностей с исходным белком.
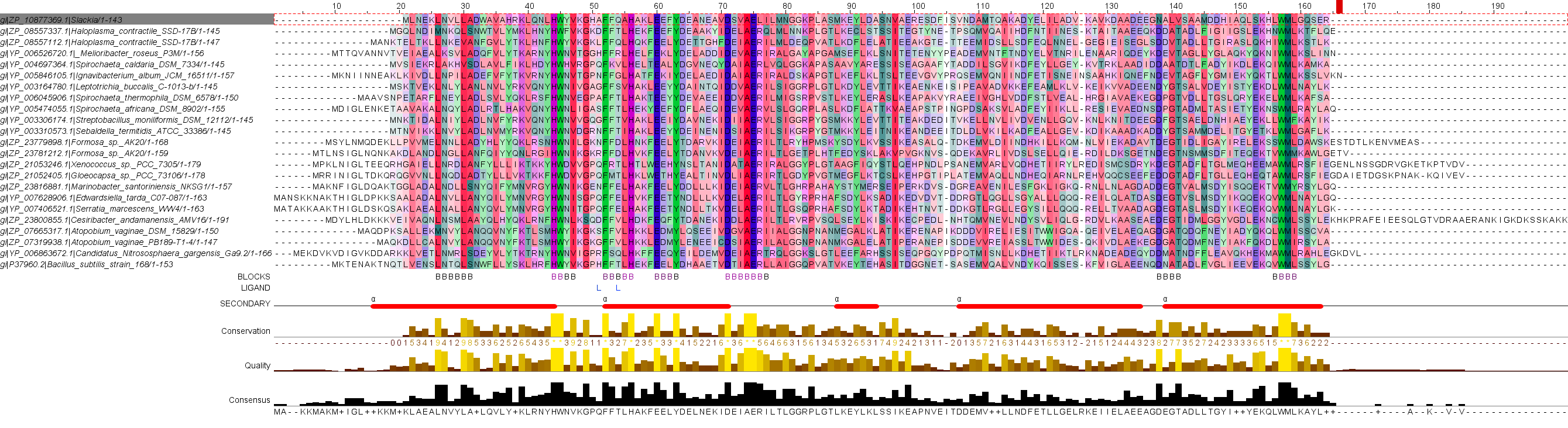
Рис.1.Множественное выравнивание последовательностей белка MRGA_BACSU и его гомологов. Красным цветом выделены гидрофобные аминокислоты, синим - отрицательно заряженные, фиолетовым - положительно заряженные, зеленым - с ароматическим радикалом, темно-бирюзовым - полярные незаряженные. Conservation Colour Increment - 15%.
Annotations - разметка выравнивания, которая позволяет добавлять информацию об особенностях выравнивания. В частности, на данном изображении это остатки, связывающие лиганд (строка аннотации - LIGAND, сами остатки помечены - "L"); блоки - участки с наиболее удачным выравниванием (строка аннотации - BLOCKS, сами блоки помечены - "B"); элементы вторичной структуры, в частности, альфа-спирали (строка аннотации - SECONDARY, альфа-спирали - α и выделены красным).
Результаты анализа множественного выравнивания гомологов белка MRGA_BACSU
Консервативность выравнивания в целом достаточно высока, гомологи были отобраны удачно и мало отличаются от исходного белка (присутствуют всего 2 колонки гэпов.) Хуже всего получилось выравнивание на концах последовательностей (они разной длины). Кроме того, элементы вторичной структуры (α-спирали) не обладают хорошей консервативностью, лишь некоторые аминокислотные остатки являются общими для большинства гомологов. Что касается остатков, связвающих лиганд, то тут сложно предположить то, что и у гомологов будет происходить связывание этого же лиганда, т.к. треонин хотя и встречается в почти половине гомологов, гистидин практически не встречается. Блоки выбирались по принципу наибольшей удачности выравнивания.
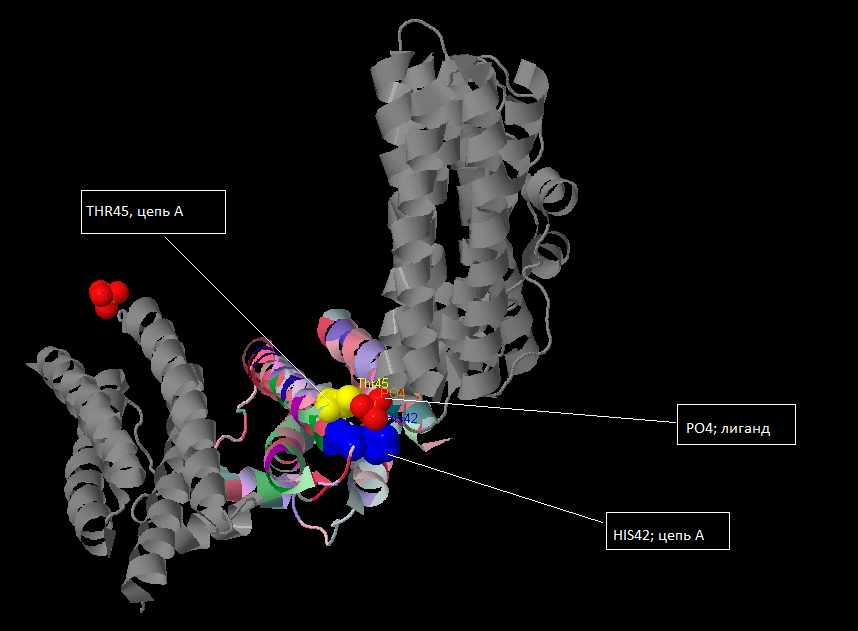
Рис.2. Структура белка MRGA_BACSU, цепь А покрашена в соответствии с консервативностью остатков.