| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Белок mrga_bacsu в Uniprot
1. Особенности (features) белка
Особенность состоит в наличии элементов вторичной структуры - альфа-спиралей. Их последовательности приведены ниже.

Helix1:
KTNQTLVENS LNTQLSNWFL LYSKLHRFHW Y
Helix2:
FFTLHEKFEE LYDHAAETVD TIAERLLA
Helix3:
VKEYTEH
Helix4:
ASEMVQALVN DYKQISSESK FVIGLAEE
Helix5:
NATADLFVGL IEEVEKQVWM LSSY
2. Описание всех записей банка трехмерных структур PDB, относящихся к белку mrga_bacsu.
Базу данных PDB найдем так: Protein function, structure and interaction databases/Protein structure databases/PDB. Нажимаем на Search. Находится всего одна стркутура с идентификатором - 2CHP. Ставим галочку и выбираем save results. Отмечаем File(txt); Number of entries to download - 10; save with view - PDBShortView.
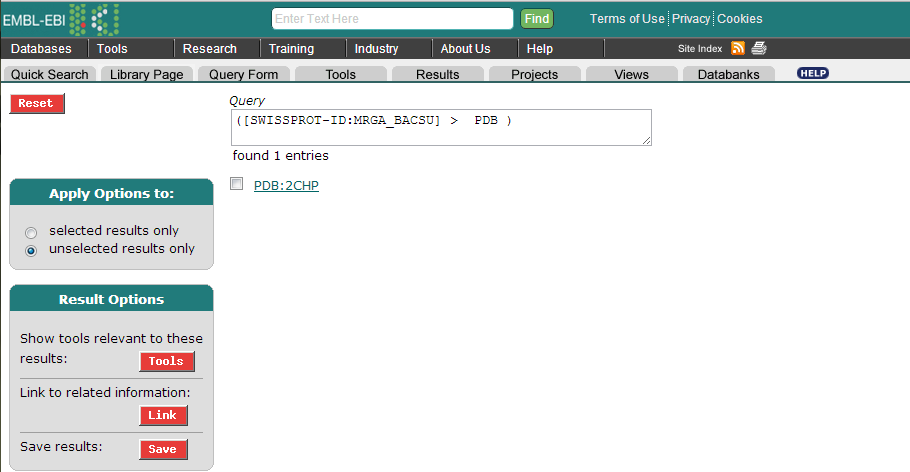
Запишем иноформацию о последовательности в файл. Он будет содержать информацию о единственной структуре в PDB белка MRGA с идентификатором - 2CHP в виде PDBShortView
Файл с информацией о последовательности
3. Полноразмерные белки из Firmicutes, выполняющие функцию, сходную с функцией белка MRGA.
В поле Taxonomy - записываем Firmicutes, в поле Comments: Comment - функцию белка, отделяя слова символом &. Чтобы выдавались только полноразмерные последовательности, в поле Description запишем *!fragment. В случае не выдачи информации по запросу, можно использовать ссылку i слева от строки запроса, в которой выбрано поле для поиска. Если в открывшееся окошко List values that match ввести искомое слово, и, при необходимости, "*" на конце, то можно получить полную информацию о его встречаемости в данном поле в данном банке.
| Формулировка функции белка | Строка запроса | Количество найденных документов |
| DNA binding (связывание с ДНК) | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:DNA*] & [swissprot-Comment:binding*]) > parent )) | 675 |
| DNA binding | (([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:DNA*] & [swissprot-Comment:binding*]) > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 670 (полноразмерные) |
| Oxidoreductase activity (катализ реакций окисления) | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:Oxidoreductase*] & [swissprot-Comment:activity*]) > parent )) | 6 |
| Oxidoreductase activity | (([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:oxidoreductase*] & [swissprot-Comment:activity*]) > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 6 (полноразмерные) |
| Response to stress (ответная реакция на стресс) | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:Response*] & [swissprot-Comment:stress*]) > parent )) | 598 |
| Response to stress | (([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:response*] & [swissprot-Comment:stress*]) > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 590 (полноразмерные) |
4. Полноразмерные последовательности найденных белков в fasta формате
Отметим 10 последовательностей:
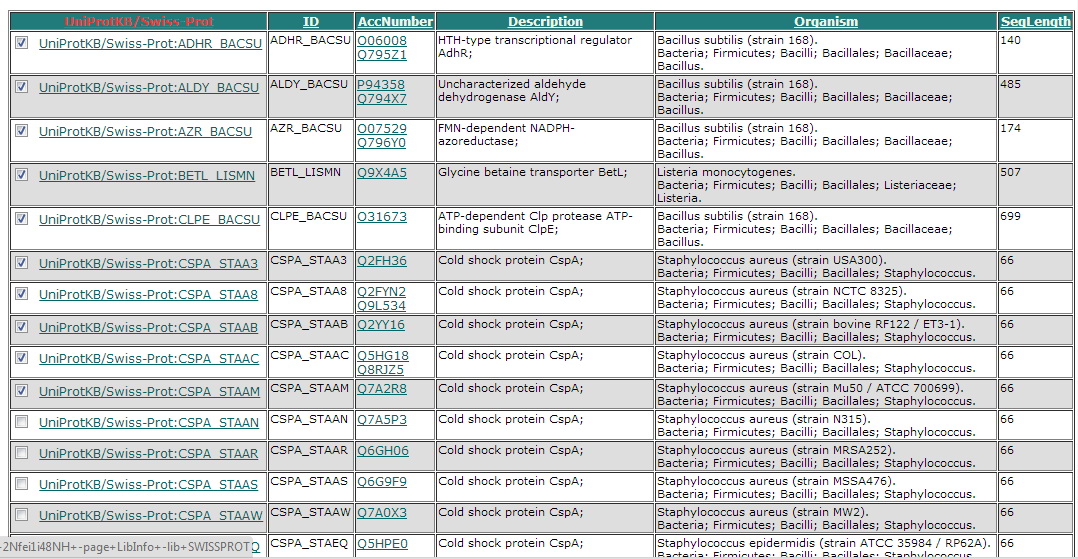
Сохраним их в fasta-формате: отметим поле File (txt), в поле Save with view - FastaSeqs.
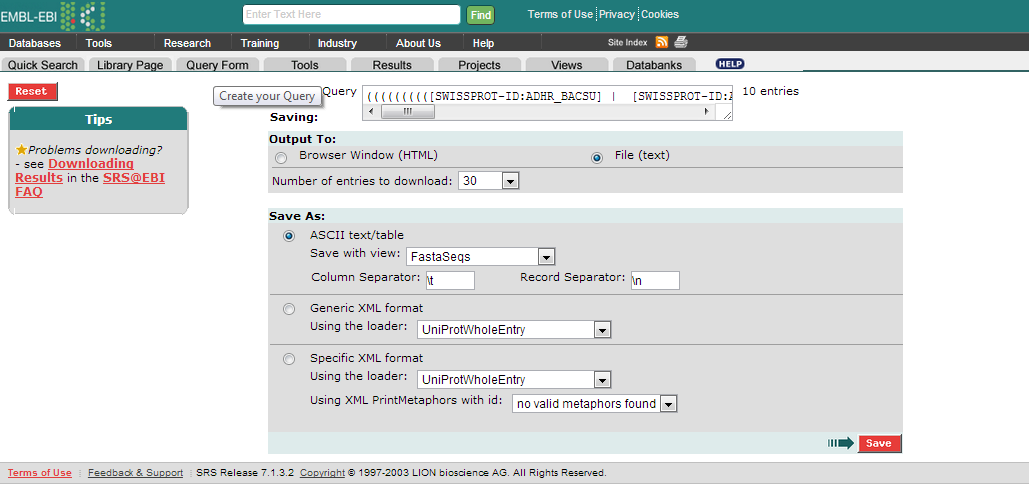
Полученный файл:
Файл
5. Cписок последовательностей из задания 4, в котором указаны ID, AC последовательностей, организм (Species), название белка (Description) и длина последовательности
В разделе Create a view выберем следующие поля: ID, AccessionNumber, Description, Species, Sequence Length. Нажимаем Search.
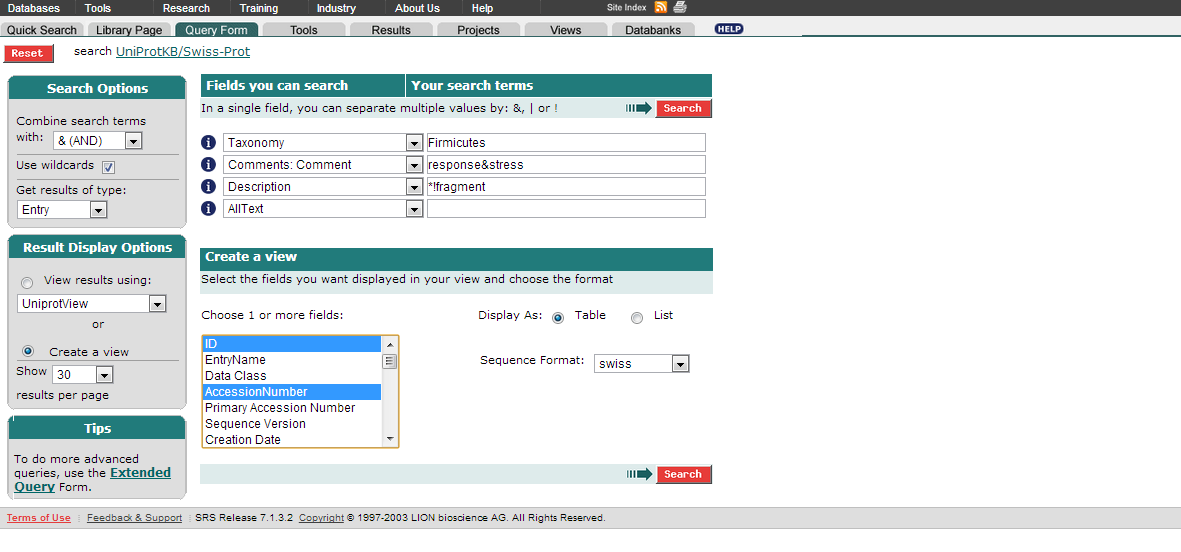
Отмечаем последовательности, нажимаем Save. Выбираем File (txt), в поле Save with view ставим SWISSPROT1.
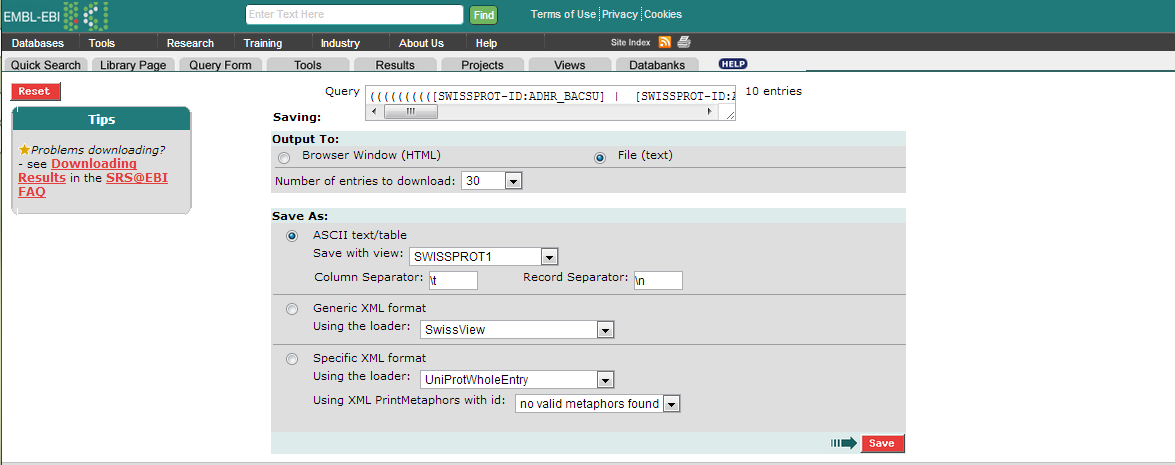
Сохраняем файл с расширением xls.
Файл
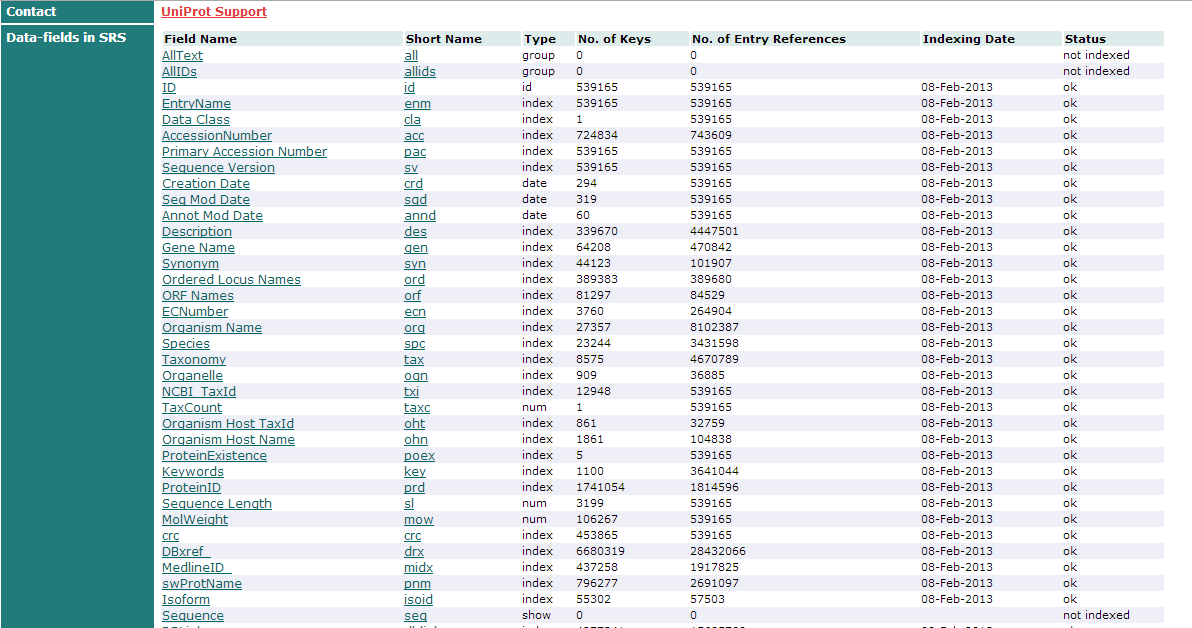
6.Поиск и изучение страницы с описанием поля "Taxonomy" банка Swissprot
Зайдем на Library Page, нажме на UniProtKB/Swiss-Prot. Откроется страница с описанием банка. В нижней части этой страницы имеется список полей; названия полей оформлены как гиперссылки на их описания. Перейдем на Taxonomy.
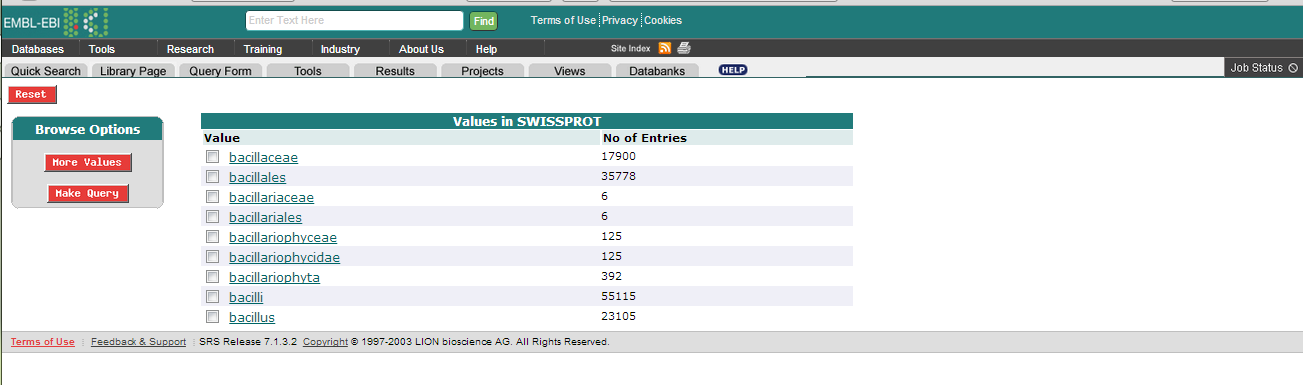
Названия каких таксонов начинаются на "bacil"?
Для ответа на этот вопрос в поле List values that match введем bacil*. Получим список таксонов, которые начинаются на bacil:
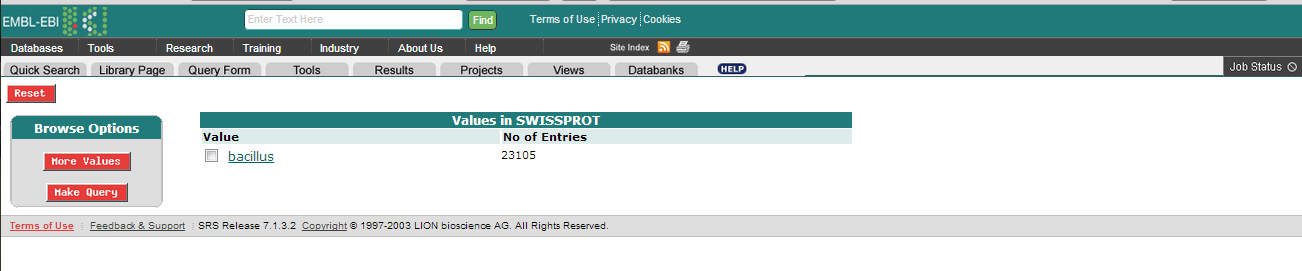
Сколько в банке Swissprot записей, описывающих белки из рода Bacillus?
Для ответа на этот вопрос в поле List values that match введем Bacillus. Получим количество записей - 23105.
Сколько в банке Swissprot записей, описывающих белки из отдела Firmicutes?
Для ответа на этот вопрос в поле List values that match введем Firmicutes. Получим количество записей - 68204.
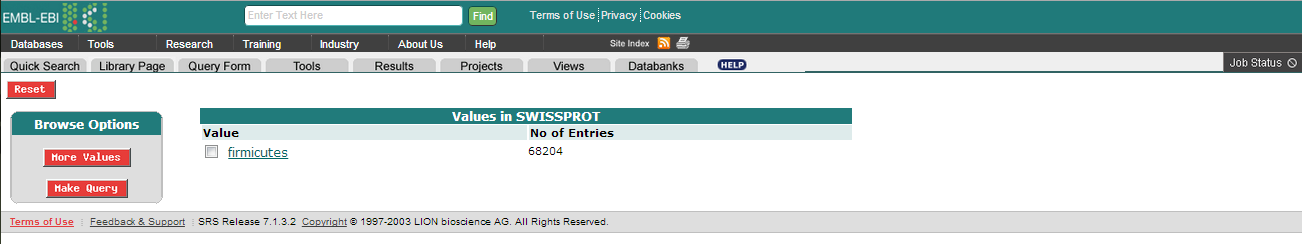
7. Поиск записей SwissProt , связанных с научными интересами известного российского (советского)биолога А.А.Нейфаха
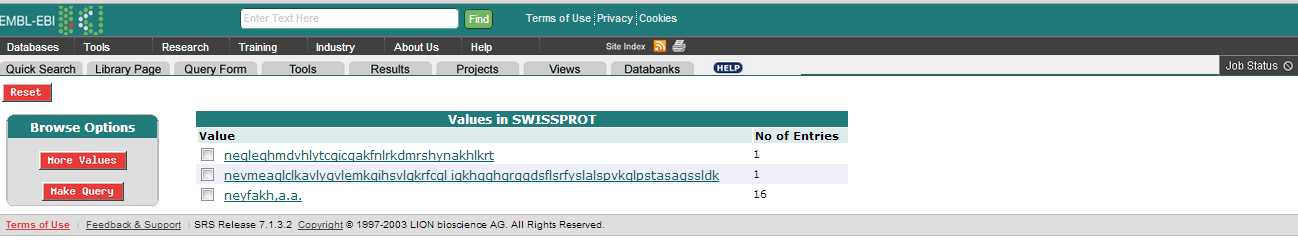
Для начала найдем, как правильно записывается фамилия ученого на английском языке. Для этого в Library Page выберем UniProtKB/Swiss-Prot, затем выберем AllText. В поле List values that match введем Ne*f*kh*. Получим результат - фамилия записывается так:neyfakh,a.a.
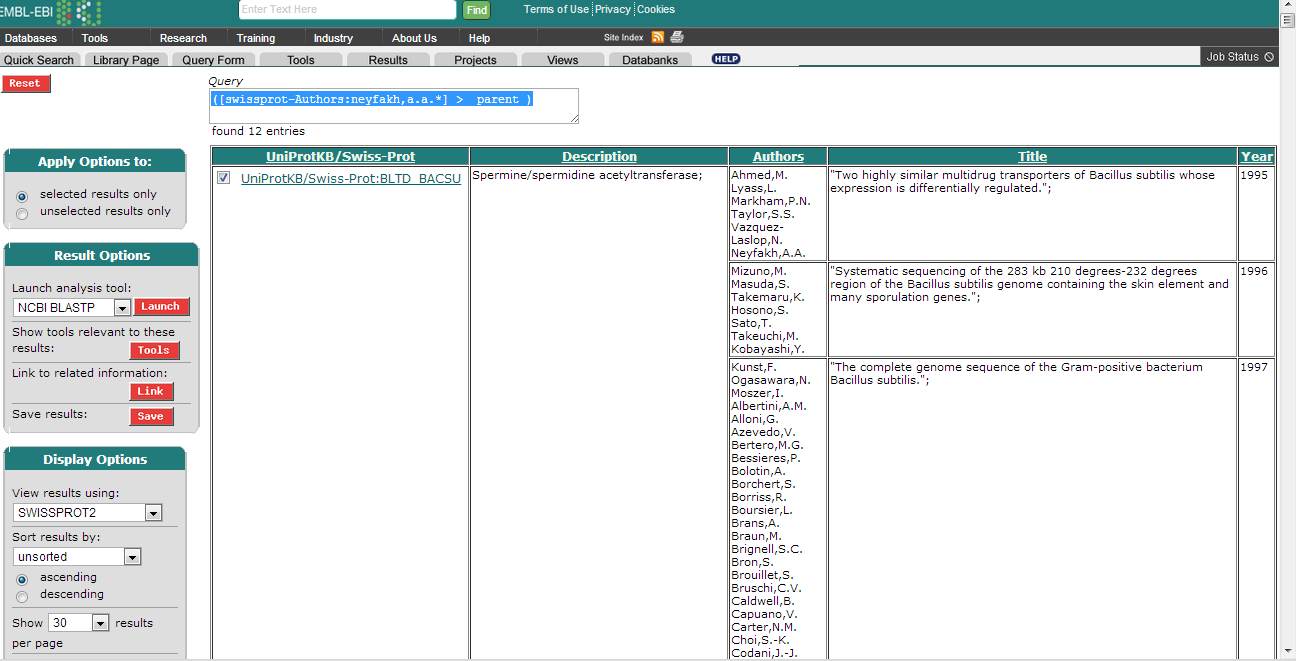
Перейдем на Query Form. Составим запрос: выберем References: Authors. Запишем фамилию ученого: neyfakh,a.a. В поле Create of view выбираем поля, которые нужно увидеть в выдаче: Description, References: Authors, References: Title и References: Year. Среди выданных результатов отметим те, которые нам нужны (где Нейфах является автором первой статьи).
Сохраним табличку Excel, включающую описание белка; авторов, название и год публикации.
Полученный файл:
Таблица
Запрос, с помощью которого была получена таблица: ([swissprot-Authors:neyfakh,a.a.*] > parent ). (были исключены элементы, которые нам не подходили вручную)
Судя по названию статей, мы можем предположить, что ученый занимался изучением бактерии Bacillus Subtilis: изучал механизмы лекрственной устойчивости и регуляцию экспрессии соответствующих белков.