| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
В соответствии с моделью Дж. Уотсона и Ф. Крика, предложенной в 1953 г. на основании ряда аналитических данных, а также рентгеноструктурного анализа молекула ДНК состоит из двух цепей, образуя правовращающую спираль, в которую обе полинуклеотидные цепи закручены вокруг одной и той же оси. Удерживаются цепи благодаря водородным связям, образующимся между их азотистыми основаниями. Необходимо указать, что конфигурация двойной спирали ДНК сильно меняется в зависимости от количественного содержания воды и ионной силы окружающей среды. Методами рентгеноструктурного анализа доказано существование по крайней мере 6 форм ДНК, названных А-, В-, С-, D-, Е- и Z-формами. В данном разделе мы рассмотрим A-, B-, Z-формы. Также мы рассмотрим некоторые аспекты структуры тРНК. Относительно вторичной структуры тРНК наиболее вероятной представляется модель, предложенная Р. Холли, плоское изображение которой напоминает клеверный лист.[1]
Задание 1. Построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
Для начала введем в командую строку следующее:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
С помощью программы fiber пакета 3DNA построим A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc". Для этого используем следующие команды:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Полученные файлы: gatc-a.pdb, gatc-b.pdb, gatc-z.pdb.
Задание 2.
Упр.1. Научиться выделять разные атомы и химические группировки, используя предопределенные множества JMol.
Будем использовать файл с А-формой.
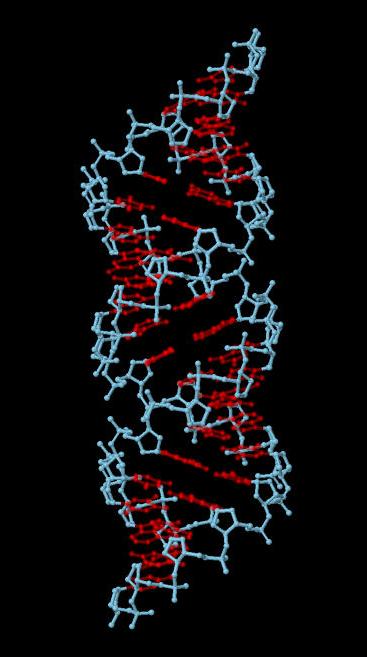 Рис.1. А-форма ДНК. Основания показаны красным цветом, сахарофосфатный остов - голубым. |
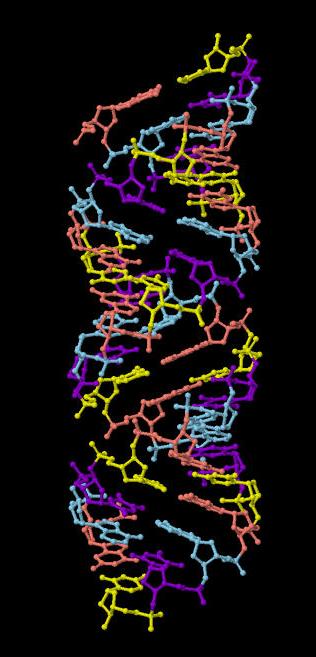 Рис.2. Нуклеотиды А-формы ДНК. Голубым цветом показан дезоксиаденозин-5-фосфат, фиолетовым - дезокситимидин-5-фосфат, розовым- дезоксигуанозин-5-фосфат, желтым - дезоксицитидин-5-фосфат. |
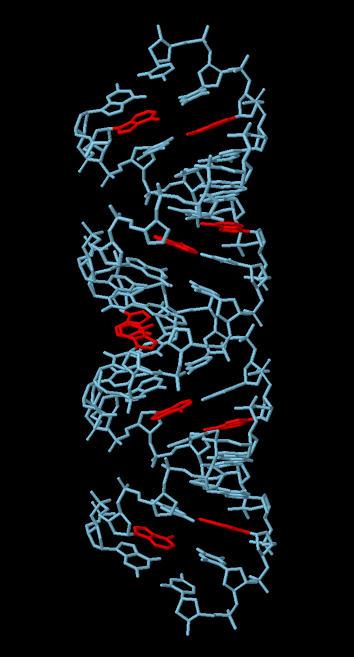 Рис.3. А-форма ДНК. Аденины выделены красным цветом. |
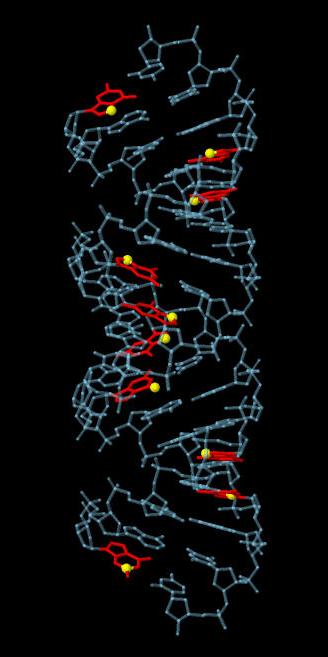 Рис.4. A-форма ДНК. Выделены все гуанины красным цветом и отдельно в каждом показан желтым цветом атом N7. |
Упр.2.
Полученные файлы: 1I9V.pdb и 1MDM.pdb.
Упр.3.
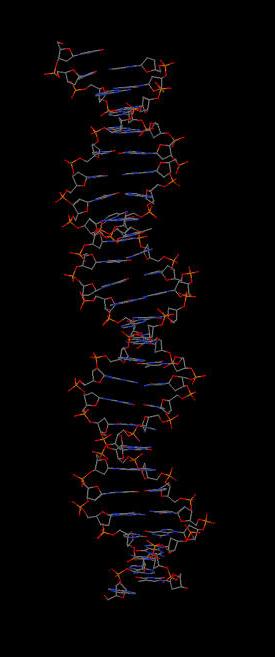
Рис.4. ДНК-белковый комплекс (INHIBITED FRAGMENT OF ETS-1 AND PAIRED DOMAIN OF PAX5 BOUND TO DNA) с идентификатором 1MDM.
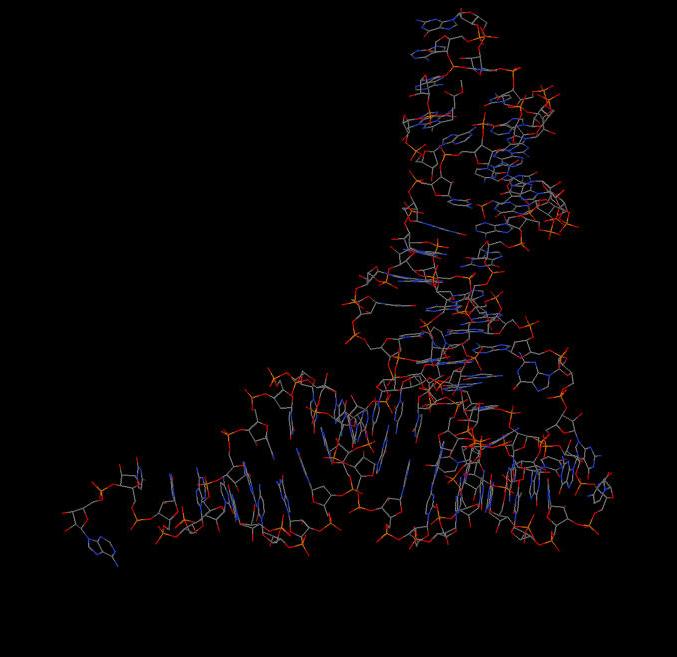
Рис.5. тРНК (TRNA-NEOMYCIN COMPLEX) с идентификатором 1I9V.
Файлы с координатами: dna.pdb и rna.pdb
Заданные структуры ДНК и РНК разрывов не имеют.
Задание 3. Сравнение структур 3-х форм ДНК с помощью средств JMol.
Упр.1.
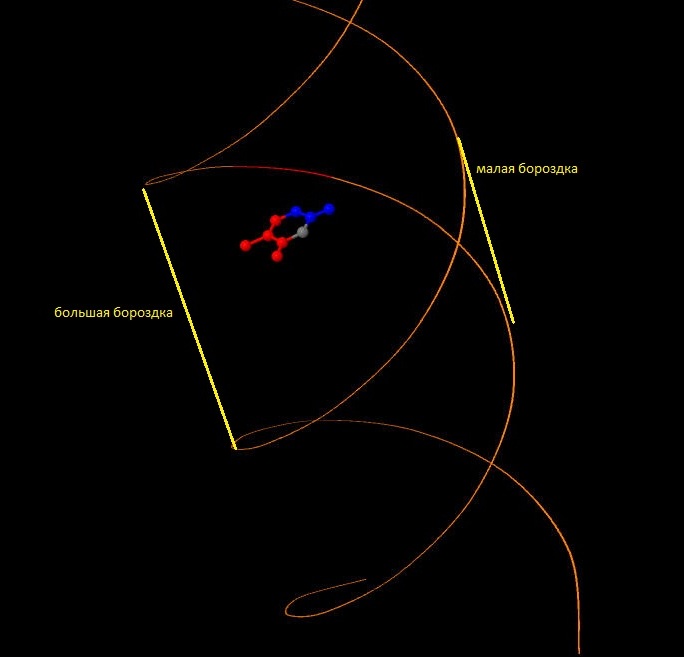
Рис.6. B-форма ДНК. Показан тимин11. Красным отмечены атомы, напрвленные в сторону большой бороздки, синим - в сторону малой.
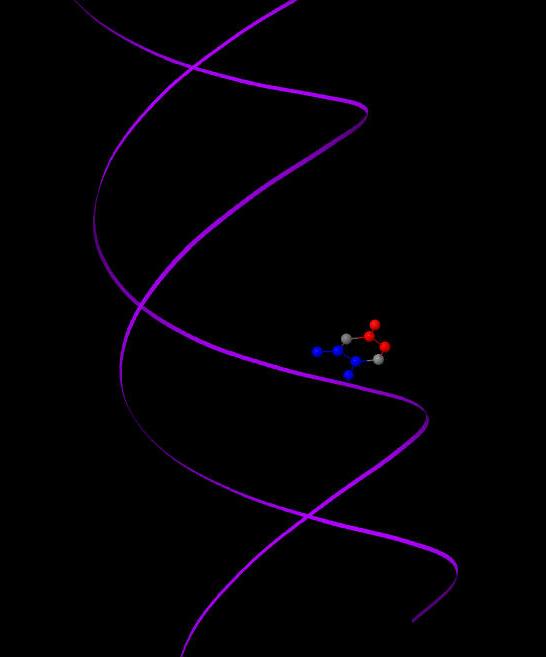
Рис.7. A-форма ДНК. Показан тимин35. Красным отмечены атомы, напрвленные в сторону большой бороздки, синим - в сторону малой.
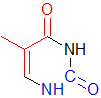
Рис.8. Тимин. Красным отмечены атомы, напрвленные в сторону большой бороздки, синим - в сторону малой.
В сторону большой бороздки обращены атомы T11.O4, T11.C5m, T11.C5, T11.C4.
В сторону малой бороздки обращены атомы T11.N1, T11.O2, T11.C2.
Остальные атомы основания - T11.N3, T11.C6 не относятся к бороздкам.
В Z-форме нет тиминов, в А-форме все наоборот: T11.O4, T11.C5m, T11.C5, T11.C4 к малой бороздке, а T11.N1, T11.O2, T11.C2 к большой.
Упр2.
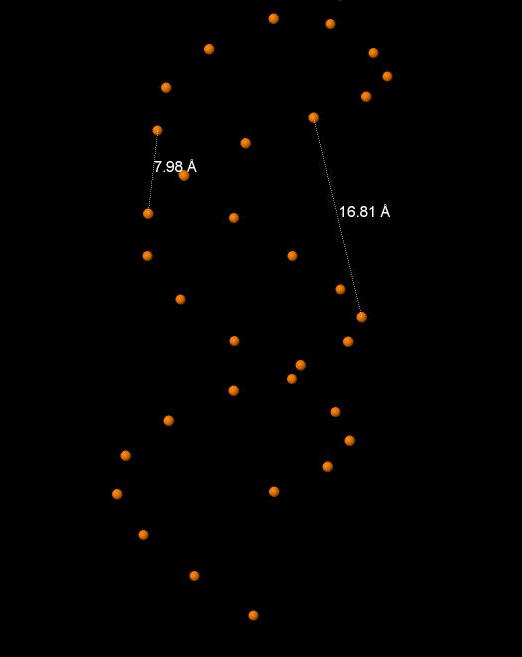
Рис.9. А-форма ДНК. Показаны значения ширины малой и большой бороздок.
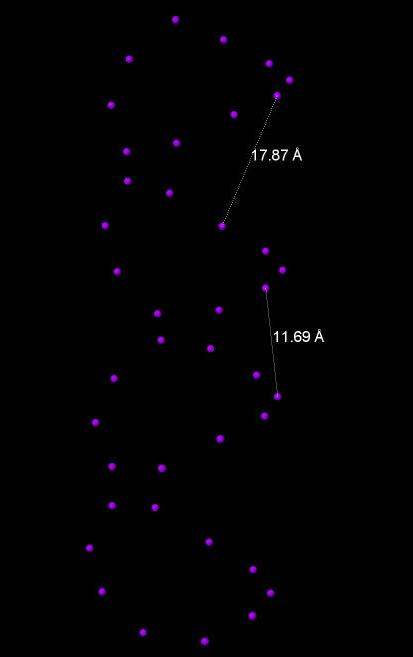
Рис.9. B-форма ДНК. Показаны значения ширины малой и большой бороздок.
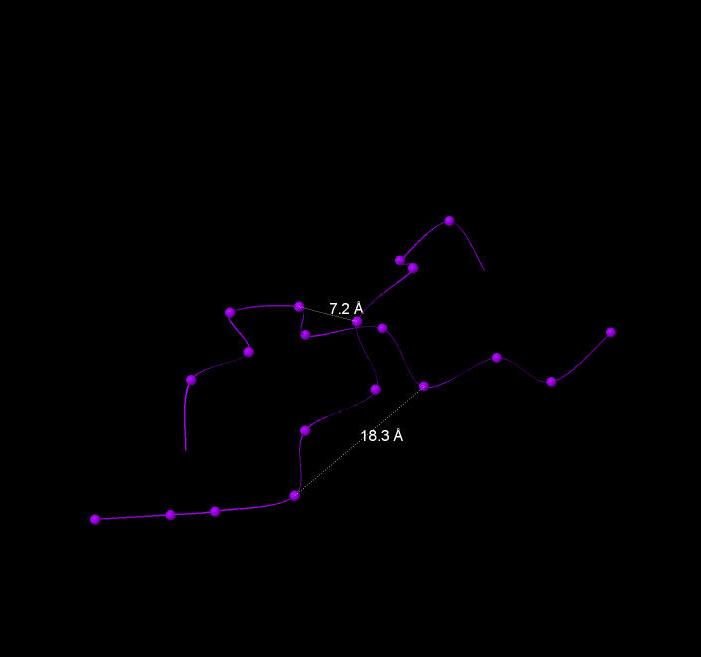
Рис.10. Z-форма ДНК. Показаны значения ширины малой и большой бороздок.
Сравнение основных спиральных параметров разных форм ДНК.
|
A-форма |
B-форма |
*Z-форма |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (A) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.81 | 18.87 | 18.3 |
| Ширина малой бороздки | 7.98 | 11.69 | 7.2 |
Упр.3
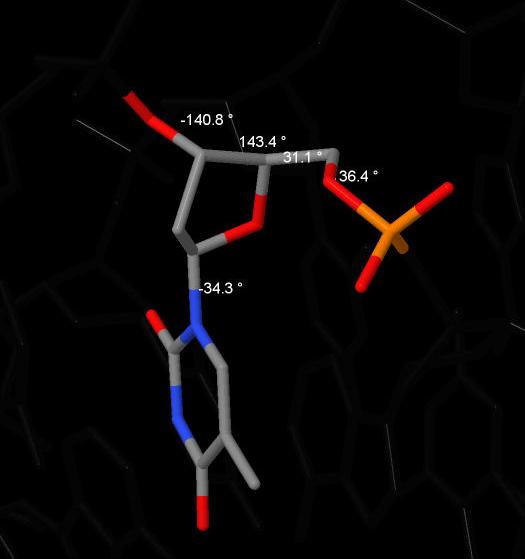
Рис.11. Некоторые торсионные углы Тимина11.
| Значения торсионных углов | |||||||
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) | |
| A-ДНК | -64.1 | 174.8 | 41.7 | 79.1 | 147.8 | -75.1 | -157.2 |
| В-ДНК | -35.2 | 136.4 | 31.1 | 143.4 | 140.8 | -160.5 | -34.3 |
| Значения торсионных углов из презентации | |||||||
| А-ДНК | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| В-ДНК | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Т.к. в некоторых случаях отличие в торсионных углах довольно существенное, можно предположить, что это связано с тем, что в таблице для сравнения приведены данные для другого нуклеотида.
Задание 4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Упр.1.
Так как пакет 3DNA работает только со старым форматом .pdb, используем программу remediator для перевода файлов 2dxi.pdb и 1by4.pdb в старый формат:
remediator --old "XXXX.pdb" > "XXXX_old.pdb"
Для каждого из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb была выполнена следующая команда:
find_pair -t gatc-x.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров. В файлах gatc-a.out, gatc-b.out и gatc-z.out можно найти значения торсионных углов. Для удобства значения торсионных углов для рассматриваемых структур приведены в файле torsion_abz.xlsx.
Если сравнить значения соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК, то в наибольшей степени различаются значения следующих углов: у A- и B-форм - ξ;δ и χ;у A- и Z-форм - α, ξ и χ, у B- и Z-форм - ξ; χ
.
Значения торсионных углов в структуре тРНК для удобства приведены в файле torsion_rna.xlsx. Данная структура больше всего похожа на A-форму ДНК.
Значения торсионных углов для ДНК приведены в файле torsion_dna.xlsx. C помощью алгебраических функций, таких как среднее значение по углам без учета крайних нуклеотидов, разница между полученным средним и углами и суммарное отклонение. Таким образом, самый деформированный нуклеотид по значению торсионных углов - G3 из цепи D.
Изучение вторичной структуры тРНК.
Упр.2.
Рассмотрим водородные связи в структуре тРНК и на основе этого попробуем определить особенности вторичной структуры. Для этого используем файл rna1_old.out
.
Strand I Strand II
1 (0.010) A:...1_:[..G]G-----C[..C]:..72_:A (0.013)
2 (0.011) A:...2_:[..C]C-----G[..G]:..71_:A (0.010)
3 (0.018) A:...3_:[..G]G-----C[..C]:..70_:A (0.008)
4 (0.014) A:...4_:[..G]G-*---U[..U]:..69_:A (0.006)
5 (0.018) A:...5_:[..A]A-----U[..U]:..68_:A (0.008)
6 (0.009) A:...6_:[..U]U-----A[..A]:..67_:A (0.006)
7 (0.013) A:...7_:[..U]Ux----A[..A]:..66_:A (0.017)
Акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72.8 (0.017) A:..49_:[..C]C-----G[..G]:..65_:A (0.021) 9 (0.009) A:..50_:[..U]U-----A[..A]:..64_:A (0.012) 10 (0.013) A:..51_:[..G]G-----C[..C]:..63_:A (0.010) 11 (0.007) A:..52_:[..U]U-----A[..A]:..62_:A (0.023)T-стебель состоит из участка 49-52 и комплементарного ему 62-65.
16 (0.014) A:..40_:[..C]C-----G[..G]:..30_:A (0.015) 17 (0.008) A:..41_:[..U]U-----A[..A]:..29_:A (0.014) 18 (0.008) A:..42_:[..G]G-----C[..C]:..28_:A (0.009) 19 (0.011) A:..43_:[..G]G-----C[..C]:..27_:A (0.014)Антикодоновый стебель состоит из участка 40-43 и комплементарного ему 27-30.
21 (0.005) A:..10_:[..G]G-----C[..C]:..25_:A (0.004) 22 (0.021) A:..11_:[..C]C-----G[..G]:..24_:A (0.014) 23 (0.019) A:..12_:[..U]U-----A[..A]:..23_:A (0.013) 24 (0.011) A:..13_:[..C]C----xG[..G]:..22_:A (0.020)D-стебель состоит из участка 10-13 и комплементарного ему 22-25. Всего 7 неканонических пар. Пример неканонической пары:
A:...4_:[..G]G-*---U[..U]:..69_:A,т.е. G4-U69. Найдем дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру. Для этого рассмотрим комплементарные пары, не имеющие отношения к стеблям.
19_:[..G]G-----C[..C]:..56То есть сюда относится пара G19-G56.
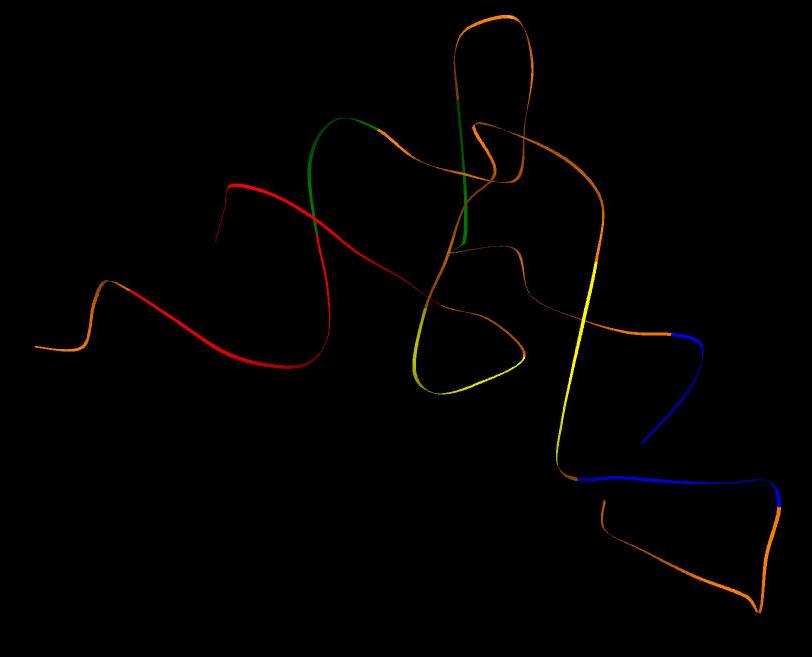
Рис.12. Вторичная структура тРНК. Красным цветом показан акцепторный стебель, зеленым - T-стебель, желтым - D-стебель, синим - антикодоновый стебель.
Упр.3.Возможные стекинг-взаимодействия.
Стэкинг взаимодействия - взаимодействия между основаниями, уложенными одно над другим. В них участвуют силы Ван-дер-Ваальса и диполь-дипольные взаимодействия.
Используем файл rna1_old.out с характеристикой структуры тРНК. Найдем данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований. Для пар с наибольшими значениями получим стандартное изображение стекинг-взаимодействия. Будем использовать следующие команды:
ex_str -8 stacking.pdb step8.pdb
stack2img -cdolt step8.pdb step8.ps
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GC/GC 3.98( 1.77) 0.00( 0.00) 0.00( 0.00) 6.04( 2.90) 10.02( 4.67) 10 GU/AC 7.09( 4.53) 0.00( 0.00) 0.00( 0.00) 3.04( 1.53) 10.13( 6.06) 12 Gu/AC 8.29( 3.00) 0.00( 0.00) 0.00( 0.00) 3.56( 0.99) 11.85( 3.99) 21 GC/GC 2.48( 0.13) 0.00( 0.00) 0.00( 0.00) 7.37( 3.95) 9.85( 4.09)
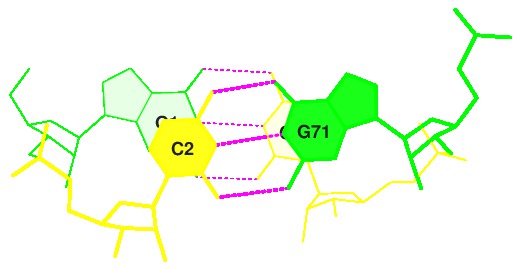 Рис.14. Стэкинг-взаимодействие GC/GC. (step1) |
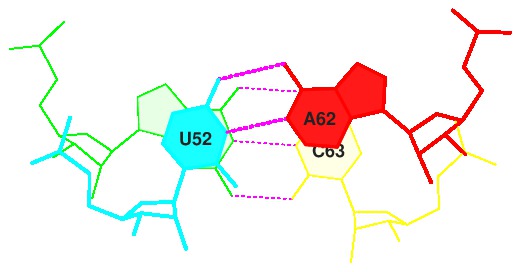 Рис.15. Стекинг-взаимодействие GU/AC. (step10) |
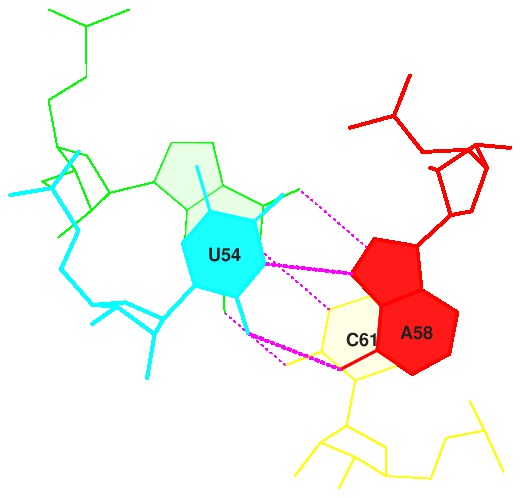 Рис.16. Стекинг-взаимодействие GU/AC. (step12) |
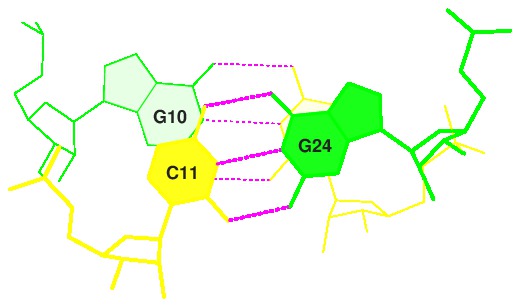 Рис.17. Стекинг-взаимодействие GC/GC. (step21) |
Рассмотрим также и пары с отсутствием перекрывания:
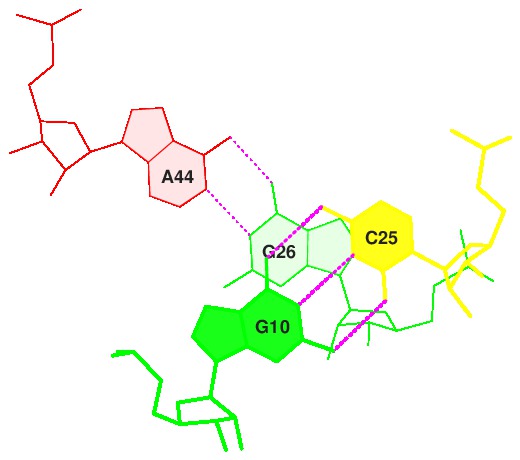 Рис.18. Step20. Стэкинг-взаимодействие минимально. |
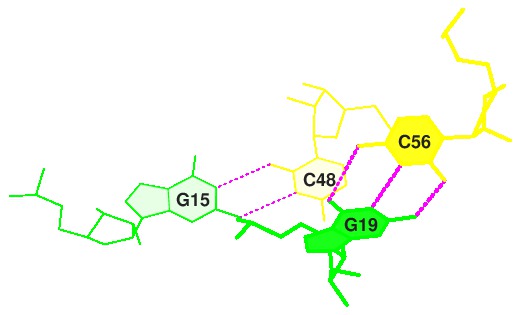 Рис.19. Step26. Стэкинг-взаимодействие минимально. |
Проверим пространственное расположение взаимодействующих пар в JMOl. Возьмем, например, пару GU/AC из step1. На рис.20 мы видим подтверждение, что эта пара имеет одно из максимальных стэкинг-взаимодействий.
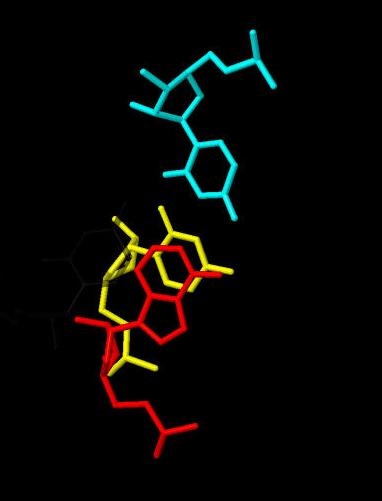
Рис.20. Пространственное расположение пары GU/AC. Красным цветом показан A62, голубым - U52, желтым - C63.