| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Задание 1. Поиск ДНК-белковых контактов в заданной структуре.
Упр.1
Полученный скрипт: script.txt
Скрипт последовательно выводит на экран ДНК в проволочной модели, выделяет множества set1, set2, set3.
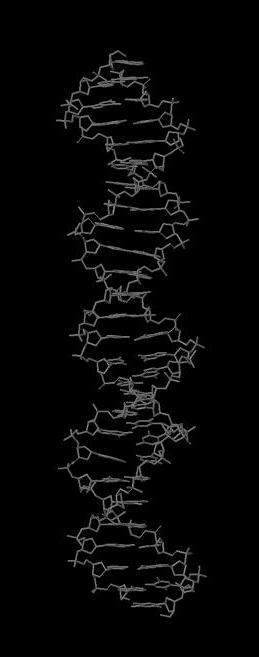 Структура ДНК в проволочной модели. |
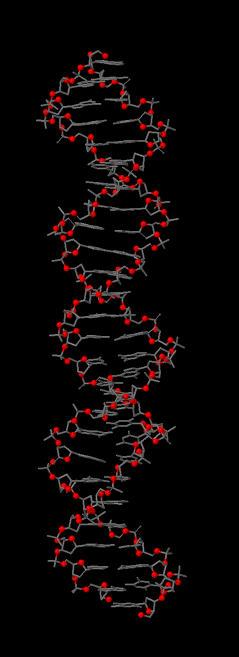 Красным цветом выделено множество set1. |
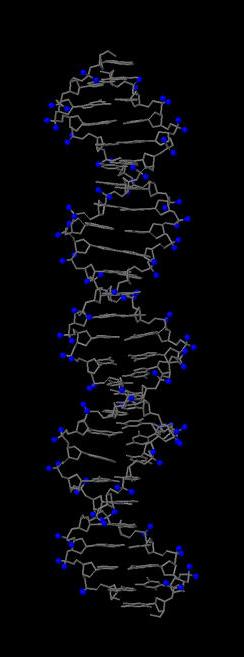 Голубым выделено множество set2. |
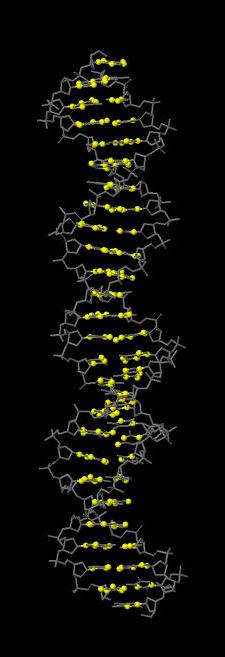 Желтым выделено множество set3. |
Упр.2 Описание ДНК-белковых контактов в заданной структуре. Сравнение количеств контактов разной природы.
Будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5A. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5A.
Полученный скрипт: script1.txt
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 7 | 53 | 60 |
| остатками фосфорной кислоты | 20 | 37 | 57 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 16 | 19 |
| остатками азотистых оснований со стороны малой бороздки | 4 | 9 | 13 |
Упр.3,4.
Используем следующие команды для получения изображений:
remediator --old "1MDM.pdb" > "1MDM_old.pdb"
nucplot 1MDM_old.pdb
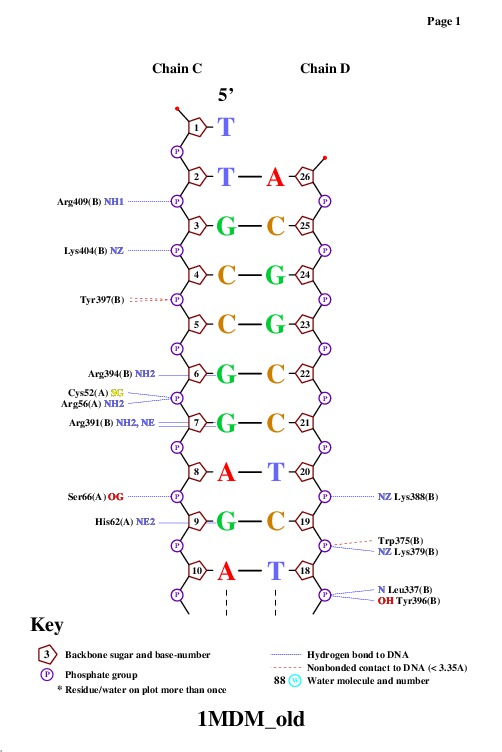 Рис.1. Схема ДНК-белковых контактов-1. |
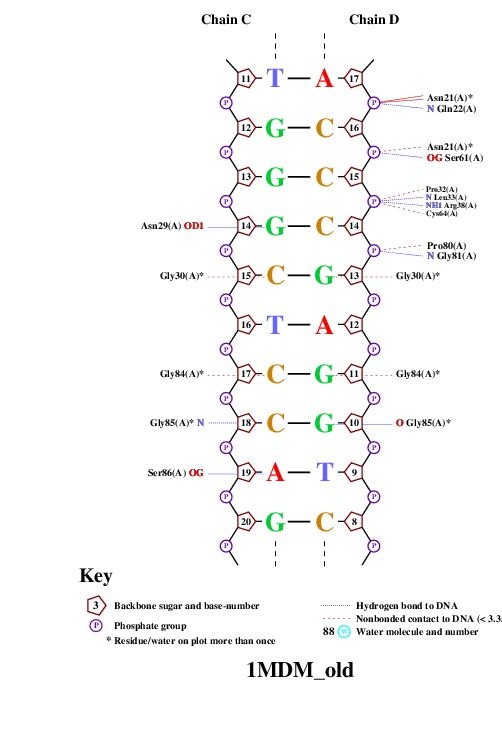 Рис.2. Схема ДНК-белковых контактов-2. |
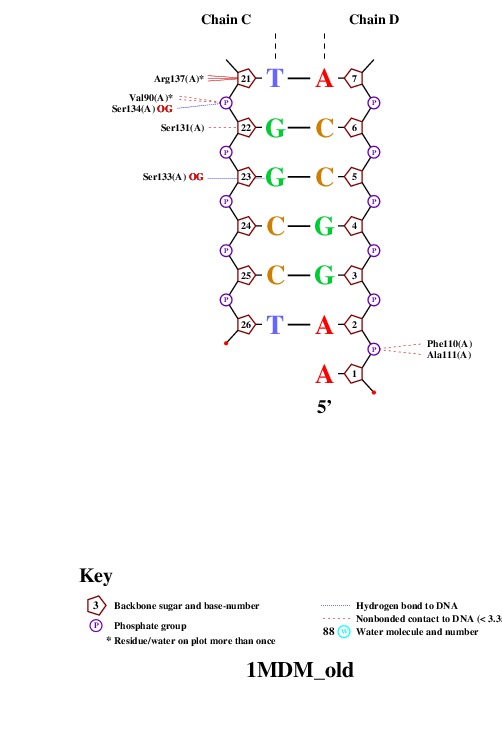 Рис.3. Схема ДНК-белковых контактов-2. |
Аминокислотных остатков с наибольшим числом указанных на схеме контактов с ДНК несколько: остатки Gly85, Gly30, Asn21, Arg391 и Gly84. Все они образуют по 2 контакта. Аминокислотный остаток, наиболее важный для распознавания последовательности ДНК - Arg, т.к. Arg56, Arg391 и Arg394 взаимодействую именно с азотистыми основаниями. |
Приведем пример контактов белка с ДНК:
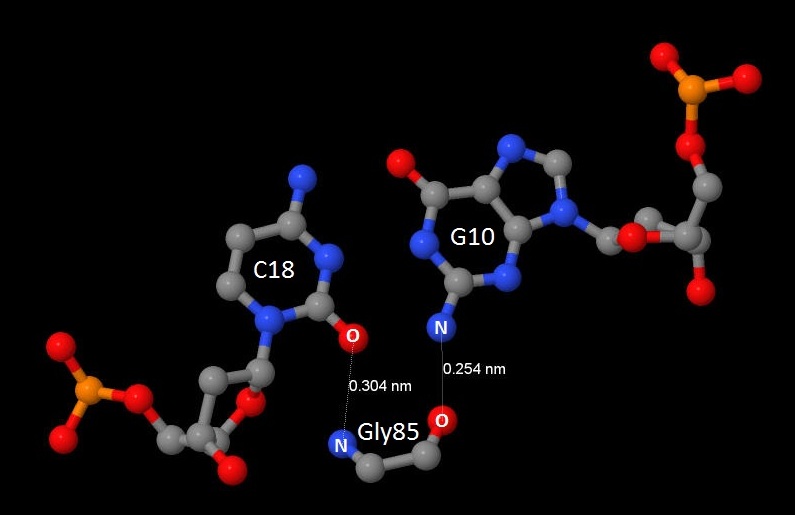 Рис.4. Изображение контакта Gly85 c ДНК. |
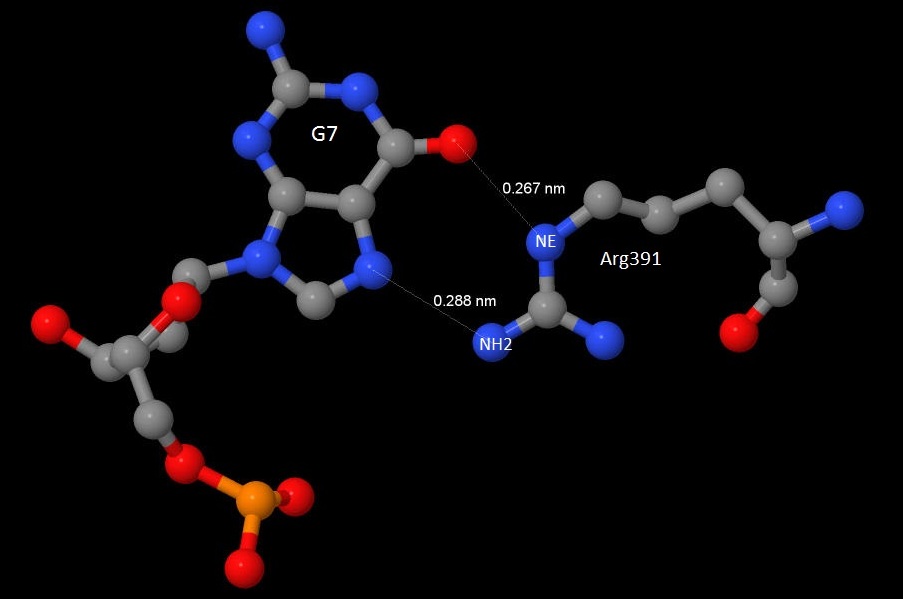 Рис.5. Изображение контакта Arg391 с ДНК. |
Задание 2. Предсказание вторичной структуры заданной тРНК
Упр.1.
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. Найдём возможные комплементарные участки в последовательности исследуемой тРНК - структуры 1I9V.pdb (последовательность тРНК можно найти в файле 1I9V.fasta) с помощью следующей команды:
einverted 1I9V.fasta
Необходимы параметры:
Gap penalty: 12
Minimum score threshold: 1
Match score: 3
Mismatch score: -3
Полученный файл: 1.inv
Упр.2. Предсказание вторичной структуры тРНК по алгоритму Зукера.
Программа mfold из пакета EMBOSS реализует алгоритм Зукера. Не изменяя параметры (P=5) получим наиболее близкую к предполагаемой структуру. (Рис.6)
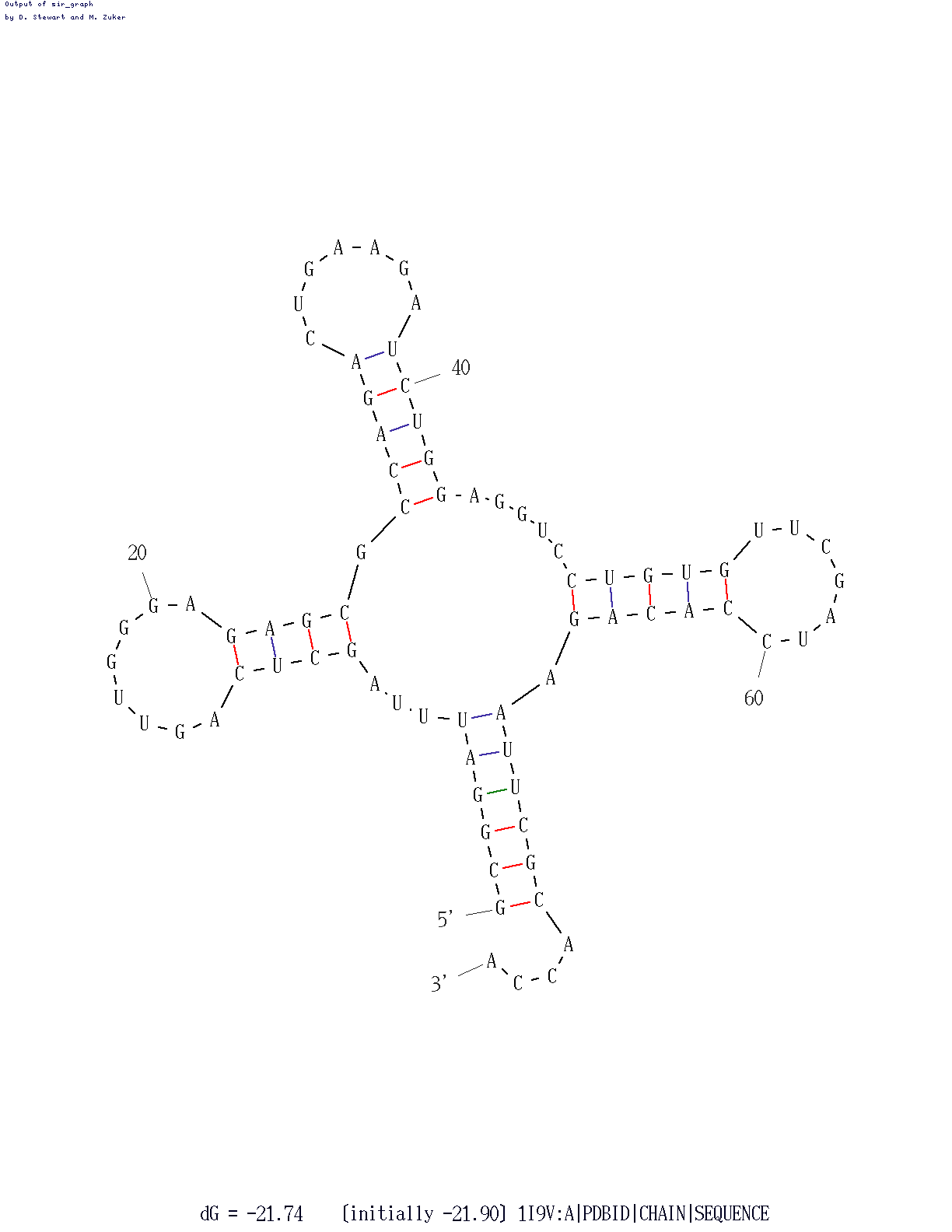
Рис.6. Структура тРНК, полученная с помощью алгоритма Зукера.
| Участок структуры | Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | (5') 1-7 (3') (5') 66-72 (3') Всего 7 пар |
предсказано 0 пар | предсказано 6 пар из 7 (кроме 7-66) |
| D-стебель | (5') 10-13 (3') (5') 22-25 (3') всего 4 пары |
предсказано 0 пар | все 4 пары предсказаны |
| T-стебель | (5') 49-52 (3') (5') 62-65 (3') всего 4 пары |
предсказано 5 пар (+ пара 53-61) | предсказаны все 4 пары + пара 53-61 |
| Антикодоновый стебель | (5') 40-43 (3') (5') 27-30 (3') всего 4 пары |
предсказано 4 пары + 26-31 и комплементарная ей цепь 48-43 | все 4 пары предсказаны + пара 39-31 |
| Общее число канонических пар нуклеотидов | 20 | 15 | 20 |