| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Задание 1.
Воспользуемся командой entret:
entret embl:D89965 -auto
Получили файл: d89965.entret. Для поиска набора трансляций всех открытых рамок данной последовательности длиной более 30 аминокислот, считая открытой рамкой последовательность триплетов от старт-кодона до стоп-кодона, при использовании стандартного кода, введем следующую команду:
getorf D89965.entret -table 0 -minsize 90 -find 1
Получили файл: d89965.orf. Определим, какая из найденных открытых рамок соответствует (полностью или частично) приведённой в поле FT кодирующей последовательности (CDS). Ниже представлены данные записи EMBL, где представлена CDS 163-435 и найденная третья из 5 открытая рамка 163-432:
FT source 1..448 FT /organism="Rattus norvegicus" FT /mol_type="mRNA" FT /sex="male" FT /tissue_type="stomach" FT /db_xref="taxon:10116" FT CDS 163..435 FT /product="RSS" FT /note="Rat Stomach Serotonin receptor-related gene" FT /db_xref="GOA:P0A7B8" FT /db_xref="InterPro:IPR001353" FT /db_xref="InterPro:IPR022281" FT /db_xref="PDB:1E94" FT /db_xref="PDB:1G4A" FT /db_xref="PDB:1G4B" FT /db_xref="PDB:1HQY" FT /db_xref="PDB:1HT1" FT /db_xref="PDB:1HT2" FT /db_xref="PDB:1NED" FT /db_xref="PDB:4G4E" FT /db_xref="UniProtKB/Swiss-Prot:P0A7B8" FT /protein_id="BAA14040.1" FT /translation="MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY FT GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA"
>D89965_5 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM
Данная запись EMBL ссылается на запись P0A7B8 в Swiss-Prot (/db_xref="UniProtKB/Swiss-Prot:P0A7B8"). Получим последовательность этой записи с помощью следующей команды:
seqret sw:p0a7b8
А теперь поищем, какой все-таки рамке соответствует полученная последовательность: hslv_ecoli.fasta
blastp -query hslv_ecoli.fasta -subject d89965.orf -out blastp.out
Получим файл: blastp.out
Query= HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2)
(Heat shock protein HslV)
Length=176
Subject= D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS,
complete cds.
Length=98
Score = 200 bits (509), Expect = 4e-71, Method: Compositional matrix adjust.
Identities = 98/98 (100%), Positives = 98/98 (100%), Gaps = 0/98 (0%)
Query 28 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 87
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
Sbjct 1 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 60
Query 88 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 125
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Sbjct 61 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 98
Сравним полученный результат со следующей рамкой:
>D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Заметим, что запись D89965 банка EMBL содержит последовательность мРНК для серой крысы, в то время как запись P0A7B8 банка Swiss-Prot содержит последовательность АТФ-зависимой субъединицы протеазы HslV кишечной палочки. Почему же это могло произойти? Дело в том, что кишечная палочка широко встречается в нижней части кишечника теплокровных организмов. Т.е. могло произойти так, что отсиквенированный геном, казалось бы, крысы, оказался геномом кишечной палочки, в результате погрешностей эксперимента (могли попасть чужие мРНК). Затем аннотация генома была проведена, скорее всего, автоматически и так появлилась неверная запись (т.к. банку SwissProt cтоит доверять в большей степени, чем Embl, ведь там аннотация идет неавтоматически и подтверждается экспериментально). Соответственно, произошла ошибка при поиске ORF.
Задание 2.
Будем использовать команды пакета emboss. Для получения всех доступных в Swissprot последовательностей алкогольдегидрогеназ используем команду:
seqret sw:adh*_* adh.fasta
Получим файл с универсальными адресами (USA) этих последовательностей с помощью следующей команды:
infoseq adh.fasta -usa -only > adh.infoseq
Получим из этого файла-списка другой, меньший:
grep -f MyOrg.txt adh.infoseq > adh_MyOrg.infoseq
И последняя команда:
seqret @adh_MyOrg.infoseq adh_MyOrg.fasta
Используемый файл:
adh_MyOrg.fasta
Полученные файлы:
adh.fasta
adh.infoseq
adh_MyOrg.infoseq
Итоговый файл:
adh_MyOrg.fasta
Задание 3. EnsEMBL
Идентификатор гена: FOG1_HUMAN. Файл с последовательностью гена: af488691.fasta. Поищем информацию о нем, используя портал EnsEMBL. Для этого воспользуемся сервисов BLAST/BLAT. Что мы видим? В блоке Alignment Locations vs. Karyotype мы видим расположение участка генома человека, который выровнялся с исходной последовательностью. В нашем случае, искомый фрагмент расположен на большом плече 16 хромосомы.
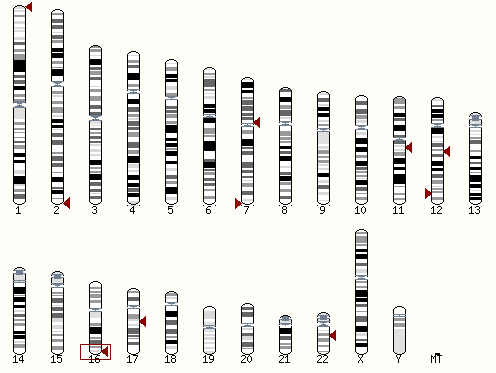
В следующем разделе Alignment Locations vs. Query в графическом виде приведена информация о полученном выравнивании (HSP - это high-scoring segment pair):

В разделе Alignment Summary приведена таблица находок с указанием различной информации, можно также выбирать строк таблицы, которые будут отображены.
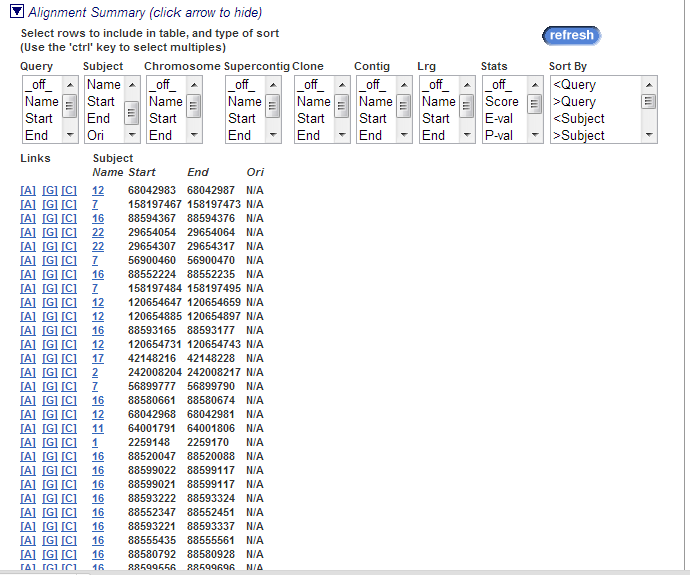
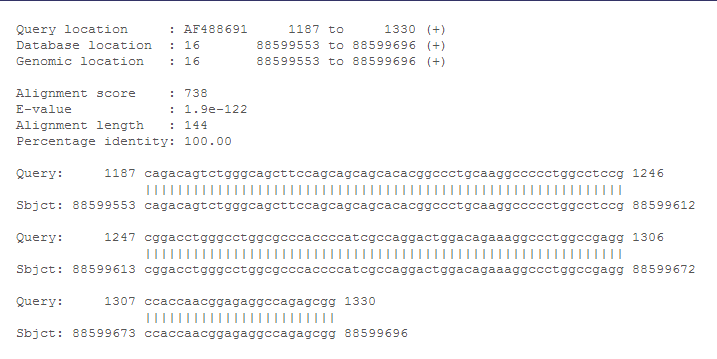
В колонке Links есть 3 ссылки. Ссылка А (Alignment) ведет нас к выравниванию, представленному ниже. На данном выравнивании:
Координаты запроса: AF488691 1187-1330 (+)
Местоположение в базе и в геноме: 16 (хромосома), 88599553-88599696 (+)
Alignment score (вес выравнивания): 738
E-value: 1.9e-122
Alignment length (длина выравнивания): 144
Percentage identity (процент совпадений): 100.00
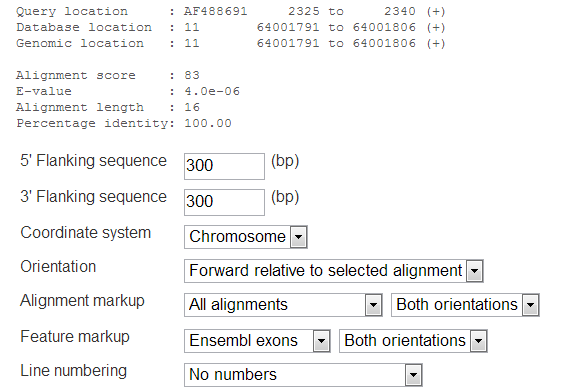
Следующий раздел G (Genome Sequence) содержит иноформацию о последовательности выровненного участка генома. Здесь можно самому залавать координаты фланкирующих областей, а также менять различные параметры, такие, как, например, ориентация (вперед по выравниванию или по координатной системе), саму координатную систему (хромосома, контиг, суперконтиг, правда не очень понятно, почему в этом окне 4 раза предлагают выбрать хрмосому) и другие.

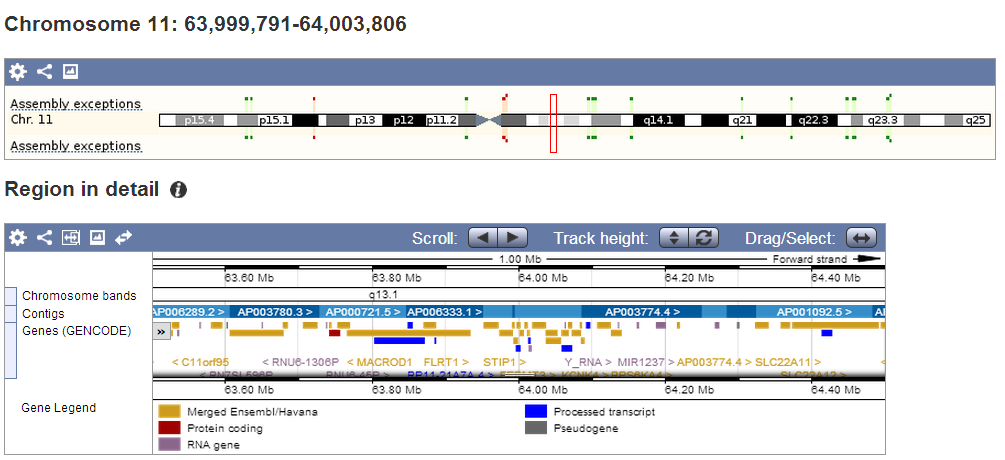
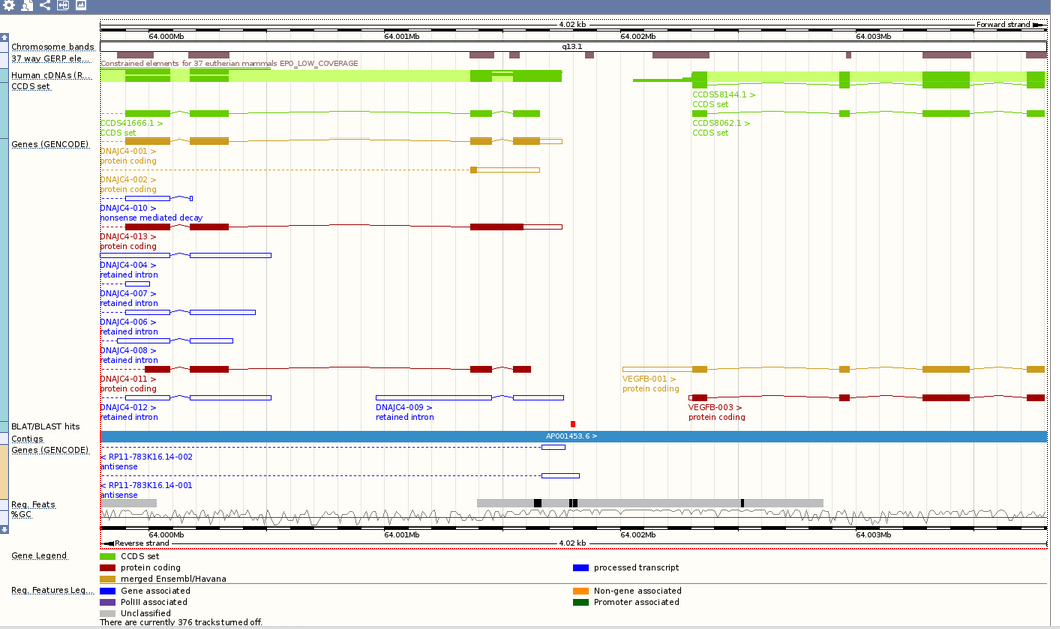
Следующая ссылка C (ContigView) позволяет рассмотреть участок человеческого генома. Здесь можно посмотреть информацию о расположенных на нем генах, о экзонах и интронах, соответствующиъ контигах (в разделе Region in detail). Можно сохранять изображение с данными в различных форматах (png, pdf...), пермещаться по участку; нажимая на названия генов, можно получить информацию о них. Также можно регулировать параметры изображений (увеличивать масштаб). Есть отличная ссылка, пройдя по которой можно подробнее изучить все составляющие выдачи: Help
.
А теперь попробуем другие возможности этого портала
Ген FOG1 можно найти, зайдя на главную страницу:
Найдено два результата: Transcript: ZFPM1-001 (транскрипт гена) и Gene: ZFPM1 (сам ген). На соответствующих страницах можно найти информацию, например, о расположении экзонов.
На портале также есть раздел BioMart. В нем мы можем выбирать базу данных и организм, можно посмотреть пример скрипта, описывающего возможности BioMart. Можно предположить, что он нужен для вывода результатов, используя набор данных, с помощью скрипта ("вытаскивание данных").
Еще один раздел Downloads позволяет скачивать различные базы данных и полезные приложения.
В разделе Help & Documentation можно посмотреть, как работать с данным порталом.
А также можно изучить данные о экспресии генов в различных тканях, узнать о заболеваниях и фенотипах, строить генетические деревья.

Подводя итог: портал многофункционален и удобен в использовании, есть специальные страницы, где можно разобраться, какие данные что иллюстрируют. Помимо известной функции поиска гена, выравнивания есть также и другие специфические функции (таблицы фенотипов, например).