| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Задание №0
К мембранным белкам относятся белки, которые встроены в клеточную мембрану или мембрану клеточной органеллы или ассоциированы с таковой. Около 25 % всех белков являются мембранными. Мембранные белки, контактирующие с гидрофобной частью липидного бислоя, должны быть амфифильными. Те участки белка, которые взаимодействуют с углеводородными цепями жирных кислот, содержат преимущественно неполярные аминокислоты. Участки белка, находящиеся в области полярных "головок", обогащены гидрофильными аминокислотными остатками. Белки мембран различаются по своему положению в мембране. Они могут глубоко проникать в липидный бислой или даже пронизывать его - интегральные белки, либо разными способами прикрепляться к мембране - поверхностные белки.
Мне был дан белок с идентификатором 3QE7 (Uniprot - P0AGM7 (URAA_ECOLI)). C помощью баз данных OPM или PDBTM для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков найдем параметры, представленные в таблице 1.
| PDB код | Тип (спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1m0l | спираль | архебактериальная мембрана Halobacterium salinarum | 31.8 ± 1.1 | 23 |
| 4hea | спираль | внутренняя мембрана Thermus thermophilus | 29.8 ± 0.5 | 23 |
| 4cg5 | спираль | эндоплазматический ретикулюм Canis lupus familiaris | 27.2 ± 0.9 | 17,5 |
| 1i78 | барель | наружная мембрана Escherichia coli | 26.5 ± 1.6 | 10 |
| 2qtk | баррель | наружная мембрана Pseudomonas aeruginosa | 23.3 ± 1.0 | 9 |
| 2lme | баррель | наружная мембрана Yersinia enterocolitica | 23.4 ± 2.2 | 9 |
Таблица 1.
Задание №1: отбор гомологов
Для отбора гомологов будем использовать программу BLAST. Белок 3QE7 c последовательностью из организма Escherichia coli (strain K12) будем использовать в качестве исходного для поиска. Увеличим максимальное количество хитов (max target hits), которые будет искать программа до 500. Исключим при поиске филум: Proteobacteria, тем самым вы запретим BLAST выдавать сходные белки из того же филума. Порог e-value поставим равным 1. В качестве базы данных используем RefSeq. Использовался алгоритм blastp (protein-protein BLAST).
Последовательности найденных гомологов: seq_homologs.fasta
Задание №2: анализ структуры выданного белка
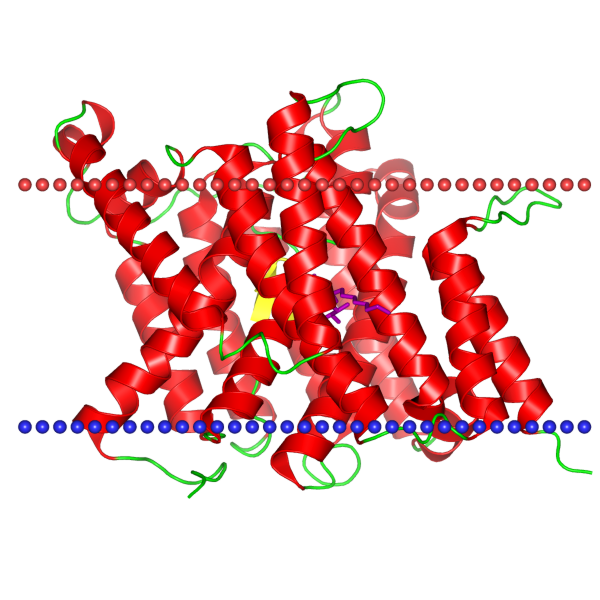
Рис.1. Структура белка 3QE7.
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей | Название белка |
| 3QE7 | Escherichia coli (strain K12) | 2.A.40.1.1 | внутренняя мембрана | 16 ± 0° | 14 | Урацил-пермеаза |
Таблица 2.Описание структуры трансмембранного белка XXXXXXXX (идентификатор PDB ZZZZ, цепь Y)
2.* - потенциал-зависимые белки-транспортеры
2.A.* - белки, облегчающие транспорт водорастворимых веществ через мембрану
2.А.40.* - семейство NCS2, белки которого катализируют следующие реакции:
Nucleobase (out) H+(out) %u2192 Nucleobase (in) H+(in)
Ascorbate (out) Na+(out) %u2192 Ascorbate (in) Na+(in)
2.A.40.1.* - пермеазы и транспортеры азотистых оснований
Задание №3: анализ множественного выравнивания трансмембранных белков
В качестве гомолога возьмем белок с идентификатором D2RJ11_ACIFV ( YP_003398378.1) из Acidaminococcus fermentans (strain ATCC 25085 / DSM 20731 / VR4). В программе Jalview с помощью Muscle with defaults построим множественное выравнивание выбранных белков. Немного поработаем с ним. Добавим 2 строки аннотации: "TM_REAL", где пометим трансмембранные спирали нашего белка и "TM_PREDICTED", куда вручную нанесем предсказанные программой TMHMM спирали гомолога. Примененим цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки (Hydrophobicity: гидрофобные остатки выделены красным цветом). Также применим By Conservation с порогом в %. Часть белка, ориентированная в n-сторону мембраны (в сторону цитоплазмы), расположена снизу. Полученное изображение представлено на рисунке 2.
Предсказанные трансмембранные элементы для гомолога:
# WEBSEQUENCE Length: 422 # WEBSEQUENCE Number of predicted TMHs: 12 # WEBSEQUENCE Exp number of AAs in TMHs: 255.65811 # WEBSEQUENCE Exp number, first 60 AAs: 36.30859 # WEBSEQUENCE Total prob of N-in: 0.76172 # WEBSEQUENCE POSSIBLE N-term signal sequence WEBSEQUENCE TMHMM2.0 inside 1 12 WEBSEQUENCE TMHMM2.0 TMhelix 13 35 WEBSEQUENCE TMHMM2.0 outside 36 38 WEBSEQUENCE TMHMM2.0 TMhelix 39 61 WEBSEQUENCE TMHMM2.0 inside 62 67 WEBSEQUENCE TMHMM2.0 TMhelix 68 90 WEBSEQUENCE TMHMM2.0 outside 91 93 WEBSEQUENCE TMHMM2.0 TMhelix 94 116 WEBSEQUENCE TMHMM2.0 inside 117 122 WEBSEQUENCE TMHMM2.0 TMhelix 123 145 WEBSEQUENCE TMHMM2.0 outside 146 159 WEBSEQUENCE TMHMM2.0 TMhelix 160 182 WEBSEQUENCE TMHMM2.0 inside 183 188 WEBSEQUENCE TMHMM2.0 TMhelix 189 211 WEBSEQUENCE TMHMM2.0 outside 212 230 WEBSEQUENCE TMHMM2.0 TMhelix 231 253 WEBSEQUENCE TMHMM2.0 inside 254 300 WEBSEQUENCE TMHMM2.0 TMhelix 301 323 WEBSEQUENCE TMHMM2.0 outside 324 326 WEBSEQUENCE TMHMM2.0 TMhelix 327 349 WEBSEQUENCE TMHMM2.0 inside 350 368 WEBSEQUENCE TMHMM2.0 TMhelix 369 391 WEBSEQUENCE TMHMM2.0 outside 392 395 WEBSEQUENCE TMHMM2.0 TMhelix 396 418 WEBSEQUENCE TMHMM2.0 inside 419 422
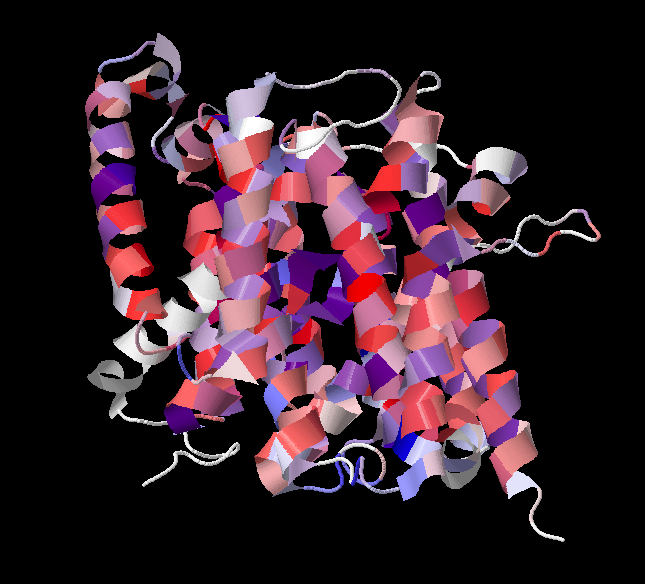
Рис. 2. Изображение белка, покрашенного по гидрофобности/гидрофильности.
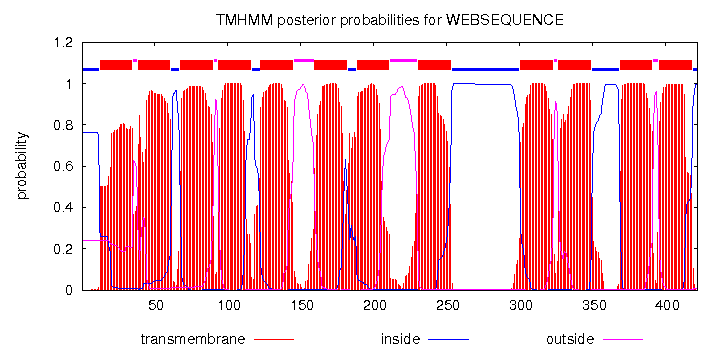
Рис. 3. Предсказание с помощью THHM.
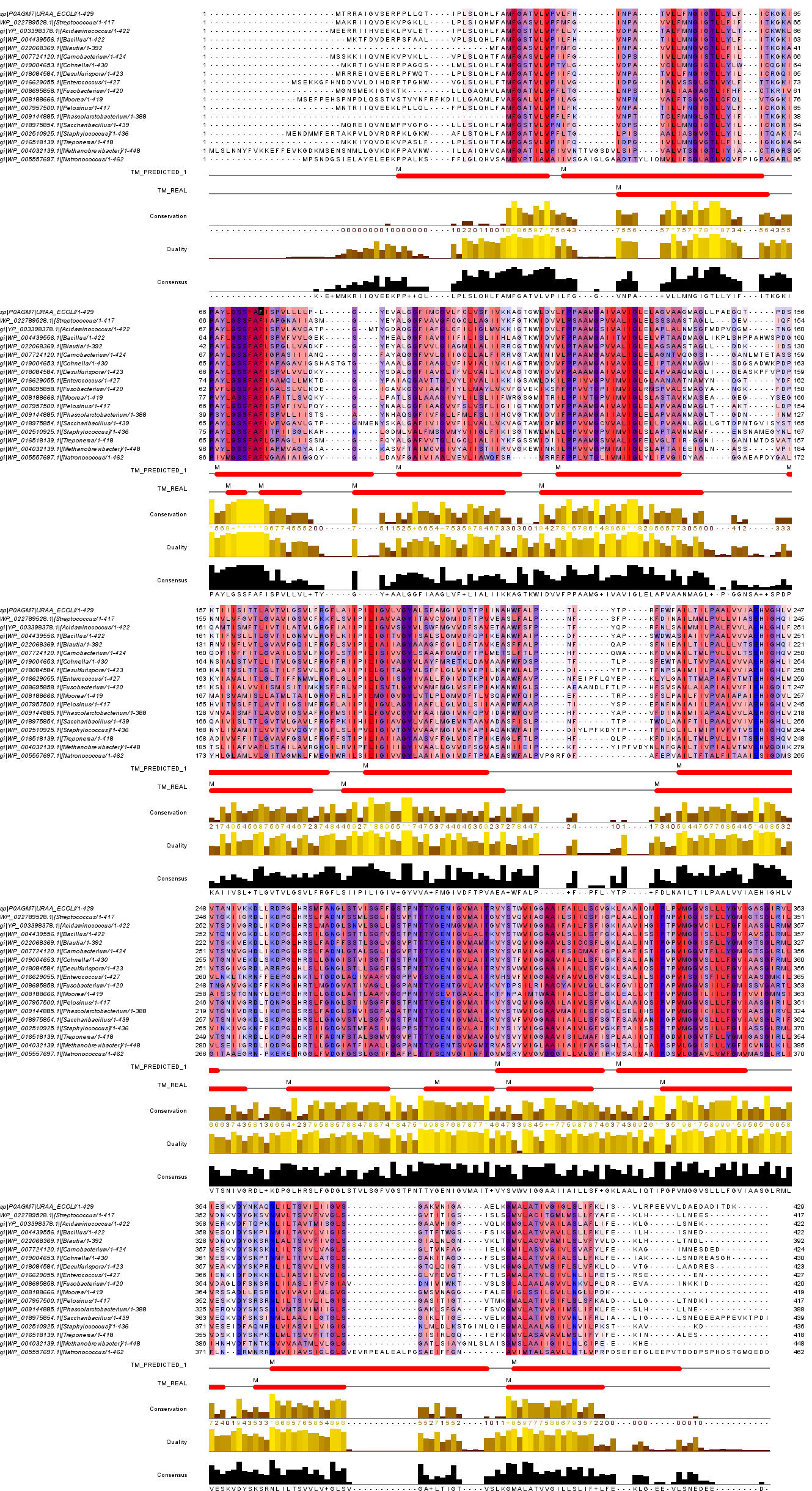
Рис.4. Выравнивание выборки белков. Для окраски использованы схемы: Hydrophobicity и By Conservation с порогом в 20%.
Полученное выравнивание можно посмотреть по ссылке: align.jar
Проанализируем полученные данные. В основном, участки, принадлежащие альфа-спиралям довольно консервативны. Наиболее часто встречающиеся в них остатки аминокислот: пролин, лейцин, валин, фенилаланин, аланин, глицин - гидрофобные. Однако, также встречаются поляные аминокислоты: серин, треонин. Это может быть связано с тем, что они могут взаимодействовать с другими молекулами, образуя сложную структуру. Поскольку эти остатки довольно консервативны, можно предположить их участие в функциональной роли белка. В межспиральных участках встречаются как консервативные, так и неконсервативные участки.
По данным предсказания TMHMM есть лишь частичные совпадения по спиралям и консеративным участкам. В исходной последовательности было 14 трансмембранных спиралей, а для гомолога предсказано 12. Сложно что-либо объяснить, т.к. для D2RJ11_ACIFV еще не известно PDB структуры.
Дополнительные задания
Увеличим клетку до размеров дыни. С чем можно сравнить толщину мембраны?
Рассмотрим в качестве примера клетку Bacillus subtilis, ее линейные размеры: 0,75 на 2—3 мкм. Возьмем толщину мембраны = 30Å. Средний диаметр дыни - 20 см. Итого мы будем увеличивать линейные размеры клетки в 8,3*10^4 раз. Тогда толщина мембраны возрастет и станет равной 250 мкм. Сравнить, в таком случае, толщину мембраны можно с листом бумаги, толщина которого составляет 106 мкм (т.е. 2 листа нужно взять).
Правило positive inside
Правило состоит в следующем: часть белка, расположенная с n-стороны от мембраны (цитоплазма бактерии, матрикс митохондрии и строма хлоропласта по отношению к внутренним мембранам и т.п.), содержит больше положительно заряженных остатков (Lys, Arg, His). Проверим достоверность это утверждения, используя белок 3QE7 (Uniprot - P0AGM7 (URAA_ECOLI)). Для этого с помощью JMol получим изображение структуры белка с покрашенными положительно заряженными аминокислотами (Рис. 5).
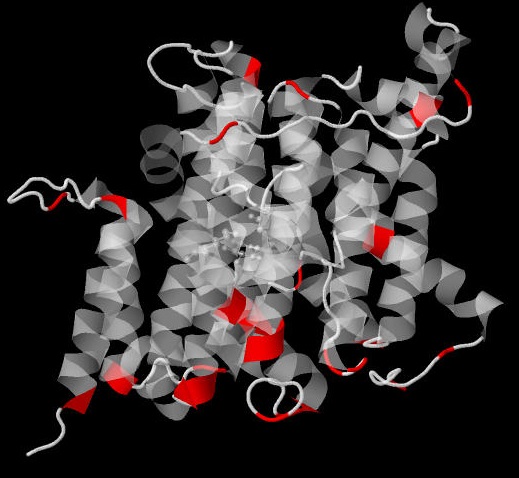
Рис. 5. Структура 3QE7. Часть белка, расположенная с n-стороны от мембраны, находится внизу. Красным выделены остатки Arg, His, Lys.
Таким образом, мы можем сделать вывод, что правило выполняется, т.к. на n-стороне от мембраны больше положительно заряженных остатков аминокислот, чем на p-стороне.
Предсказание ТМ бета-баррель по последовательности
Для выполнения этого задания был взят белок 3EMN (VDAC-1 канал в митохондриях) из Mus musculus. В качестве его гомолога был взят белок с неизвестной PDB структурой F7AKB9 (F7AKB9_CALJA) из Callithrix jacchus (White-tufted-ear marmoset). Воспользуемся сервисом PRED-TMBB.
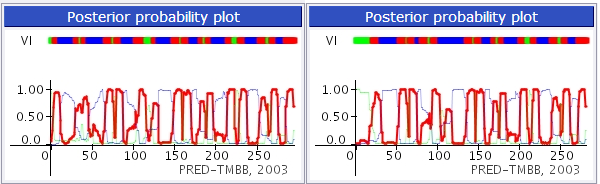
Рис. 6. Предсказание бета-баррелей для последовательностей 3EMN и F7AKB9.
Судя по полученным данным, программа нашла для исходной последовательности нашла 14 трансмембранных структур (хотя их заявлено 19) и столько же для гомолога.
Гидрофобность остатков бета-барреля, обращенные в мембрану
Для примера возьмем тот же белок 3EMN. Воспользуемся сервисом SheeP. На рис.7 представленна карта остатков, на которой красным цветом выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь.
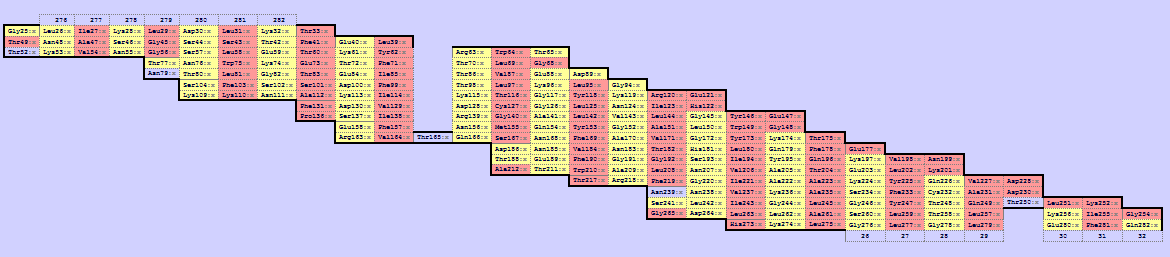
Рис. 7. Карта остатков белка 3EMN, полученная в SheeP.
Можно заметить, что значительная часть остатков, обращенных наружу, гиддрофобны (фенилаланин, валин, аланин, лейцин, метионин, глицин, изолейцин). Однако также встречаются и гидрофильные аминокислоты: треонин, серин, глутаминовая кислота, аргинин, гистидин. Рассмотрим их положение в структуре (рис. 8):
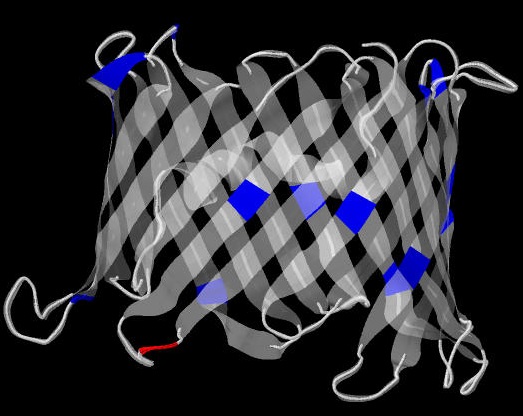
Рис. 8. Изображение 3D-структуры 3EMN с выделенными синим цветом гидрофильными остатками.
Часть из этих гидрофильных остатков принадлежит перегибам, однако часть из них находится прямо на бета-листах. Это может быть связано с особенностями функциональной роли этого белка.