| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Реконcтрукция филогенетических деревьев
ТАКСОНЫ БАКТЕРИЙ
Таксоны бактерий представлены в таблице:
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Clostridium botulinum | CLOB1 | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Clostridium tetani | CLOTE | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Finegoldia magna | FINM2 | Firmicutes | Clostridia | Clostridiales | Clostridiales incertae sedis |
| Bacillus anthracis | BACAN | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Lactobacillus delbrueckii | LACDA | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Streptococcus pneumoniae | STRPN | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
| Staphylococcus epidermidis | STAES | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
На дереве мы можем наблюдать ветви, выделяющие таксоны.
Нетривиальная ветвь {CLOT,CLOB,FINM2}vs{LACDA,STRPN,BACAN,STAES} отделяет классы Firmicutes и Clostridia, а также отряд Clostridiales.
Отдельной ветвью в случае {LACDA,STRPN}vs{BACAN,STAES,CLOTE,CLOB1,FINM2} выделен отряд Lactobacillales,
также как и отряд Bacillales: {BACAN,STAES}vs{LACDA,STRPN,CLOTE,CLOB1,FINM2}.
Ветвь {CLOTE,CLOB1}vs{FINM2,LACDA,STRPN,BACAN,STAES} отделяет семейство Clostridiaceae.
МНОЖЕСТВЕННОЕ ВЫРАВНИВАНИЕ И РЕКОНСТРУКЦИЯ ДРЕВЬЕВ
Был взят рибосомный белок S2 с мнемоникой RS2. Для получения последовательностей белков выбранных бактерий используем команду:
seqret sw:RS2_CLOB1
Аналогично поступим с остальными организмами и получим файл в fasta-формате, где изменены названия на мнемонику видов.
В JalView получим выравнивание, используя программу Muscle with Defaults, в "блочной" форме (Format > Wrap в меню), с блоками шириной 100 (получилось 99) остатков, с раскраской по проценту идентичности, представленный ниже.
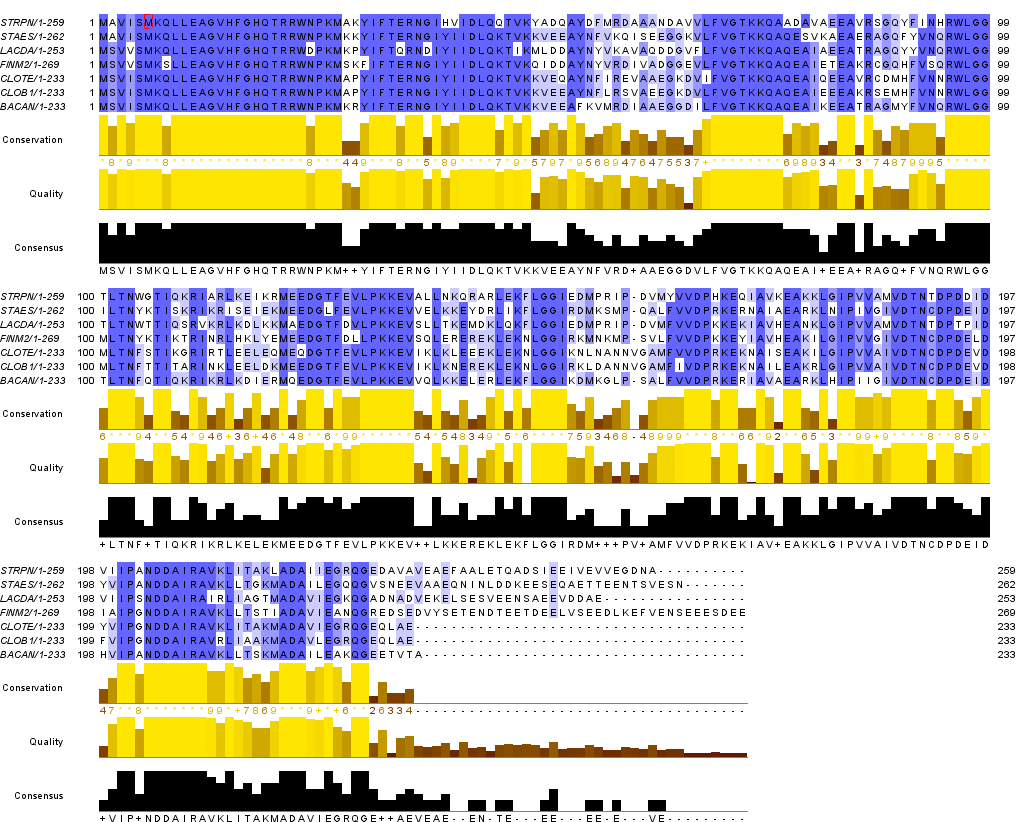
Полученное выравнивание в формате.jar и .fasta приведено в соответствующих файлах.
В выравнивании можно найти несколько диагностических позиций, по которым можно судить о принадлежности к определенному таксону. Ниже преставлено изображение выравнивания с указанными позициями:
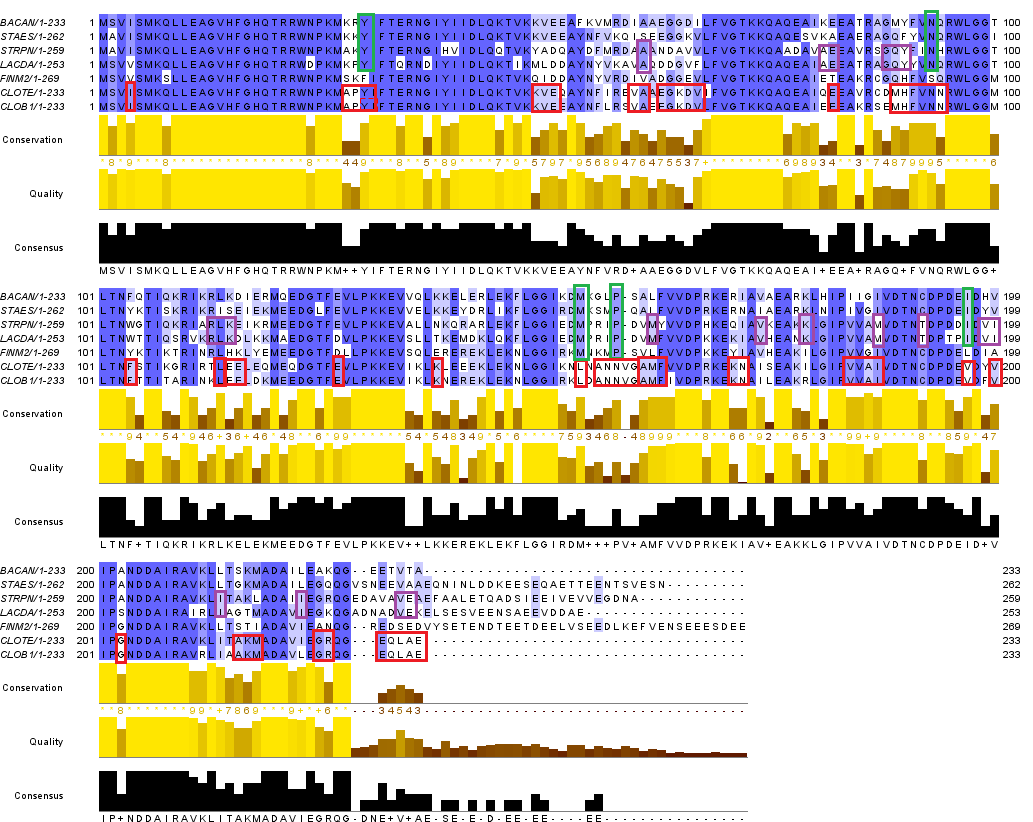
Красным цветом выделены позиции, предположительно отвечающие за семейство Clostridiaceae, фиолетовый - за отряд Lactobacillales, зеленый - за класс Bacilli.
В JalView доступны четыре метода для реконструкции филогенетического дерева, которые доступны из меню Calculate > Calculate Tree. Ниже приведены изображения, полученные с помощью программы MEGA.
Average Distance Using % Identity
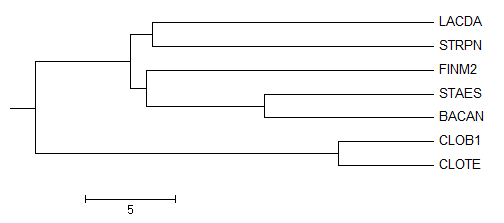
Отличие от правильного дерева (есть в этом дереве, но нет в правильном): {LACDA,STRPN,CLOB1,CLOTE}vs{STAES,BACAN,FINM2}
Есть в правильном дереве, но нет в этом:
{FINM,CLOTE,CLOB1}vs{LACDA,STRPN,BACAN,STAES}
Для этого дерева мы можем проследить большее сходство белка бактерии Finegoldia magna из класса Clostridia и белков из класса Bacilli. Это может подтверждаться позициями в выравнивании.
Общее:
{CLOTE,CLOB1}vs{FINM2,LACDA,STRPN,BACAN,STAES}
{LACDA,STRPN}vs{BACAN,STAES,CLOTE,CLOB1,FINM2}
{BACAN,STAES}vs{LACDA,STRPN,CLOTE,CLOB1,FINM2}.
Average Distance Using BLOSUM62
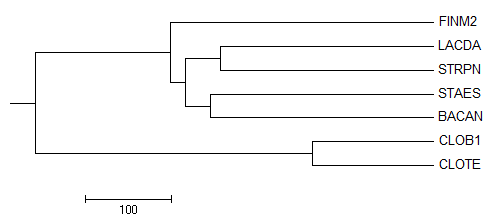
Есть в этом дереве, и нет в правильном:
{STRPN,LACDA,FINM2}vs{STAES,BACAN,CLOB1,CLOTE}
Есть в правильном дереве, но нет в этом:
{FINM,CLOB1,CLOTE}vs{BACAN,STAES,STRPN,LACDA}
Опять наблюдаем выделение белка бактерии Finegoldia magna из класса Clostridia в класс Bacilli.
Общее:
{CLOT,CLOB,FINM2}vs{LACDA,STRPN,BACAN,STAES}
{CLOTE,CLOB1}vs{FINM2,LACDA,STRPN,BACAN,STAES}
{LACDA,STRPN}vs{BACAN,STAES,CLOTE,CLOB1,FINM2}
{BACAN,STAES}vs{LACDA,STRPN,CLOTE,CLOB1,FINM2}.
Neighbour Joining Using % Identity
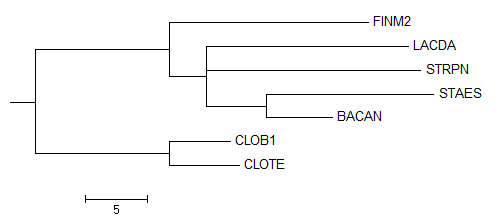
Это дерево не является бинарным. Отличие запишем в виде скобочной формулы: ((FINM2,(LACDA,STRPN,(STAES,BACAN))),(CLOTE,CLOB1)). Т.е. LACDA и STRPN не объединены общим предком, как это было в правильном дереве, а FINM2 также соотносится с классом Bacilli.
Все ветви в этом дереве правильные, но есть в правильном дереве отличия. Есть в правильном дереве, но нет в этом:
{LACDA,STRPN}vs{BACAN,STAES,FINM2,CLOB1,CLOTE}
Общее:
{CLOT,CLOB,FINM2}vs{LACDA,STRPN,BACAN,STAES}
{CLOTE,CLOB1}vs{FINM2,LACDA,STRPN,BACAN,STAES}
{BACAN,STAES}vs{LACDA,STRPN,CLOTE,CLOB1,FINM2}.
Neighbour Joining Using BLOSUM62
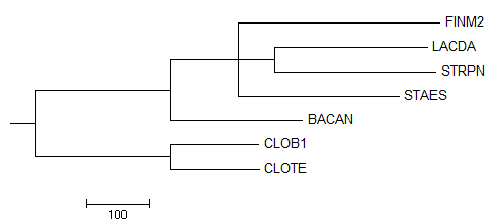
Это дерево тоже является небинарным. Есть в этом дереве, но нет в правильном:
{BACAN,CLOB1,CLOTE}vs{STRPN,STAES,FINM2,LACDA}
Есть в правильном дереве, но нет в этом:
{BACAN,STAES}vs{FINM2,LACDA,CLOTE,CLOB1,STRPN}
{BACAN,STAES,STRPN,LACDA}vs{CLOB1,CLOTE,FINM2}
Здесь уже BACAN и STAES не имеют общего узла, отделенного от {LACDA,STRPN}.
Общее:
{CLOTE,CLOB1}vs{FINM2,LACDA,STRPN,BACAN,STAES}
{LACDA,STRPN}vs{BACAN,STAES,CLOTE,CLOB1,FINM2}.
А теперь воспльзуемся другой программой для построения деревьев - MEGA. Импортируем файл в .fasta-формате и построим дерево с помощью алгоритма "Maximum Parsimony":
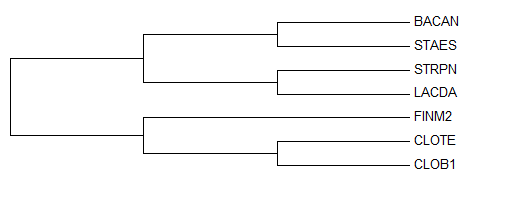
Отличий нет (при должном укоренении).