| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Построение дерева по нуклеотидным последовательностям
Построим филогенетическое дерево тех же бактерий, что были в предыдущих заданиях, используя при этом последовательности РНК малой субъединицы рибосомы (16S rRNA). Для начала получим последовательности РНК. Для этого найдем AC генома соответствующей бактерии, затем с помощью слдующих команд найдем координаты соответствующей РНК и вырежем в геноме этот участок:
entret embl:ac_number
grep -A 3 ' rRNA '
seqret embl:ac_number -sask
Для бактерии Streptococcus pneumoniae попалась неаннотированная запись генома, поэтому с ней пришлось немного помучиться. Был взят организм Lactococcus garvieae из рода Lactococcus, семейства Streptococcaceae, с координатами 16S-РНК: complement(1584062..1585593). Его последовательность РНК была выравнена с геномом Streptococcus pneumoniae c помощью команд:
makeblastdb -in STRPN_GENOME.fasta -dbtype nucl
blastn -task blastn -query search.fasta -db STRPN_GENOME.fasta -out file.out
Из полученного файла file.out были найдены предположительные координаты 16S-РНК в Streptococcus pneumoniae и прямое расположение на цепи.
Полученные данные (AC записи EMBL, в которой нашлась последовательность 16S rRNA, координаты этой РНК в этой записи, на прямой или комплементарной последовательности оказалась последовательность этой рРНК) запишем в таблицу:
| Название | Мнемоника | AC записи EMBL | Начало | Конец | Последовательность (+/-) |
| Clostridium botulinum | CLOB1 | CP000726 | 11004 | 13905 | + |
| Clostridium tetani | CLOTE | AE015927 | 176113 | 177621 | + |
| Finegoldia magna | FINM2 | AP008971 | 197837 | 199361 | + |
| Bacillus anthracis | BACAN | AE016879 | 9335 | 10841 | + |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160 | 46720 | + |
| Streptococcus pneumoniae | STRPN | AE005672 | 15359 | 16887 | + |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006 | 1599559 | - |
Полученные нуклеотидные последовательности (файл nucleotide.fasta) были выравнены с помощью JalView, Muscle with Default. Дерево реконструировалось методом минимальной эволюции. Изображение дерева с указанными мнемониками видов:
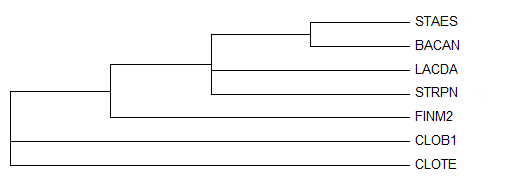
Правильное дерево:
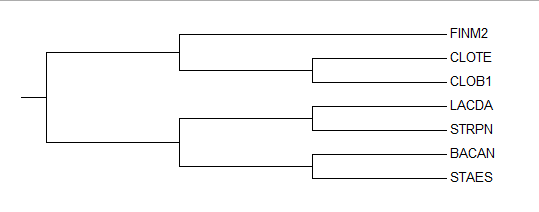
Дерево вышло небинарным. Совпали ветви: {STAES;BACAN}vs{LACDA;STRPN;FINM2;CLOB1;CLOTE} и {LACDA,STRPN,BACAN,STAES}vs{CLOB1,CLOTE,FINM2}. По сравнению с деревьями, построенными по белкам - построение по нуклеотидным последовательностям все же проигрывает, несмотря на консервативность 16S рРНК. Это может быть связано с тем, что нуклеотидный код составляют всего 4 нуклеотида, а аминокислотный - 20. Точечные мутации в нуклеотидной последовательности могут заметно ее изменять, не изменяя при этом белковую (синонимичные или "молчащие" замены). Попробуем использовать более долгий метод построения деревьев, но более качественный - метод наибольшего правдоподобия. Полученное дерево:
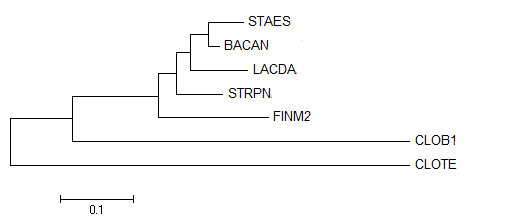
В этом случае дерево бинарное, общие ветви: {STAES;BACAN}vs{LACDA;STRPN;FINM2;CLOB1;CLOTE}, {LACDA,STRPN,BACAN,STAES}vs{CLOB1,CLOTE,FINM2}, {CLOB1,CLOTE}vs{LACDA,STRPN,BACAN,STAES,FINM2}. Существенных изменений, кроме удаления небинарности, не произошло.
Построение и анализ дерева, содержащего паралоги
Найдем в бактериях достоверные гомологи белка CLPX_BACSU. Для этого воспользуемся следующими командами:
makeblastdb -in proteo.fasta -dbtype prot -out prot
blastp -query clpx_bacsu.fasta -db prot -out clpx_gomologs_ids.txt -evalue 0.001
Полученный файл: clpx_gomologs_ids.txt
Затем отберем последовательности белков ранее рассматриваемых бактерий: gomologs.fasta.. С помощью программы JajView и Muscle with Defaults построим выравнивание последовательностей: align.fasta.
Полученное дерево (Neighbor Joining, в программе MEGA):
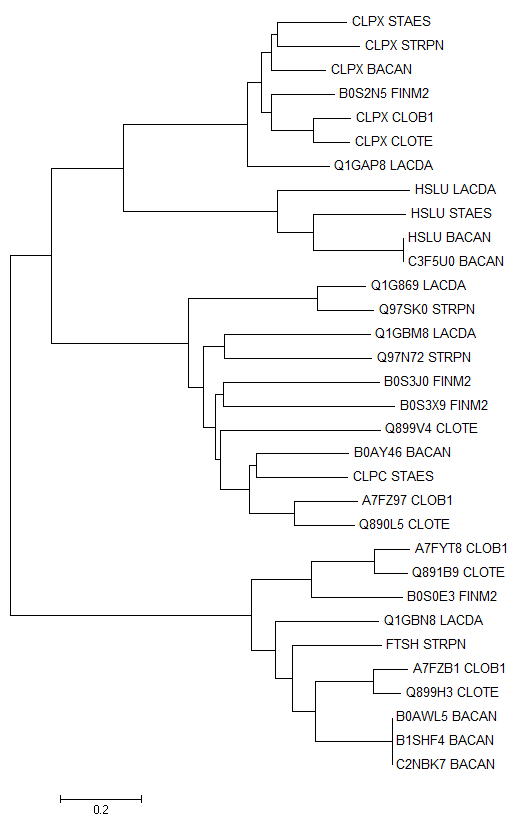
Два гомологичных белка будем называть ортологами, если они из разных организмов; и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.
На построенном дереве ортологами являются, например, CLPX_STAES и CLPX_STRPN; CLPX_CLOTE и CLPX_CLOB1. Паралогами являются B0S3J0_FINM2 и B0S3X9_FINM2, Q1GAP8_LACDA и HSLU_LACDA.
Пример дупликации гена: HSLU_BACAN и C3F5U0_BACAN; Q891B9_CLOTE и Q899H3_CLOTE.
Пример видообразования: B0S2N5_FINM2 и CLPX_CLOB1.