| Главная | Семестры | Проекты | Заметки | О себе | Полезные ссылки |
Построение поверхности, раскраска участка поверхности: pymol
1. Получение изображений поверхностей контактов.
Для задания была взята структура 2PUA (ДНК-связывающий белок, репрессор транскрипции PurR типа спираль-поворот-спираль). Сначала выделяем отдельно множества ДНК и отдельно мономеры белка. Затем уже ищем соответствующие контакты.
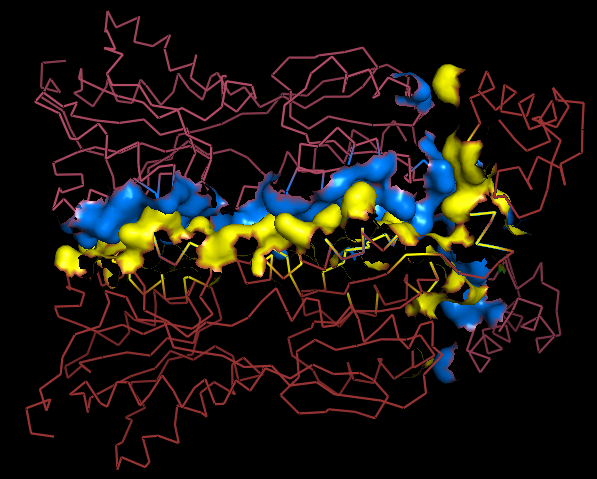
Рис.1. Поверхность контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера.
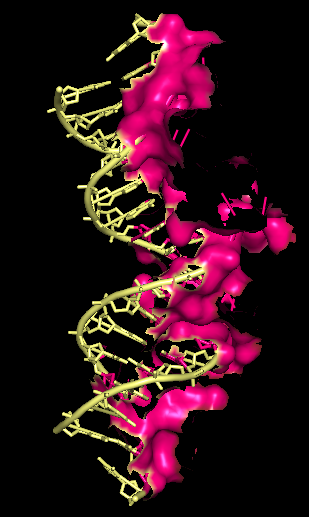
Рис.2. Поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт
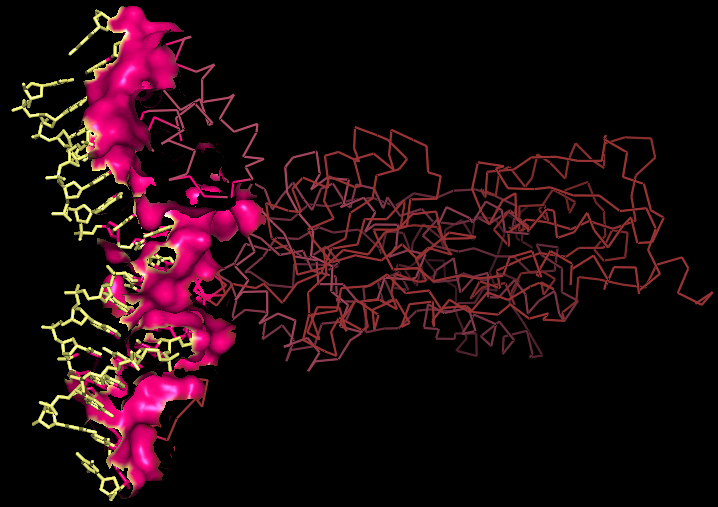
Рис.3. Поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали.
2. Изображение гидрофобных поверхностей (CluD).
Немного (или даже много) поколдовав над .pdb файлом, даем его на вход программе CluD и просим искать контакты между цепями (рис.4). Находятся 5 кластеров (порог ставили в 5Å, минимальное число остатков в кластере - 10). Затем воспользуемся текстовым файлом из выдачи CluD, с помощью данных из него запишем команду для выделения гидрофобных участков взаимодействия:command.txt
.
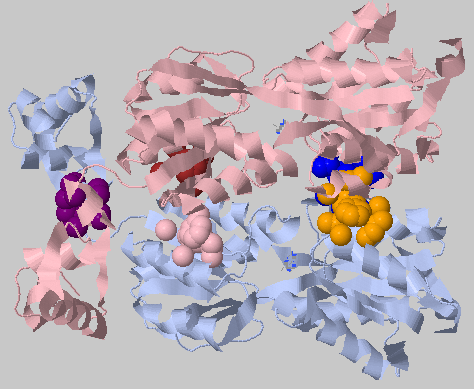
Рис.4. Изображение гидрофобных кластеров между мономерами в структуре 2PUA.
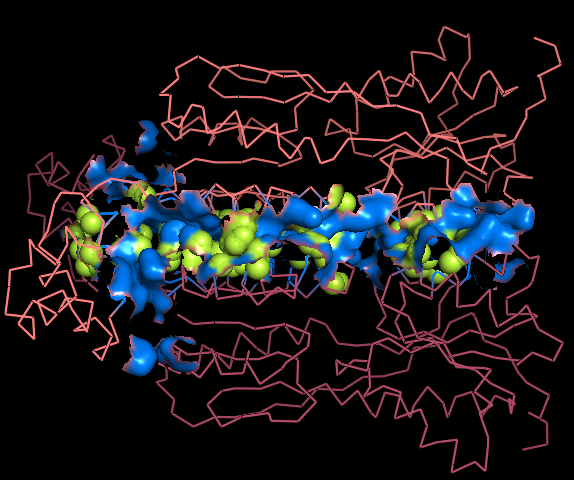
Рис.5. Поверхность контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера, гидрофобная поверхность выделена желтым цветом.