Миниобзор генома бактерии Paracidovorax citrulli
Павличенко Федор
Факультет биоинженерии и биоинформатики, Московский государственный университет
имени М. В. Ломоносова, Ленинские горы д. 1 стр. 73, 119234, Москва, Россия
Получено 10.12.2024; отредактировано 18.12.2024; принято 19.12.2024
Ключевые слова: Paracidovorax citrulli, transposase, IS element, transposon, operon, CDS, GC procent
РЕЗЮМЕ
В данном мини-обзоре был исследован геном бактерии Paracidovorax citrulli. Были построены гистограммы длин белков, процента GC CDS, промежутков между CDS и пересечений CDS. Были выдвинуты предположения о существовании трех оперонов и семнадцати IS-элементов и транспозонов.
1 ВВЕДЕНИЕ
Paracidovorax citrulli - грамотрицательная бактерия, поражающая
растения семейства Cucurbitaceae
(Тыквенные).[1]
Таксономическое положение изучаемой бактерии указано в таблице 1. На рисунке 1
изображена микрофотография данной бактерии. Ранее Paracidovorax citrulli именовалась как Pseudomonas
pseudoalcaligenes citrulli и Acidovorax citrulli [2].
Из-за своего поражающего свойства изучение данной бактерии является актуальной проблемой.
| Надцарство | Bacteria |
|---|---|
| Тип | Pseudomonadota |
| Класс | Betaproteobacteria |
| Порядок | Burkholderiales |
| Семейство | Comamonadaceae |
| Род | Paracidovorax |
| Вид | Paracidovorax citrulli |
2 МАТЕРИАЛЫ И МЕТОДЫ
2.1 Материалы
Материалами, на которых основывалось дальнейшее исследование, являлись следующие файлы:
- Таблица особенностей генома бактерии Paracidovorax citrulli:
Ссылка. - Таблица кодирующих последовательностей генома бактерии Paracidovorax citrulli:
Ссылка. - Последовательность хромосомы бактерии Paracidovorax citrulli:
Ссылка.
Все координаты генов, указанные ниже, соответствуют координатам генов в последовательности генома в файле GCF_022493915.1_ASM2249391v1_genomic.fna.
Ссылка на страницу, содержащую ссылки на вышеуказанные файлы, находится в разделе сопроводительные материалы (С1).
2.2 Методы
Построение гистограмм и прочих графиков, приведенных ниже в обзоре, было осуществлено при помощи электронных таблиц Google Sheets. Для получения данных использовались различные возможности электронных таблиц, такие как функции =СЧЁТЕСЛИ, =СЧЁТЕСЛИМН, =МАКС, =МИН, =СРЗНАЧ, =СРОТКЛ а также поиск по таблице. Кроме этого, обнаружение оперонов (3.8 Гипотетические опероны в геноме бактерии Paracidovorax citrulli) путем поиска групп близлежащих относительно друг друга генов, которые кодируют субъединицы белка или которые участвуют в одном и том же метаболическом процессе бактерии.
Также первые два файла , указанные в предыдущем пункте (Материалы), были перенесены в электронные таблицы(С2, С3).
Кроме вышеперечисленного, электронные таблицы использовались для обработки данных, полученных при помощи средств языка программирования Python или Bash (3.6 Старт-кодоны последовательных кодирующих последовательностей (CDS); 3.7 Стоп-кодоны последовательных кодирующих последовательностей (CDS); 3.10 Анализ мобильных генетических элементов генома бактерии Paracidovorax citrulli). Обработка полученных данных (указанных в подпункте 2.3 “Средства языка программирования Python”) проводилась в отдельной таблице “Mobile elements in genome of Paracidovorax citrulli”(С4). Отдельные листы именуются как “MEL_*номер*”, где номер - какой по очереди ген транспозазы на (+)-цепи генома бактерии Paracidovorax citrulli, где нумерация начинается с гена транспозазы по координатам 232341, 233288 нуклеотидов.
2.3 Средства программирования Python
При помощи средств языка программирования Python были получены результаты для следующих пунктов:
- 3.7 Стоп-кодоны последовательных кодирующих последовательностей (CDS).
- 3.9 Состав нуклеотидов A, T, G, C по репликонам бактерии Paracidovorax citrulli.
- 3.10 Анализ мобильных генетических элементов генома бактерии Paracidovorax citrulli
Для получения стоп-кодонов использовались 2 метода из вышеуказанного файла:
- improveCDS: принимает на вход файл с кодирующими последовательностями и строку. Выводит в файл, названный введенной строкой, сохраняя названия последовательностей, но убирая переносы строк между нуклеотидной последовательностью.
- countStopCodons: принимает на вход файл, полученный при выполнении функции improveCDS, и булево значение (если True, то считает стоп кодоны псевдогенов, если False, то только обычных генов). Выводит список трех последних нуклеотидов каждой последовательности. Сортировка и подсчет происходит при помощи средств Bash.
Гипотетический мобильный элемент выбирался путем соотнесения наименьших длин мобильного генетического элемента и максимальных длин инвертированных повторов. Но, для всех последовательностей, содержащих транспозазы, которые идентично повторяются множество раз в геноме, выбирается, в качестве наиболее правдоподобной лишь одна.
В вышеуказанном файле Python приведены краткие комментарии о работе всех функций, использованных в работе.
2.4 Средства Bash
Средства Bash используются для следующих пунктов:
- 3.6 Старт-кодоны последовательных кодирующих последовательностей (CDS).
- 3.7 Стоп-кодоны последовательных кодирующих последовательностей (CDS).
Ссылка на файл с использованными скриптами указана в сопроводительных материалах(С6).
3 РЕЗУЛЬТАТЫ
3.1 Длины белков, закодированных в геноме бактерии Paracidovorax citrulli
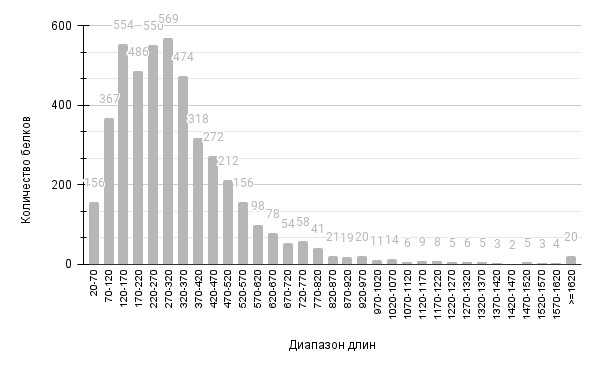
На рисунке 2 представлена гистограмма длин белков, закодированных в геноме Paracidovorax citrulli.
Из гистограммы видно, что наиболее часто встречающимися длинами белков, являются длины в диапазоне от 270 до 320 аминокислот включительно.
| Максимум | 2857 | PLxRFG domain-containing protein |
|---|---|---|
| Минимум | 26 | hypothetical protein |
| Средняя длина | 330.8 | NAD(P)H-quinone oxidoreductase (330); 2-dehydropantoate 2-reductase (331) |
| Медиана | 289 | ATP-binding cassette domain-containing protein |
В таблице 2 приведены некоторые статистические данные о длине белков бактерии. Заметим, что медианное значение немного отличается от среднего: это происходит по причине того, что среднее значение восприимчиво к статистическим выбросам, а медиана нет.
Также в таблице 2, напротив указанного минимума, стоит hypothetical protein. Наименьшим же белком, не являющимся гипотетическим, является K(+)-transporting ATPase subunit F, длин которого длина равняется 32 аминокислотам.
3.2 Распределение значений % GC в белок-кодирующих последовательностях
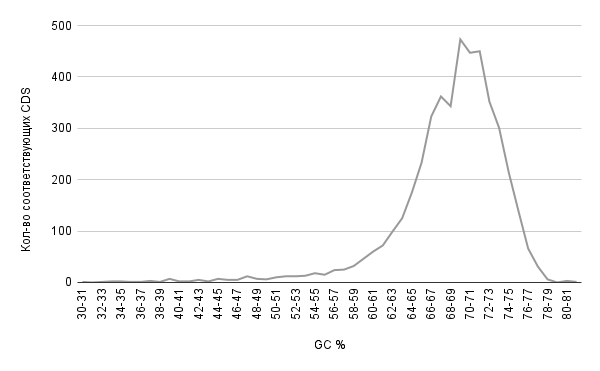
На рисунке 3 представлена гистограмма содержания GC в белок-кодирующих последовательностях.
Из рисунка видно, что наиболее часто встречаемыми значениями % GC являются значения в диапазоне 69-70 включительно.
В таблице 3 указаны некоторые статистические данные о значениях % GC.
В связи с тем, что у бактерии большой % GC, можно сделать предположение о ее термофильности. Это также обсуждается далее, в пункте 3.9 (Состав нуклеотидов A, T, G, C по репликонам бактерии Paracidovorax citrulli).
| Максимум | 81 |
|---|---|
| Минимум | 30.38 |
| Среднее значение | 68.46 |
| Медиана | 69.44 |
3.3 Расстояние между последовательными кодирующими последовательностями (CDS) на (+) и (-) цепи бактерии Paracidovorax citrulli
Распределение длин (в нуклеотидах) межгенных промежутков на (+) и (-) цепях в геноме Paracidovorax citrulli представлено на рисунках 4 и 5 соответственно.
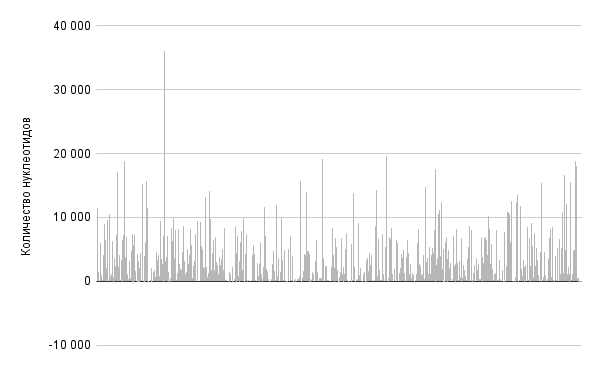
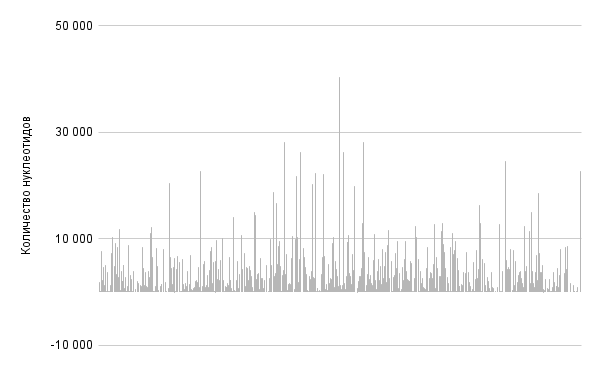
Из рисунков 4 и 5 можно сделать вывод о том, что длины межгенных промежутков не зависят от места расположения на цепи.
В таблице 4 приведены некоторые статистические данные о длинах межгенных промежутков.
На рисунках 6 и 7 представлены гистограммы расстояний между кодирующими последовательностями. Можно заметить, что существуют расстояния отрицательной длины. Это означает, что кодирующая последовательность одного белка частично перекрывает кодирующую последовательность другого белка. Перекрывания последовательностей обсуждаются ниже.
| (+)-цепь | (-)-цепь | |
|---|---|---|
| Количество | 2215 | 2144 |
| Максимум | 35996 | 40439 |
| Минимум | -113 | -100 |
| Медиана | 103 | 114 |
| Среднее значение | 1202 | 1289 |
На рисунках 6 и 7 представлены гистограммы расстояний между кодирующими последовательностями. Можно заметить, что существуют расстояния отрицательной длины. Это означает, что кодирующая последовательность одного белка частично перекрывает кодирующую последовательность другого белка. Перекрывания последовательностей обсуждаются ниже.
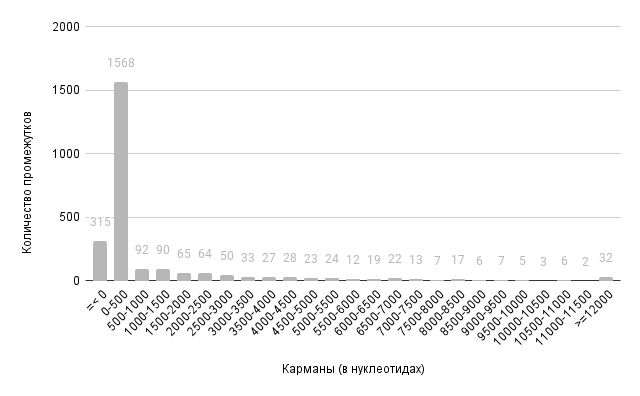
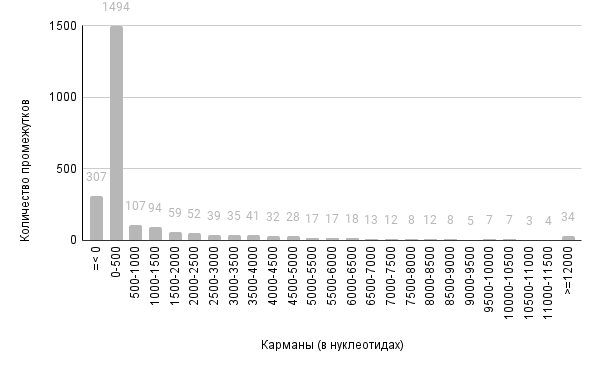
Заметим, что большинство длин межгенных промежутков лежит в пределах 0 до 500 нуклеотидов. То есть гены лежат между собой достаточно близко.
3.4 Сравнение числа генов белков и генов различных РНК хромосомы и плазмиды pAC53 бактерии Paracidovorax citrulli
В таблице 5 приведены количества генов белков и генов различных РНК на хромосоме и на плазмиде pAC53 бактерии Paracidovorax citrulli.
Как видно из таблицы, плазмида pAC53 содержит исключительно гены белков. Это можно связать с тем, что бактерии могут как принимать, так и отдавать плазмиды. То есть в них не могут содержатся гены, необходимые нормальной жизнедеятельности гены. А именно такими генами, в своем большинстве, являются гены, кодирующие различные РНК, отличные от мРНК. Поэтому скорее всего в плазмиде содержатся гены, которые не являются обязательными, но которые могут увеличивать жизнеспособность бактерии.
| (+)-цепь | (-)-цепь | |
|---|---|---|
| Количество | 2215 | 2144 |
| Максимум | 35996 | 40439 |
| Минимум | -113 | -100 |
| Медиана | 103 | 114 |
| Среднее значение | 1202 | 1289 |
3.5 Гистограмма длин пересечений последовательных кодирующих последовательностей (CDS) на (+) и (-) цепи бактерии Paracidovorax citrulli
Как мы заметили ранее, в геноме бактерии Paracidovorax citrulli расстояния межгенных промежутков могут быть отрицательными, то есть в геноме существуют перекрывающиеся гены. На рисунках 8 и 9 изображены длины пересечений последовательных кодирующих последовательностей в той последовательности, в которой они представлены в геноме бактерии Paracidovorax citrulli.
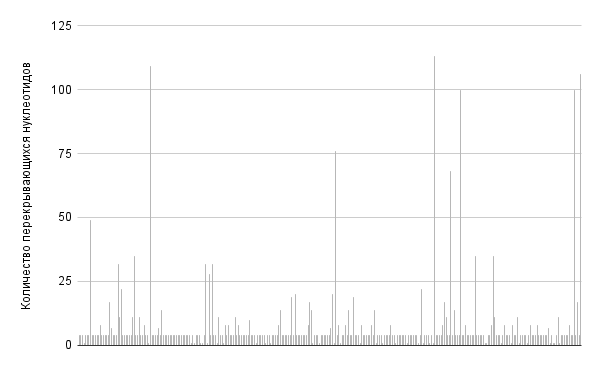
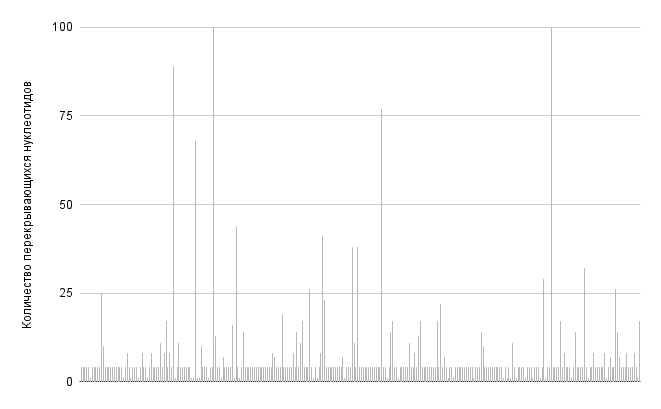
Из этих рисунков можно сделать вывод о том, что длины пересечений не зависят от места расположения на цепи.
В таблице 6 представлены некоторые статистические данные о длинах пересечений на (+) и (-) цепях хромосомы.
На рисунках 10 и 11 представлены гистограммы длин пересечений на (+) и (-) цепях хромосомы соответственно.
| (+)-цепь | (-)-цепь | |
|---|---|---|
| Количество | 2215 | 2144 |
| Максимум | 35996 | 40439 |
| Минимум | -113 | -100 |
| Медиана | 103 | 114 |
| Среднее значение | 1202 | 1289 |
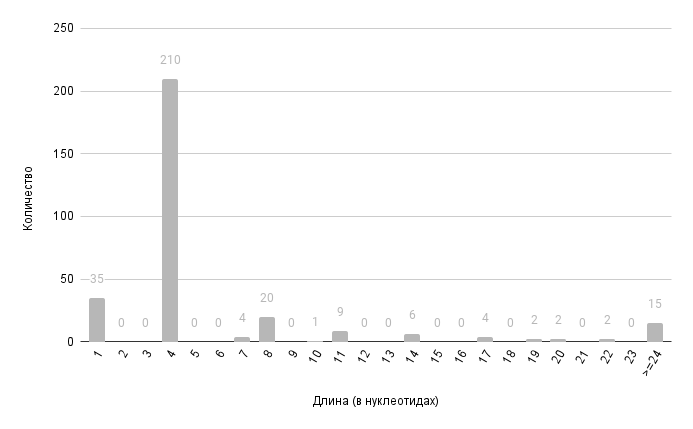
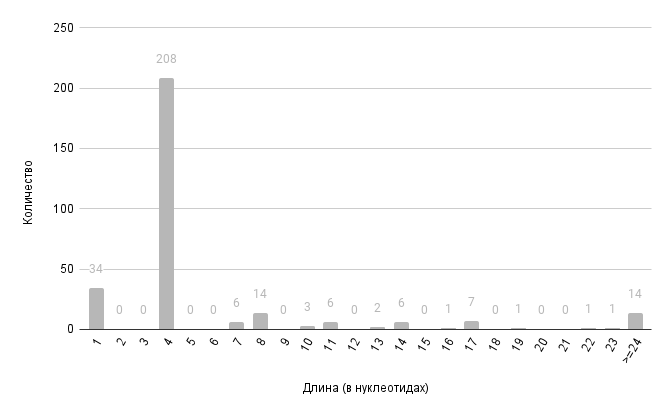
Из рисунков 10 и 11 можно увидеть, что и среднее длины, и медианное значения схожи на (+) и (-)-цепях. Можно сказать, что нет глобальных различий между пересечениями на разных цепях. Интересное наблюдение, которое можно сделать из данных рисунков заключается в том, что не встречаются пересечения, длина которых делится на 3. Это можно связать с тем, что при пересечении с длиной, делящейся на три, стоп-кодон одного гена будет попадать под рамку считывания другого, из-за чего синтез второго будет невозможен.
3.6 Старт-кодоны последовательных кодирующих последовательностей (CDS)
В таблице 7 указаны варианты старт кодонов и их количество для генов в геноме бактерии Paracidovorax citrulli.
В таблице 8 указаны варианты старт кодонов и их количество для псевдогенов в геноме бактерии.
Как мы видим из таблиц 7 и 8, количество вариантов старт кодонов у псевдогенов сильно больше чем у обычных генов (6 вариантов для обычных генов и 23 у псевдогенов).
Такую разницу можно объяснить тем, что мутации в старт кодонах обычных генов не дают синтезироваться соответствующему белку, что может приводить к снижению жизнеспособности и конкурентоспособности данной особи. Так как псевдогены не транскрибируются, то мутации в них не влияют на жизнеспособность бактерии, из-за чего у мутаций в псевдогенах больше вероятности сохраниться.
Заметим, что даже для нормальных генов (не являющихся псевдогенами), существует множество отличных старт-кодонов, отличных от “идеального” - ATG. Это связано с тем, что в начале транскрипции старт-кодон не является единственным сайтом связывания рибосомы. Вместе со старт-кодоном с рибосомой связываются близлежащая последовательность, которая находится перед старт-кодоном. Именно из-за этого “допустимо” небольшое изменение последовательности старт-кодона. Заметим также, что для генов, последовательности старт-кодонов отличаются от ATG не более чем на один нуклеотид. Кроме этого важно сказать, что изменение старт-кодона не влияет на считываемую с него аминокислоту. Из-за особенностей инициации прокариот первой аминокислотой всегда будет формил-метионин, вне зависимости от кодона.[3]
| ATG | 3963 |
|---|---|
| GTG | 312 |
| TTG | 117 |
| CTG | 15 |
| ATC | 5 |
| ATT | 3 |
| ATG | 46 |
|---|---|
| GTG; ATC | 5 |
| TTG; TTC | 3 |
| GGC | 2 |
| GCG; GCC; GAT; GAC; CTC; CGC; CGA; CCG; CCC; CCA; CAG; CAA; ATT; AGG; AGA; ACG; ACC |
1 |
| ATT | 3 |
3.7 Стоп-кодоны последовательных кодирующих последовательностей (CDS)
В таблице 9 указаны варианты стоп кодонов и их количество для генов в геноме бактерии Paracidovorax citrulli.
В таблице 10 указаны варианты стоп арт кодонов и их количество для псевдогенов в геноме бактерии Paracidovorax citrulli.
Как мы видим из таблиц 9 и 10, разнообразие вариантов стоп кодонов у псевдогенов гораздо больше, чем у обычных генов. Это можно объяснить тем, что псевдогены, в отличие от обычных генов, не считываются, а, следовательно, мутации, которые со временем в них происходят, никак не влияют на жизнеспособность бактерии и постепенно накапливаются.
| TGA | 3591 |
|---|---|
| TAG | 440 |
| TAA | 384 |
| TGA | 35 |
|---|---|
| TAG | 11 |
| GGC; CGC; CAT | 4 |
| TTC; CTG; ACC | 3 |
| GCG; GAG; CCC; ATG; AAC | 2 |
| TCG; TAA; GTT; GTC; GGG; GCT; GAT; GAC; CTT; ACG |
1 |
3.8 Гипотетические опероны в геноме бактерии Paracidovorax citrulli
При анализе промежутков между кодирующими последовательностями бактерии Paracidovorax citrulli можно заметить, что промежутки между генами белков, участвующих в одних цепях последовательных реакций или же между генами белков, являющимися субъединицами, они либо небольшие, либо эти гены вообще перекрываются. Это может послужить основанием для предположения, что данные гены находятся в одном опероне. Еще одним важным подтверждением нахождения данных генов в одном опероне может служить так называемый Прибнов-бокс (Pribnow box)- последовательность, являющаяся частью промоторного участка и лежащая примерно на 10 нуклеотидов назад от первой кодирующей последовательности оперона и обычно схожая с последовательностью TAATAT.[4]
3.8.1 Гипотетический оперон 1
Данный оперон находится на (+)-цепи хромосомы бактерии Paracidovorax citrulli и может кодировать следующие белки:
- monovalent cation/H+ antiporter subunit A
- Na+/H+ antiporter subunit C
- monovalent cation/H+ antiporter subunit D
- Na+/H+ antiporter subunit E
- K+/H+ antiporter subunit F
- monovalent cation/H(+) antiporter subunit G
Кроме этого, все вышеперечисленные субъединицы являются субъединицами одного белка.
И, наконец, примерно в 10 нуклеотидах от начала кодирующей последовательности белка monovalent cation/H+ antiporter subunit A лежит последовательность TAAGAT, которая практически идентична Прибнов-боксу (координата первого тимина предположительного Прибнов-бокса - 4564579, координата второго тимина - 4564584, а координата начала кодирующей последовательности первого гена - 4564594)
3.8.2 Гипотетический оперон 2
Данный оперон находится на (+)-цепи хромосомы бактерии Paracidovorax citrulli и может кодировать следующие белки:
- precorrin-4 C(11)-methyltransferase
- sirohydrochlorin chelatase
- precorrin-8X methylmutase
- cobalt-precorrin-5B (C(1))-methyltransferase
- bifunctional cobalt-precorrin-7 (C(5))-methyltransferase/cobalt-precorrin-6B (C(15))-methyltransferase
- precorrin-2 C(20)-methyltransferase
- cobalamin biosynthesis central domain-containing protein
- cobalamin biosynthesis protein
- energy-coupling factor ABC transporter permease
- precorrin-3B C(17)-methyltransferase
- (2Fe-2S) ferredoxin domain-containing protein
Вышеперечисленные белки участвуют в одном биохимическом пути: они являются ферментами пути синтеза кобаламина (витамин B12).[5]
На (+)-цепи хромосомы в последовательности на координатах от 1151866 до 1151881 нуклеотида можно наблюдать частую встречаемость тимина, которая может являться признаком того, что это последовательность - это Прибнов-бокс гипотетического оперона 2 (начало кодирующей последовательности первого гена - 1151885).
3.8.3 Гипотетический оперон 3
Данный оперон находится на (+)-цепи хромосомы бактерии Paracidovorax citrulli и может кодировать следующие белки:
- succinate dehydrogenase, cytochrome b556 subunit
- succinate dehydrogenase, hydrophobic membrane anchor protein
- succinate dehydrogenase flavoprotein subunit
- succinate dehydrogenase iron-sulfur subunit
- succinate dehydrogenase assembly factor 2
- citrate synthase
Первые пять белков, синтезирующихся в этом опероне являются субъединицами сукцинатдегидрогеназы. Шестой белок являются цитратсинтазой. Все эти белки участвуют в цикле Кребса.
На (+)-цепи на координатах между 4024371 и 4024376 (включительно) содержатся несколько тиминов, которые могут являться признаком, что последовательность между этими координатами является Прибнов-боксом гипотетического оперона 3 (начало кодирующей последовательности первого гена располагается на координате 4024393).
3.9 Состав нуклеотидов A, T, G, C по репликонам бактерии Paracidovorax citrulli
В таблице 11 представлены количества нуклеотидов и их процент от общего количества на (+)-цепи хромосомы бактерии Paracidovorax citrulli.
Стоит заметить, что незачем отдельно анализировать (-)-цепи хромосомы и плазмиды на состав нуклеотидов, ведь отношение сумм комплементарных нуклеотидов не изменится.
В таблице 12 представлены количества нуклеотидов и их процент от общего количества на (+)-цепи плазмиды pAC53 бактерии Paracidovorax citrulli.
Как можно понять из таблиц 11 и 12, процент A и T больше в плазмиде, а процент C и G больше в хромосоме. Доля GC в хромосоме равняется 68.9%, а в плазмиде pAC53 - 61.5%.
Связь между гуанином и цитозином образована тремя водородными связями, в то время как связь между аденином и тимином лишь двумя. Следовательно, связь GC прочнее, чем AT, то есть больший процент гуанина и цитозина может указывать на повышенную устойчивость ДНК бактерии. А это, в свою очередь, может указывать на термоустойчивость данного организма.
| Количество | % | |
|---|---|---|
| A | 759980 | 15.5 |
| T | 766684 | 15.6 |
| G | 1691030 | 34.5 |
| C | 1684538 | 34.4 |
| Количество | % | |
|---|---|---|
| A | 10783 | 20 |
| T | 9961 | 18.5 |
| G | 16351 | 30.4 |
| C | 16729 | 31.1 |
3.10 Анализ мобильных генетических элементов генома бактерии Paracidovorax citrulli
Как известно, мобильные генетические элементы можно разделить на IS-элементы и транспозоны. IS-элементы содержат в себе лишь гены, которые нужны для их перемещения. Транспозоны же помимо генов, ответственных за перемещение, содержат в себе и другие гены. Из-за переноса генов, транспозоны могут являться важным объектом наблюдения, ведь они могут переносить, например, гены резистентности к антибиотикам. [6]
Одним из главных и обязательных признаков, по которым можно найти в геноме транспозон и IS-элемент, кроме гена транспозазы, является наличие так называемых инвертированных повторов на конце мобильного генетического элемента. Инвертированные повторы - это последовательности нуклеотидов на конце мобильного генетического элемента, являющиеся комплементарными друг другу. [7]
Кроме наличия инвертированных повторов одним из важных признаков является наличие прямых повторов (direct repeats): при встраивании в геном, транспозон дублирует несколько нуклеотидов последовательности и встраивается между двумя прямыми повторами.[7]
3.10.1 Качественный и количественный анализ транспозаз в геноме бактерии Paracidovorax citrulli
В таблице 13 указаны виды транспозаз и их количество в исследуемом геноме.
Получаем, что на (+)-цепи расположено 46% генов мобильных генетических элементов, а на (-)-цепи расположено 54%. Можно предположить, что примерное равенство значений означает, что мобильные генетические элементы встраиваются в (+) и (-)-цепи с одинаковой вероятностью. Однако, данное значение не показывает как распределены по цепям отдельные виды мобильных генетических элементов. В таблице 11 приведены отношения количеств генов различных видов транспозаз на различных цепях к их общему количеству. В таблице не приведены виды мобильных генетических элементов, которые встречаются по одному разу, так как их количество не дает возможность оценить их специфичность к одной из цепей генома.
Из данных, указанных в таблице 14 можно сделать предположение, что транспозазы IS3 с большей вероятностью будут встраиваются на (+)-цепь, в то время как транспозазы IS5 и IS481 будут встраиваться с большей вероятностью на (-)-цепь. Данный факт может объясняться тем, что на одной из цепей, по каким-то причинам, находиться больше сайтов для связывания и встраивания с конкретным видом транспозазы.
Предположение нуждается в проверке путем аналогичного анализа большого количества генов транспозаз в геномах других бактерий.
| Вид | Количество в геноме | Количество на (+)-цепи | Количество на (-)-цепи |
|---|---|---|---|
| IS481-like element ISAav5 family transposase | 23 | 9 | 14 |
| IS3 family transposase | 15 | 9 | 6 |
| IS5 family transposase | 10 | 3 | 7 |
| Mu transposase C-terminal domain-containing protein | 1 | 1 | 0 |
| IS110 family transposase | 1 | 1 | 0 |
| transposase | 1 | 1 | 0 |
| Вид | Доля на (+)-цепи | Доля на (-)-цепи |
|---|---|---|
| IS481-like element ISAav5 family transposase | 39.1% | 60.9% |
| IS3 family transposase | 60% | 40% |
| IS5 family transposase | 30% | 70% |
3.10.2 Гипотетические транспозоны
При помощи написанной программы на языке Python были получены 17 гипотетических мобильных генетических элементов: получены их предположительные координаты, длины, инвертированные и прямые повторы. В таблице 15 указаны 17 гипотетических транспозонов, а также некоторая информация о них.
В ходе анализа результата случайно было обнаружено, что в геноме бактерии Paracidovorax citrulli существует 11 идентичных последовательностей длиной 1772 нуклеотида, со следующими координатами (указаны координаты начала):
(+)-цепь:
- 473508
- 699040
- 2873897
- 3162839
- 3879180
- 4067510
- 559186
- 1080373
- 2702982
- 3473128
- 4618722
Кроме последовательностей длины 1772 нуклеотида также были случайно обнаружены идентичные последовательности длиной 869 нуклеотида, со следующими координатами: (+)-цепь:
- 2178111
- 855810
- 4414640
| Вид | Длина | GC% | Переносит ли гены белков? |
|---|---|---|---|
| IS481 (232286..233556) | 1485 | 67.74 | GreA/GreB family elongation factor |
| IS3 (473508..475279) |
1772 | 69.24 | Нет |
| IS481 (575127..576123) | 997 | 66.70 | Нет |
| IS3 (699040..700811) |
1772 | 69.24 | Нет |
| IS481 (1207112..1208133) |
1022 | 67.12 | Нет |
| IS481 (1312087..1313172) |
1086 | 67.77 | Нет |
| IS481 (1574233..1575318) |
1086 | 67.50 | Нет |
| transposase (2014153..2015050) |
898 | 66.04 | Нет |
| IS5 (2067295..2068167) |
873 | 64.38 | Нет |
| IS481 (2147696..2148788) |
1093 | 65.78 | Нет |
| IS5 (2178111..2178979) |
869 | 64.44 | Нет |
| IS5 (2735615..2736486) |
872 | 64.56 | Нет |
| IS481 (2814189..2815196) |
1008 | 66.37 | Нет |
| IS3 (2873897..2875668) |
1772 | 69.24 | Нет |
| IS3 (3162839..3164610) |
1772 | 69.24 | Нет |
| IS110 (3580799..3582000) |
1202 | 64.48 | Нет |
| IS3 (3609618..3613516) |
3899 | 63.22 | 2xCdiA family toxin; hypothetical protein |
3.10.3 Пример строения транспозона
Как уже говорилось ранее, помимо IS-элементов, существуют другие мобильные генетические элементы - транспозоны. Они представляют гораздо большее значение для людей, ведь вместе с транспозонами могут переноситься различные гены, например, резистентности к антибиотикам из полученной от другой бактерии плазмиды.
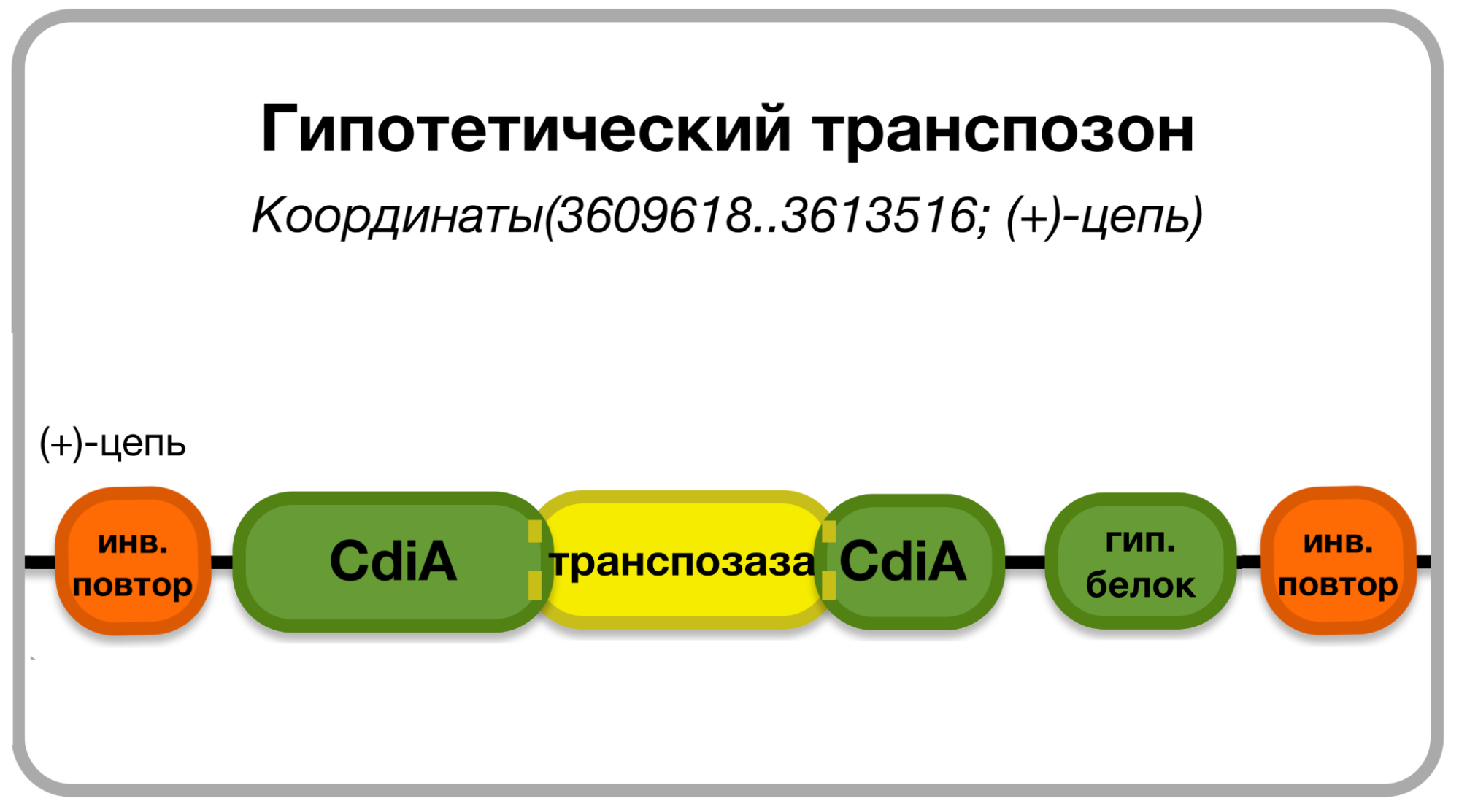
На рисунке 12 представлено строение предполагаемого транспозона находящегося по координатам 3609618-3613516 на (+)-цепи хромосомы бактерии Paracidovorax citrulli.
Он включает в себя гены следующих белков:
- Два различных CdiA family toxin C-terminal domain-containing protein
- IS3 family transposase
- hypothetical protein
У данного участка присутствуют оба признака, характерные для транспозонов: инвертированные и прямые повторы. Инвертированные повторы представлены двумя последовательностями: с 3’ стороны TGGCCGCAGGTGTGACGGCAGCC и с 5’ стороны CGCTGCCGTGAGGACAGCGGGCA. Данные последовательности комплементарны друг другу с различием в 7 нуклеотидов. Прямой повтор представлен последовательностью GCA с обеих концов гипотетического транспозона.
ЗАКЛЮЧЕНИЕ
В ходе данной работы были проанализированы как кодирующие последовательности, так и некодирующие участки генома бактерии Paracidovorax citrulli. Были выдвинуты предположения о наличии трех оперонов и термофильности бактерии.
Кроме этого, были обнаружены гипотетические IS элементы и транспозоны. Было выдвинуто предположение, о том, что эти мобильные генетические элементы изначально встроились в бактерию в единичном количестве, но потом их количество увеличилось.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
- Электронная база данных NСBI для бактерии Paracidovorax citrulli:
https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/022/493/915/GCF_022493915.1_ASM2249391v1 - Электронная таблица Google sheets “СDS from genome of Paracidovorax citrulli”.
https://docs.google.com/spreadsheets/d/14PX85vv0JvW_cIeLNdRZ3Lde-u7w1aVtqZO-knzNUio/edit?usp=sharing - Электронная таблица Google sheets “Feature table from genome of Paracidovorax citrulli”:
https://docs.google.com/spreadsheets/d/1sl8HV2bept1CfjsB3B4jxst7Beo0xwVuh3PNBvpJFR8/edit?usp=sharing - Электронная таблица Google sheets “Mobile elements in genome of Paracidovorax citrulli”:
https://docs.google.com/spreadsheets/d/1oHuvbLdZeSrssEhySHkSWkvxtvNSRAiJ8uYAeDbpqRc/edit?usp=sharing - Исполняемый файл Python:
https://drive.google.com/file/d/14bUvrEZrTNFSB0XB4q6MXLcIYjnrWBIe/view?usp=share_link - Файл со скриптами для Bash:
https://drive.google.com/file/d/1vnAk3fsfjKUMg80kSbUNxyjV13DuOVY3/view?usp=sharing
СПИСОК ЛИТЕРАТУРЫ
[1] Schaad NW, Sowell Jr G, Goth RW, Colwell RR, Webb RE. Pseudomonas pseudoalcaligenes subsp. citrulli subsp. nov. Int J Syst Bacteriol 1978;28:117–125.
[2] Parte, A.C., Sardà Carbasse, J., Meier-Kolthoff, J.P., Reimer, L.C. and Göker, M. (2020). List of Prokaryotic names with Standing in Nomenclature (LPSN) moves to the DSMZ. International Journal of Systematic and Evolutionary Microbiology, 70, 5607-5612.
[3] Lobanov AV, Turanov AA, Hatfield DL, Gladyshev VN. Dual functions of codons in the genetic code. Crit Rev Biochem Mol Biol 2010; 45(4):257-65.
[4] D. Pribnow. Nucleotide sequence of an RNA polymerase binding site at an early T7 promoter. Proc Natl Acad Sci USA . 1975 Mar;72(3):784-8.
[5] A. I. Scott , C. A. Roessner, N. J. Stolowich, J. B. Spencer, C Min, S. I. Ozaki. Biosynthesis of vitamin B12. Discovery of the enzymes for oxidative ring contraction and insertion of the fourth methyl group. Volume 331, number 1,2, 105-108 0 1993 Federation of European Biochemical Societies.
[6] Kurt P. Timmerman, Chen-Pei D. Tu. Complete sequence of IS3. Nucleic Acids Research, Volume 13, Issue 6, 25 March 1985, Pages 2127–2139.
[7] Jacques Mahillon, Michael Chandler. Insertion Sequences. Microbiol Mol Biol Rev. 1998 Sep;62(3):725–774.
[8] Aoki, S. K. (2005). Contact-Dependent Inhibition of Growth in Escherichia coli. Science, 309(5738), 1245–1248.