Семейство белков:
IS200 похожие транпозазы.
Информация о белковом семействе.
В таблице 1 указаны основные сведения о выбранном белковом семействе. Данное семейство белков является семейством транпозаз IS200 - ферментов, катализирующих перенос вставочных последовательностей (insertion sequence) и транспозонов по ДНК.
| Информация | |
|---|---|
| AC pfam | PF01797 |
| ID pfam | Y1_Tnp |
| #SEED | 77 |
| #All | 10539 |
| #SW | 4 |
| #architectures | 282 |
| #3D | 16 |
| Taxonomy | |
| #eukaryota | 0 |
| #archaea | 2073 |
| #bacteria | 51847 |
| #viruses | 68 |
Выравнивание последовательностей.
Из базы данных Pfam были скачаны и загружены в JalView выровненные последовательности для выбранного семейства белков. Информация о найденных блоках представлена в таблице 2.
| Информация | Комментарии | |
|---|---|---|
| Выравнивание seed | 77 последовательностей | |
| Максимальный достоверный блок, включающий ВСЕ последовательности. (МДБ-all) |
Колонки 67-72 | В JalView: max_d_block |
| 100% консервативные колонки в МДБ-all |
Колонка 67 [HY] - функционально | Колонки найдены при помощи окрашивания выравнивания Clustal и заданной идентичности в 100 процентов. Есть только колонка с функциальной идентичностью 100%. Кроме этого колонки 68[IVWCM], 69[H], 70[LIVMF], 71[LIVMFC], 72[ALIVF] - почти 100% функционально идентичны. |
| Максимальный достоверный блок, включающий НЕ ВСЕ последовательности. (МДБ-notAll) |
70 последовательностей; Колонки 66-91 |
В JalView: not_all_max_block |
| 100% консервативные колонки в МДБ-notAll |
Колонка 67[D], а также функционально колонки: 67[HY]; 68[IVWCM]; 84[ALIVM]; 91[TS] |
Кроме вышеописанных, есть несколько консерватинвных функционально (не 100%, но близко): 67; 70-72; 83-84; 88. |
| Участок выравнивания, в котором нет никаких достоверных подблоков, и потому маловероятно, что выравнивание на этом участке отражает ход эволюции. |
Колонки 27-29 | В JalView: non_evolution |
Карта локального сходства двух белков.
В таблице 3 указаны названия белков с доменами вышеуказанного семейства. На рисунке 1
изображена карта локального сходства (dotplot) двух последовательностей этих белков.
Координаты общего домена (PF01797):
- Q749H2_GEOSL: 1-166
- A0A1F3K7B4_9BACT: 589-736
| Информация | |
|---|---|
| Доменная архитектура 1 | PF01797 - PF08299 |
| Белок с архитектурой 1 | Q749H2_GEOSL |
| Доменная архитектура 2 | PF20465 - PF07669 - PF01797 - PF07669 - PF12950 |
| Белок с архитектурой 2 | A0A1F3K7B4_9BACT |
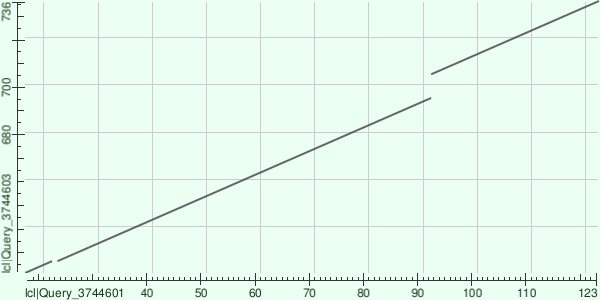
Как мы можем заметить из рисунка 1, в локальном выравнивании последовательностей
присутствуют два инделя: длиной 1 гэп и длиной 10 гэпов.
Так как в последовательности был только один общий домен, то в карте локального сходства
мы не видим каких-либо других участков последовательности.