Нуклеотидный BLAST.
Поиск δ-субъединицу АТФ-синтазы.
Как и предыдущем задании мы продолжаем анализировать геном и протеом красной водоросли Cyanidioschyzon merolae. При помощи поиска по ключевым словам "subunit" и "delta" в текстовом редакторе, была получена последовательность гена δ-субъединицу АТФ-синтазы.
Файл с белковой последовательностью: atpase_delta_subunit.fasta.
Файл с нуклеотидной последовательностью: nucleotide_atpase_delta_subunit.fasta.
Запись в базе данных Protein NCBI: XP_005537275
Запись в базе данны Nucleotide NCBI: XM_005537218
Gene ID: 16995353
Запись локуса в базе данных Nucleotide NCBI: NC_010140.1
На рисунке 1 увазано расположение упомянутого выше гена относительно соседних генов на 14 хромосоме.
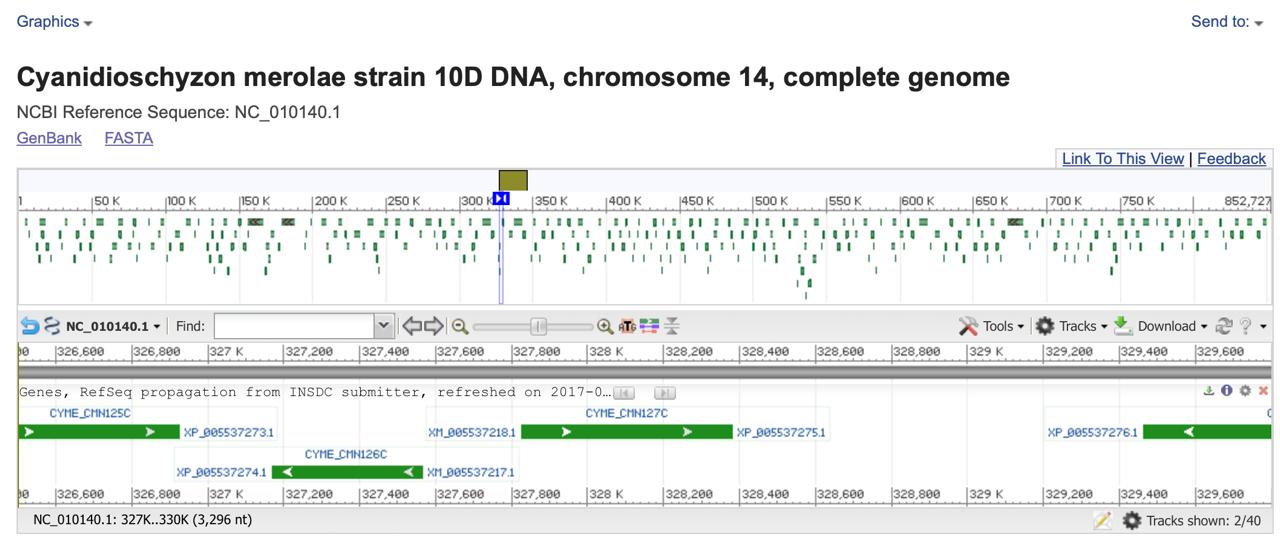
mitochondrial F-type ATPase F1 subunit delta, precursor NC_010140 - LOCUS nucleotide_atpase_delta_subunit.fasta .
Поиск гомологичных последовательностей у далеких организмов.
Для поиска последовательностей гомологичных δ-субъединице АТФ-синтазы выбранной красной водоросли
были использованы два различных способа: blastn и tblastn. Использовался именно blastn, а не высокоточный
megablast, так как вероятнее всего из-за дальней родственности второй алгоритм не нашел бы ничего.
tblastn был выбран вместо tblastx, так как нам уже достоверна известна аминокислотная
последовательность субъединицы.
blastn был запущен с длиной слова 7 (минимально допустимая) и порогом e-value=1 (лучшая
находка имеет e-value 0.53). Графический результат показан на рисунке 2.
tblastn был запущен с длиной слова 2, остальное оставлено без изменений. Графический результат
показан на рисунке 3.
blastn и tblastn проводились по базе RefSeq геномов семейства Aranae (Пауки).
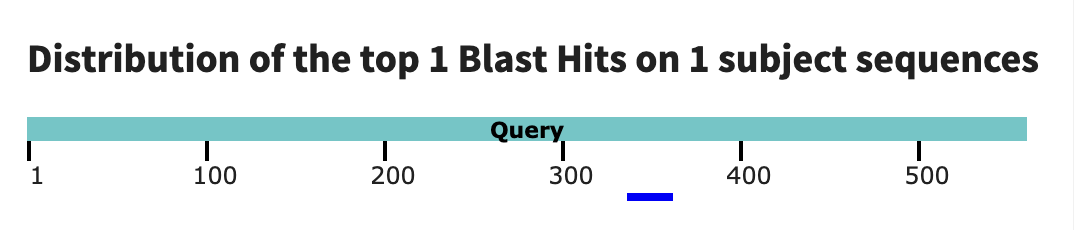
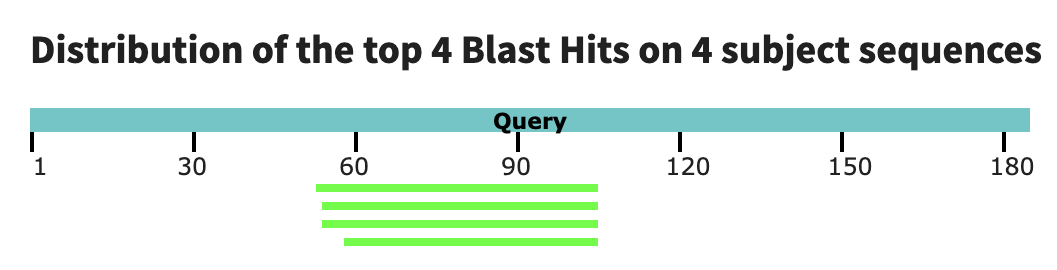
Как мы видим, даже лучшая находка blastn имеет очень большое e-value. Однако tblastn
позволил найти во всех 4 базах данных (Parasteatoda tepidariorum, Stegodyphus dumicola,
Uloborus diversus, Argiope bruennichi).
Можно объяснить этот факт следующим образом: нуклеотидная последовательность
всегда менее консервативна, чем аминокислотная последовательность. Поэтому по последовательности
нуклеотидов мы уже не можем достоверно найти гомологов, а по аминокислотной все еще можем.
Поиск рРНК красной водоросли Cyanidioschyzon merolae по гомологам из E.Coli
Для того, чтобы осуществить поиск по последовтельностям рРНК из E.Coli нужно проиндексировать геном нашей красной водорсли:
makeblastdb -in GCF_000091205.1_ASM9120v1_genomic.fna -dbtype nucl
Далее при помощи программы blastn мы провели поиск одновременно по обеим рРНК E.Coli:
blastn -task blastn -query files/rRNA_ecoli.fasta -db GCF_000091205.1_ASM9120v1_genomic.fna -out rrna_red_algae.txt -word_size 4 -evalue 0.05
Результат представлен в вайле: rrna_red_algae.txt.
Убрав из выдачи статистически незначимые выравнивания, можно увидеть, что в геноме по 3 гомолога и для 16S рРНК (две на 17 хромосоме и одна на 18 хромосоме) и для 23S рРНК (две на 17 хромосоме и одна на 18 хромосоме), которые выравниваются одинаковым образом.
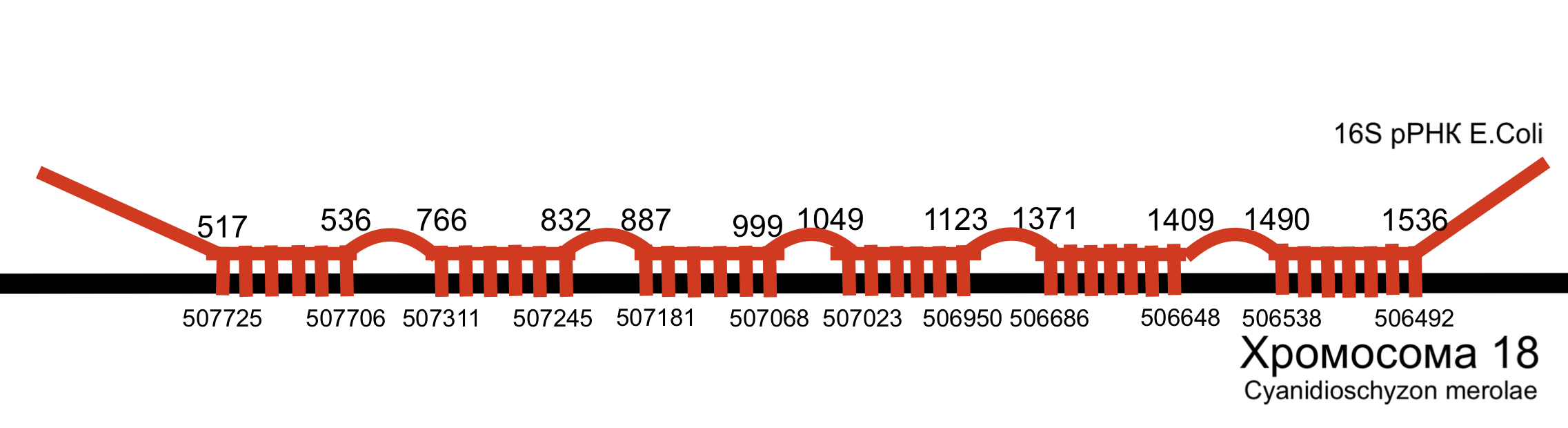
На рисунке 4 показана визуализация выравнивания 16S рРНК и одного из гомологов.
Как видно из рисунка, рРНК выравнялись некоторыми участками. Можно предположить, что это связано с тем, что выравнялись те участки, которые которые непосредственно участвует во взаимодействии с мРНК.Пример выравнивания 23S рРНК (одной из трех гомологов)
2442 2613 500837 500665
2222 2266 501059 501015
1899 1983 501407 501323
1674 1711 501724 501687
431 493 503369 503307
198 279 503655 503574
Пример выравнивания 16S рРНК (одной из трех гомологов)
1490 1536 506538 506492
1371 1409 506686 506648
1049 1123 507023 506950
887 999 507181 507068
766 832 507311 507245
517 536 507725 507706
Карты локального сходства двух штаммов бактерии Paracodovrax citrulli.
Для построения карт локального сходства были выбраны два штамма бактерий вида Paracodovrax citrulli, имеющие нуклеотидные записи в NCBI NZ_CP023687.1 и NZ_CP042323.1. Используя эти записи были построены карты локального сходства при помощи алгоритмов megablast и blastn. Также была осуществлена попытка построить карту при помощи tblastx, но видимо из-за размера генома и избытка образующихся рамок считывания, построить карту не удалось. Карты показаны на рисунках 5 и 6 для megablast и blastn соответственно.
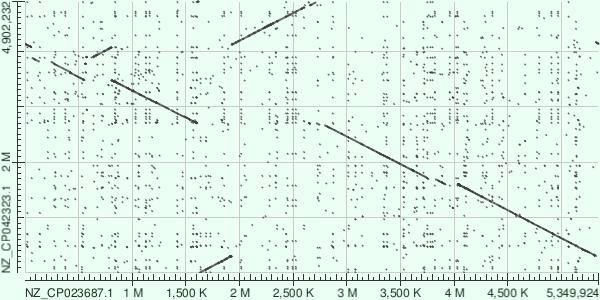
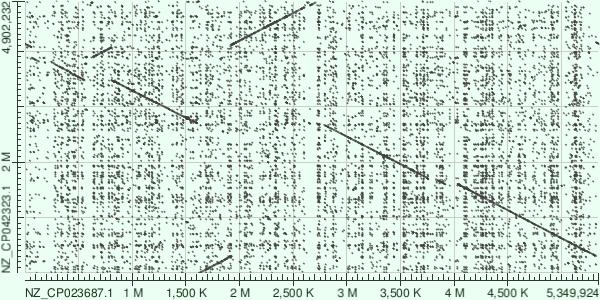
Судя по тому что цепь идет сверху вниз, в этим штаммах были выбраны разные цепи как основные. При этом, кажется, точки начала были выбраны одинаковые. На графиках также можно видеть транслокацию и инверсию.