ПРАКТИКУМ 11
Гомология и выравнивание
Из таблицы доменов Pfam я выбрала PF01701 Photosystem I reaction centre subunit IX / PsaJ. Ниже можно видеть таблицу характеристик.
| AC pfam | ID pfam | #SEED | #All | #SW | #architectures | #3D | Taxonomy |
|---|---|---|---|---|---|---|---|
| PF01701 | Photosystem I reaction centre subunit IX / PsaJ | 28 | 14k | 172 | 11 | 108 | 21k |
Функции домена
- Входит в состав реакционного центра фотосистемы I у фотосинтезирующих организмов
- Взаимодействует с субъединицей PsaF реакционного центра
- Предполагается, что играет роль в поддержании правильной ориентации PsaF, что обеспечивает эффективный перенос электронов от растворимых донорных белков к реакционному центру P700+
- Играет важную роль для нормального функционирования фотосистемы I
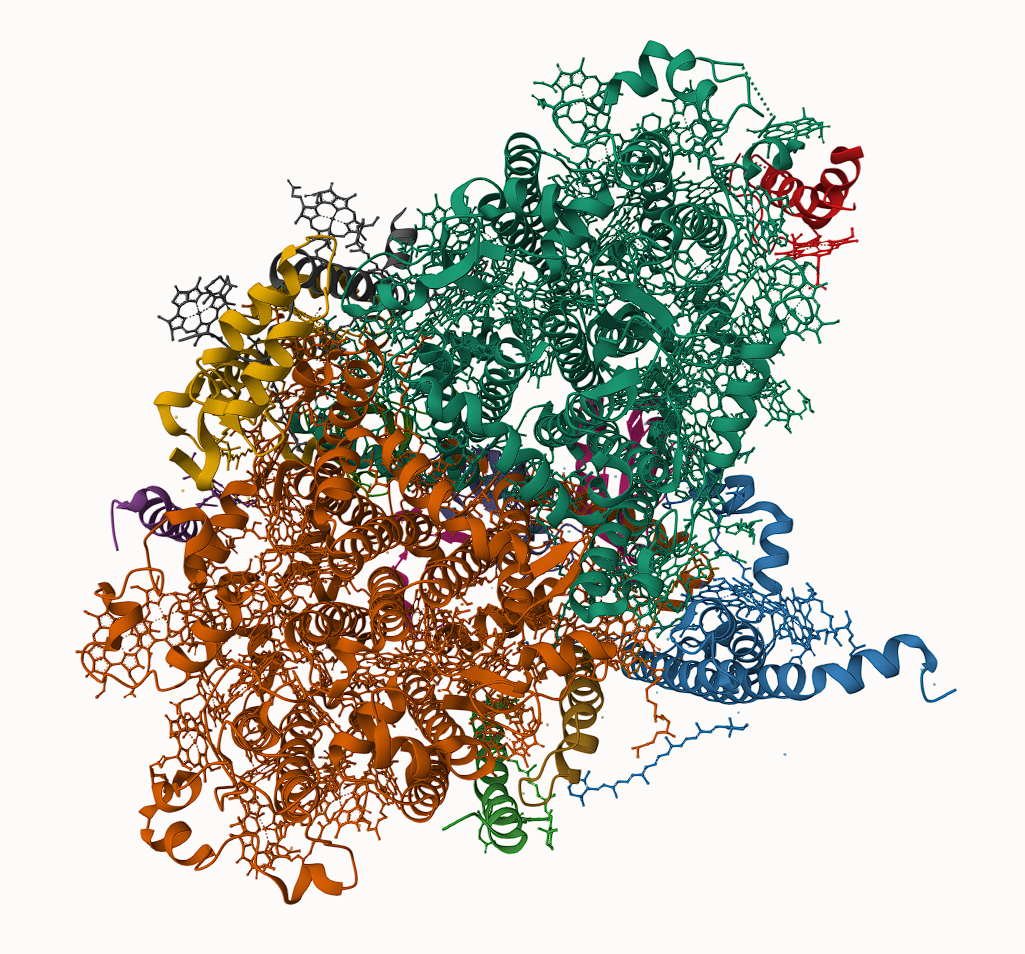
3Д структура
Таксономическая распространённость
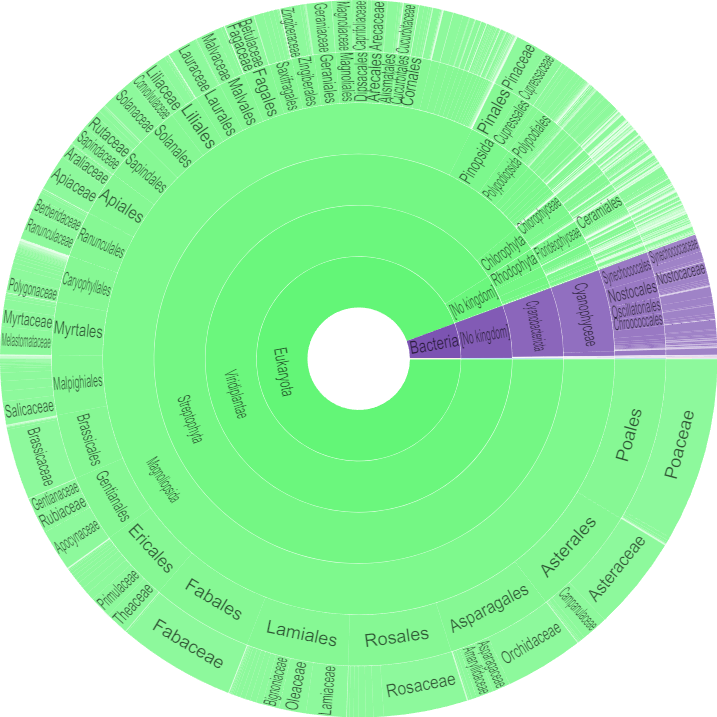
Эукариоты составляют 94.42% из всех видов-обладателей белков с данным семейством доменов, бактерии - 5.58%
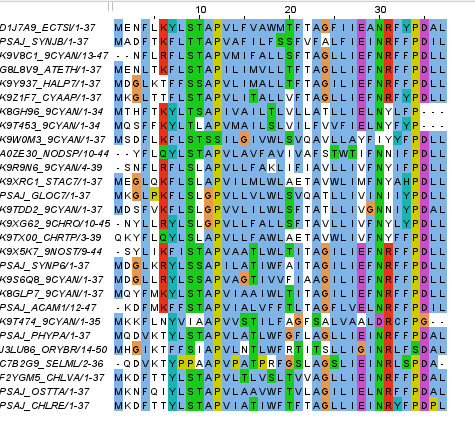
Bыравнивание seed с точки зрения гомологичности всех последовательностей или их подмножества
| Характеристика | Информация |
|---|---|
| Выравнивание seed | последовательностей - 28, колонок - 37 |
| Максимальный достоверный блок, включающие ВСЕ последовательности (МДБ-all) | 6 - 34 |
| 100% консервативные колонки в МДБ-all | 12:[P] (27 из 28), 30:[N] (26 из 28), 34:[P] (27 из 28) |
| Максимальный достоверный блок, включающий НЕ ВСЕ последовательности. (МДБ-notAll) | 7 - 19 |
| 100% консервативные колонки в МДБ-notAll | 1:[M], 25:[L], 33:[F] |
| Участок выравнивания, в котором нет никаких достоверных подблоков, и потому маловероятно, что выравнивание на этом участке отражает ход эволюции. | 1 - 5 |
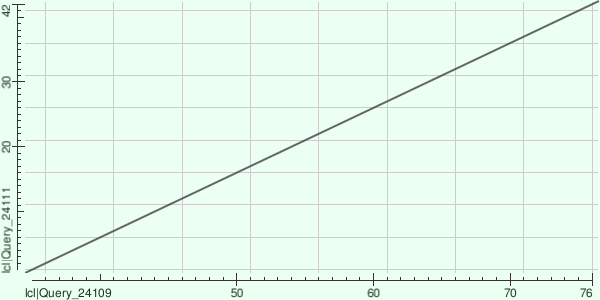
Карта локального сходства (dotplot) двух последовательностей с разной доменной архитектурой.
| Характеристика | Информация |
|---|---|
| Доменная архитектура 1 | PF01701 |
| Белок с архитектурой 1 | P12191 |
| Доменная архитектура 2 | PF02529 - PF01701 |
| Белок с архитектурой 2 | A0A0R0G5A8 |
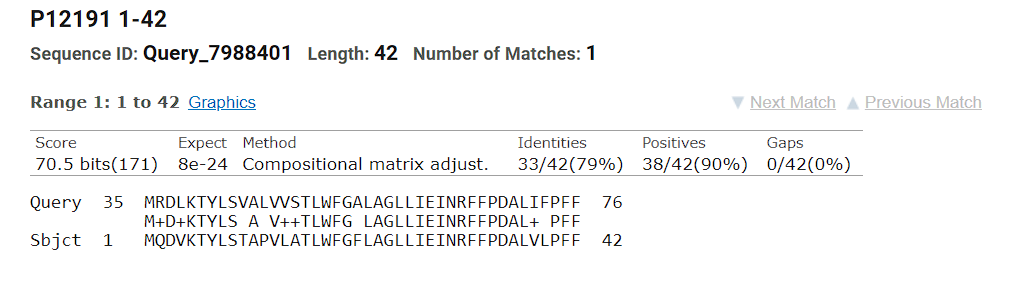
E-Value: 8e-24
Per. Ident.: 78,57%
Query Cover: 53%
По диаграмме DotPlot (рис.5.) можно сделать
вывод, что отсутствие разрывов на диаграмме указывает на тот факт, что выравнивание не содержит инделей, а
данные области очень похожи друг на друга. А также стоит заметить, что выровнялись только последовательности
доменов.