Выравнивание последовательностей
Глобальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| Acylphosphatase | ACYP_ECOLI | ACYP_BACSU | 141.5 | 32.4% | 45.4% | 33 | 3 |
| Citrate synthase(/synthase 1) | CISY_ECOLI | CISY_BACSU | 489.0 | 28.4% | 47.6% | 65 | 12 |
| Dihydrofolate reductase | DYR_ECOLI | DYR_BACSU | 359.0 | 41.7% | 60.7% | 9 | 2 |
Локальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Acylphosphatase | ACYP_ECOLI | ACYP_BACSU | 150.0 | 42.9% | 59.7% | 5 | 1 | 88.04% | 92.31% |
| Citrate synthase(/synthase 1) | CISY_ECOLI | CISY_BACSU | 497.0 | 31.7% | 52.9% | 30 | 10 | 97.19% | 97.81% |
| Dihydrofolate reductase | DYR_ECOLI | DYR_BACSU | 359.0 | 43.8% | 63.8% | 1 | 1 | 100% | 95.24% |
Программы выравнивания неродственных белков
| Выравнивание | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| Needle | BLC_ECOLI | SBOA_BACSU | 14.0 | 1.4% | 2.9% | 196 | 2 |
| Water | BLC_ECOLI | SBOA_BACSU | 22.5 | 33.3% | 38.1% | 10 | 1 |
Данное выравнивание показывает, что анализируемые белковые последовательности не являются гомологичными. Об этом свидетельствуют следующие факторы:
Низкий вес выравнивания
Низкий процент сходства и идентичности
Большое количество гэпов в выравнивании
Таким образом, данные результаты свидетельствуют, что исследуемые белки не являются гомологичными.
Множественное выравнивание
Для выполнения этого пункта практикума я искала все белки, которые имеют мнемонику "ACYP". Полное имя: "Acylphosphatase". Таких нашлось 248 штук. Далее я случайным образом отобрала ACYP_MOOTA, ACYP_GRABC, ACYP_PASMU, ACYP_LISMF, ACYP_STAAR
(команды: "infoseq 'sw:ACYP_*' -only -name -nohead -out protein.txt" , "wc -l protein.txt")
Далее в программе Jalview я провела выравнивание методом "Muscle with Defaults" и окрасила методом "by Percentage Identity".
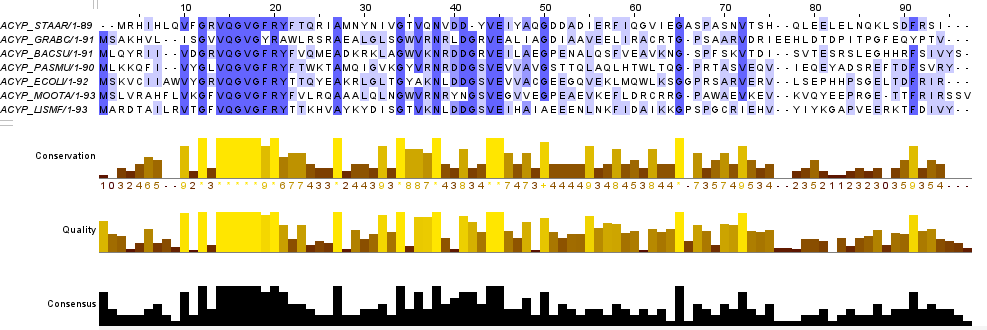
-
Рис. 1.
Множественное выравнивание
Белки являются гомологичными, если судить по результатам выравнивания, так как имеют большие консервативные участки: 14-21, а также менее консервативные: 41-42, 44-45. Ссылка на проект