Практикум 10
Поиск в нуклеотидных банках по аннотации. Выравнивание геномов
Для выполнения данного практикума я выбрала бактерию Mycobacterium tuberculosis, который относится к роду Mycobacterium
Поиск последовательностей проводила в "сборной" банке "Nucleotides" на NCBI (ссылка на ресурс) с помощью advanced search по запросу: ((chromosome[Title]) AND Mycobacterium tuberculosis[Organism]) AND RefSeq
Было найдено 654 записей, из которых я выбрала штаммы:
1. Mycobacterium tuberculosis strain PSNK363; Accession: NZ_CP143267
2. Mycobacterium tuberculosis strain AST-T2; Accession: NZ_CP133040
Далее я построила карты локального сходства с помощью megablast с длиной слова 28 и blastn с длиной слова 11, оставшиеся параметры я оставила стандартными. Карты локального сходства представлены дальше.
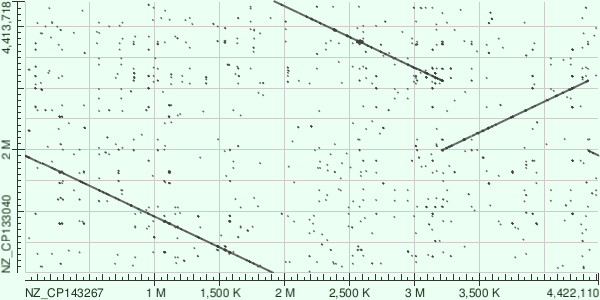
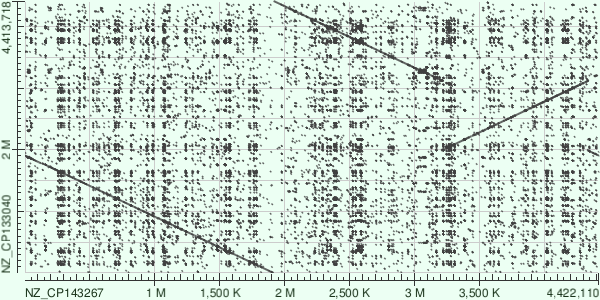
Анализ DotPlot
Прерывания в линиях, которые связаны с делецией и инсерцией не наблюдаю. Потом делаю вывод, что делеции и инсерции в выравнивании отсутствуют. Также не наблюдаю смещений линий, то есть отсутствуют транслокации. На графике можно четко увидеть сплошные линии, которые свидетельствуют о наличии гомологичных участках. Но также на графике можно наблюдать инверсию. На графике, полученный алгоритмом megablast видно меньшее количество точек, что свидетельствует о более жестком пороге выравнивания. Это указывает на то, что megasblast ориентирован на нахождение только очень высококонсервативных областей. В свою очередь на графике blastn наблюдается гораздо больше точек на графике, что указывает на более мягкие критерии для выравнивания. Четкие диагональные линии сохраняются, но появляется больше точек в областях, где сходство менее очевидное. Это указывает на способность blastn обнаруживать и более слабые гомологии. Из вышесказанного могу сделать вывод, что megablast подходит для анализа высококонсервативных участков. Диагональные линии более четкие и изолированные, что упрощает интерпретацию ключевых сходств. А алгоритм blastn показывает больше деталей, включая вариации и потенциальные слабые гомологии. Сложнее интерпретировать график из-за «шума», вызванного большим количеством выравниваний.