- Алгоритм megablast.
- Алгоритм tblastx.
Нуклеотидный Blast
Выбранный участок генома
Я произвела поиск в файле последовательностей белков последовательности δ-субъединицы АТФ-синтазы по запросу: ATP synthase subunit delta. В результате поиска нашла белок с идентификатором: XP_009432540.1/1-168 ATP synthase subunit delta, mitochondrial [Pan troglodytes]
Файл с последовательностью δ-субъединицы АТФ-синтазы
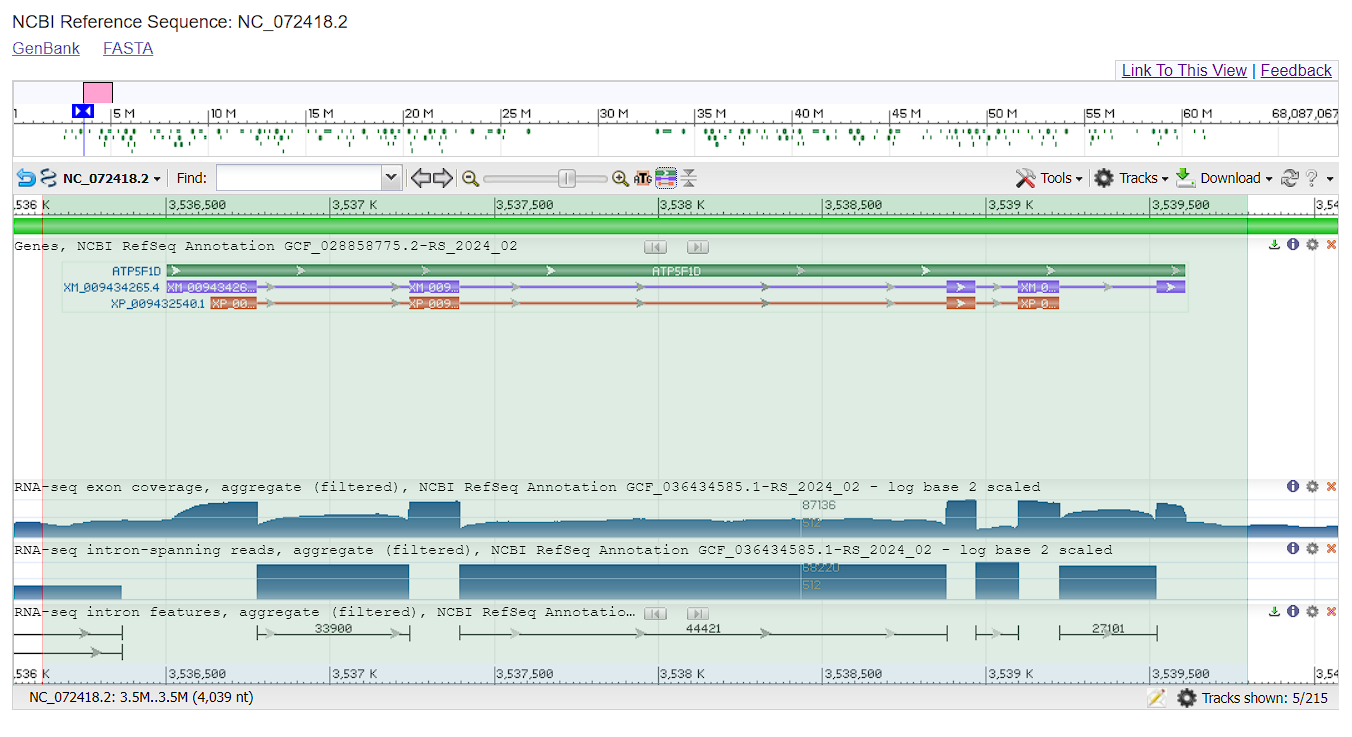
Далее я искала в нуклеотидной последовательности генома идентификатор записи нуклеотидной последовательности. В результате поиска я выяснила, что идентификатор нуклеотидной записи - NC_072418.2 (ссылка на запись) и что ген находится в 20 хромосоме. Далее я выбрала участок с координатами 3536120..3539799 (3,680 п.н.) из 20 хромосомы. На этом фрагменте ДНК расположен CDS XP_009432540.1, в котором аннотирован ген ATP5F1D (3,107 п.н.).
Файл с окрестностью гена ATP5F1D
Поиск BLAST в NCBI
Для выполнения данного задания необходимо было выбрать достаточно удаленное от моего организма таксон. Так как обыкновенный шимпанзе [Pan troglodytes] относится к вторичноротым, то таксон выбирался среди первичноротых. В итоге я пришла к тому, что решила провести поиск среди пауков (Araneae). Поиск я решила провести с помощью двух типов алгоритмов: blastn и tblastn. В качестве базы данных для поиска была взята база данных RefSeq Genome Database.
Поиск по алгоритму megablast с длиной слова 28 выдало 5 находок, в среднем 90% идентичности. Такой результат предполагает, что найденные последовательности имеют относительно высокий уровень идентичности с искомой, но при этом могут быть различия в отдельных сегментах. В целом в окрестностях этого числа находок я и ожидала результат
Поиск по алгоритму tblastx с длиной слова 3 выдало 20 находок, в среднем от 40% до 60% идентичности. Учитывая тот факт, что пауки и шимпанзе значительно отличаются, то подобный результат говорит о недостаточной консервативности последовательности гена, ответственного за кодирование дельта субъединицы АТФ-синтазы. Это вызывает сомнения в высокой степени совпадения между их генетическим материалом.
Поиск в геноме генов основных рибосомальных РНК по далекому гомологу
Чтобы проиндексировать последовательность генома, я воспользовалась следующей программой:
makeblastdb -in GCF_028858775.2_NHGRI_mPanTro3-v2.0_pri_genomic.fna -dbtype nucl
Далее я осуществила локальный поиск BLAST по 16S и 23S рРНК Escherichia coli. 16S рРНК выполняет структурную функцию, выступая в качестве каркаса малой субъединицы рибосомы прокариот, а так же на 3'-конце содержит последовательность анти-Шайна-Дальгарно, с помощью которой связывается с мРНК. 23S рРНК входит в состав большой субъединицы рибосомы прокариот и входит в каталитический пептидилтрансферазный центр на рибосоме
Я решила использовать blastn, так как нужно было сравнить нуклеотидные последовательности неблизкородственных организмов. В качестве парметров я взяла evalue 0.05, чтобы отсеять плохие находки, а также outfmt 7, чтобы получить результаты в виде таблицы с комментариями
Команды:
blastn -task blastn -evalue 0.05 -query 16S.txt -db GCF_028858775.2_NHGRI_mPanTro3-v2.0_pri_genomic.fna -out blastn_16S.txt -outfmt 7
blastn -task blastn -evalue 0.05 -query 23S.txt -db GCF_028858775.2_NHGRI_mPanTro3-v2.0_pri_genomic.fna -out blastn_23S.txt -outfmt 7
Результаты выполнения команд:
Для 16S рРНК 39 находок, среди которых 38 гомологов: файл с выдачей
Для 23S рРНК 158 находок, среди которых примерно 153 гомологов: файл с выдачей