Практикум 10. Мотивы в белках паттерны и PSSM для их поиска
Задание 1. Найти консервативные мотивы в выравнивании
Для выполнения данного практикума с учетом заданных критериев я выбрала домен PF00320 - GATA zinc finger
В выравнивании seed имеется 56 последовательностей, а в SwissProt 165 находок с этим доменом. Далее я скачала это выравнивание с сайта InterPro. Загрузила выравнивание в программу JalView. После удаления избыточных последовательностей, которые были на 90% идентичные, последовательностей осталось все также 56 штук, то есть последовательности идентичные на 90% отсутствуют.
Далее я выбрала Color Clustal и выставила Above identity threshold 100%. Выделились 4 изолированных консервативных колонок. Продолжала снижать порог пока не выделился мотив. Наиболее консервативным мне показался мотив, расположенный на позициях 29 - 34. Мотив проявился на значении в 73% (Рис. 1)
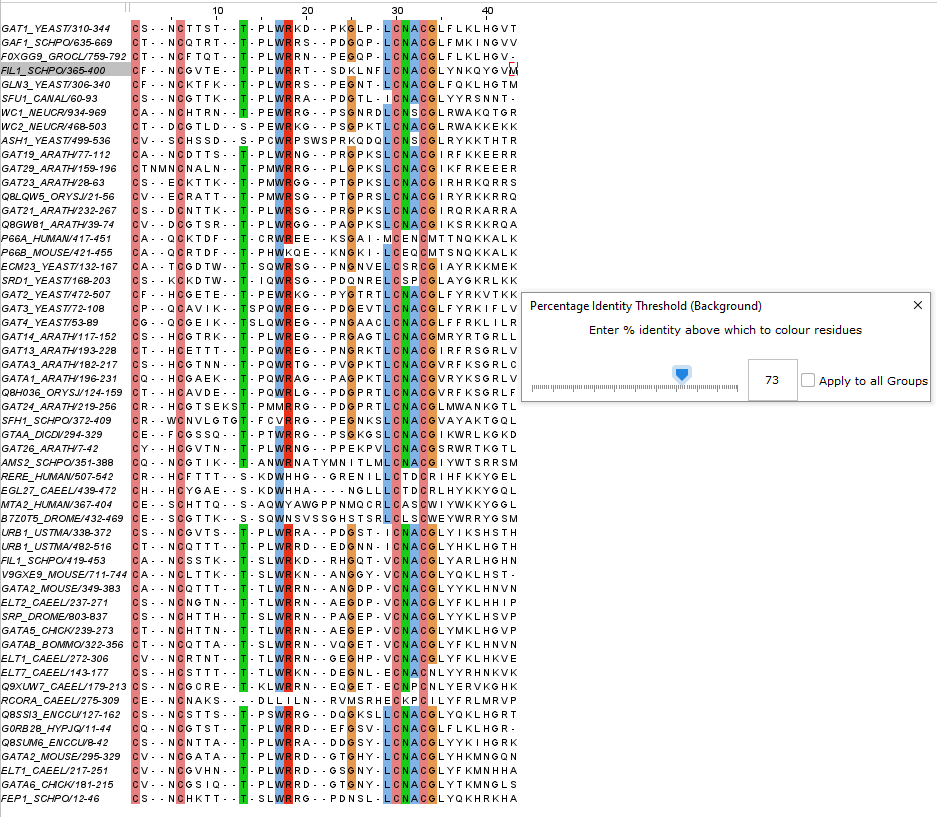
Составила паттерн jalView: [LIMVE]C[NESTALK][ASNQRPD]C[GMRWNI]. Выполнила поиск паттерна во всех выравнивании. Было найдено 56 находок из 56 последовательностей всего, все друг под другом (Рис. 2).
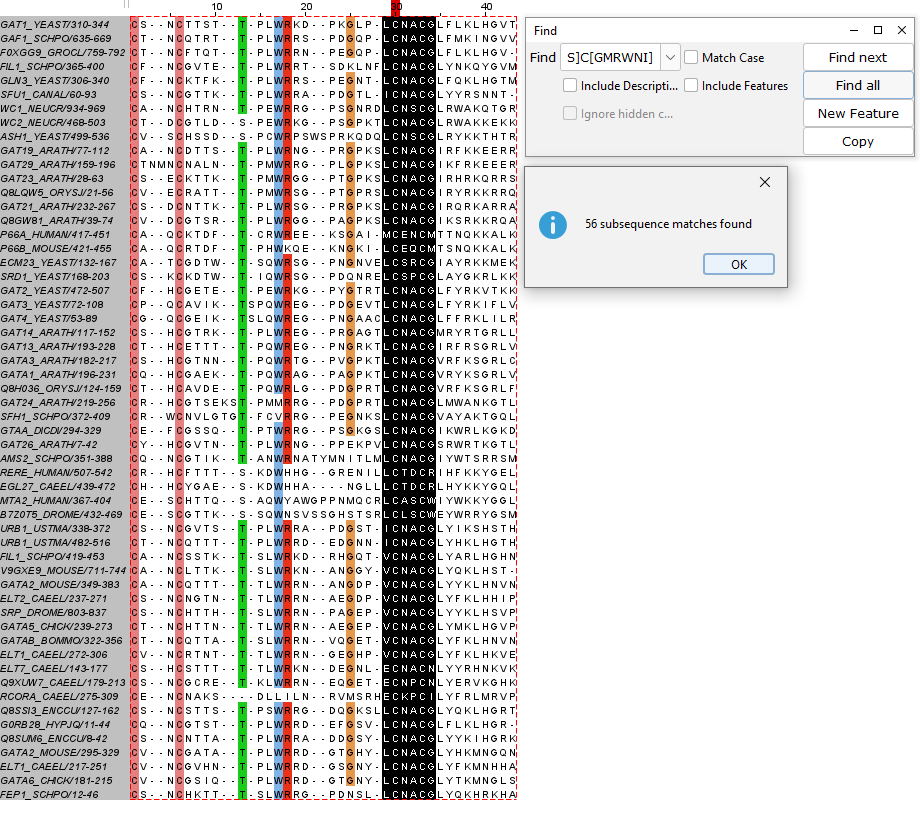
Перевела паттерн JalView в формат Prosite: [LIMVE]-C-[NESTALK]-[ASNQRPD]-C-[GMRWNI]. Далее я провела поиск по паттерну в формате Prosite в базе данных SwissProt, используя инструмент scanProsite. Поиск выдал мне 1065 находок в 1000 последовательностях. Выдача поиска ScanProsite. После поиска мотива в выравнивании этих последовательностей видно, что мотив не находится в выравнивании друг под другом и также распознается в большем количестве мест, чем всего последовательностей.
Задание 2. Мотив, специфичный для одной клады филогенетического дерева
Для выполнения этого задания построила филогенетическое дерево методом NJ и выбрала кладу (выделена синим цветом на Рис. 3).
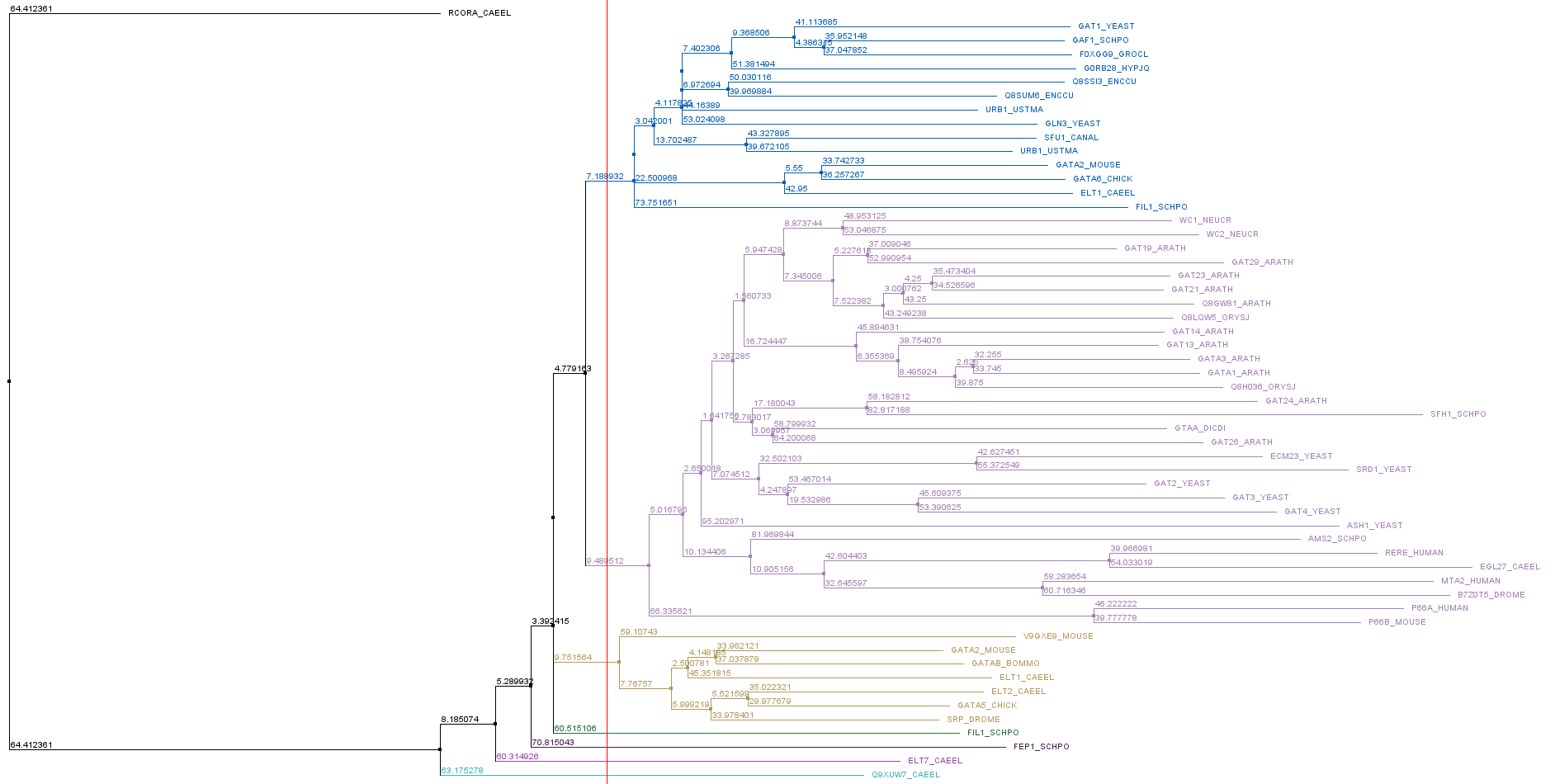
После того как выбрала кладу, я выделила последовательности этой клады в отдельное окно и нашла мотив: P[LS]WR[KR]. Провела поиск паттерна во всех выравниваниях. Этот мотив нашелся только в последовательностях, входящих в кладу. Делаю из этого вывод, что этот мотив специфичен для данной клады (Рис. 4).
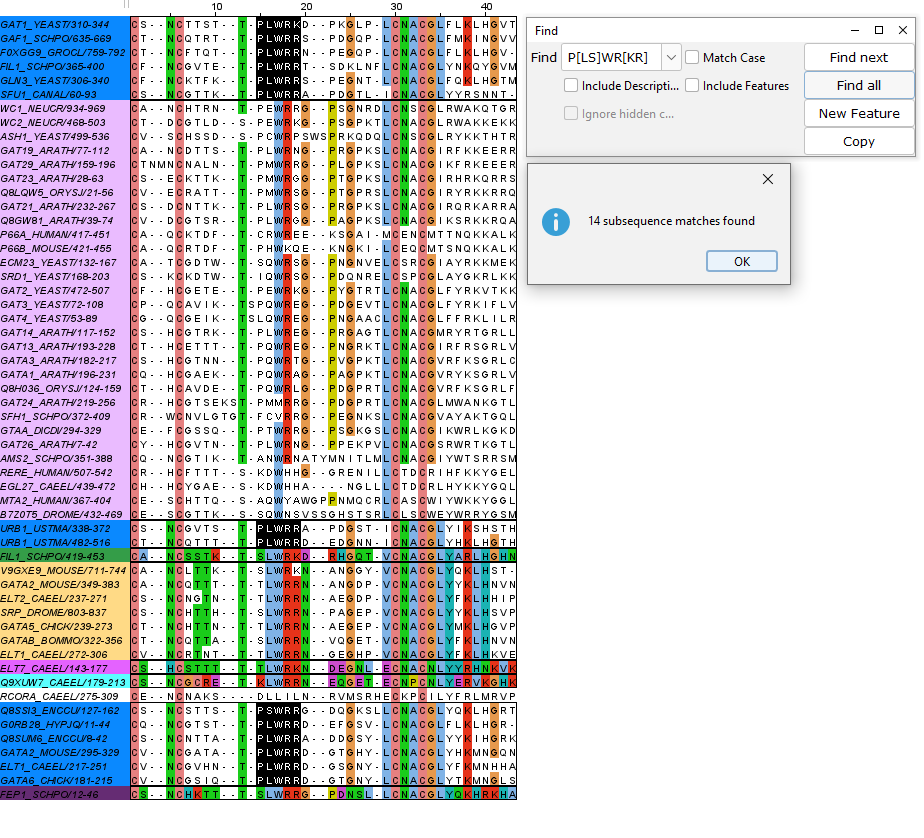
Задание 3. PSI-BLAST
Я из списка выбрала AC: P17265. Организм - Rhizobium meliloti (strain 1021) (Ensifer meliloti) (Sinorhizobium meliloti). Функция белка - ribosome hibernation promotion factor (необходим для димеризации активных 70S рибосом в 100S рибосомы в стационарной фазе; рибосомы 100S трансляционно неактивны и иногда присутствуют во время экспоненциального роста.).
Запустила алгоритм PSI-BLAST с поиском по базе данных swissprot.
| Номер итерации | Число назодок выше порога (0,005) | Идентификатор худшей нахожки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 17 | P0A147.1 | 7e-04 | P26983.1 | 0,028 |
| 2 | 27 | P33987.1 | 1e-08 | 0 | 0 |
| 3 | 28 | P9WMA8.1 | 0,002 | 0 | 0 |
После третьей итерации алгоритма в таблицу находок перестали добавляться новые.
По какой-то причине E-value худшей находки выше порога на второй итерации понизился, а потом повысился на несколько порядков. Я перепроверила дважды: в обоих случаях так происходило. В любом случае скачки E-value происходят в несколько порядков, а среди находок BLAST большинство белков имеют ту же функцию, что и взятый изначально. Только на третьей итерации в таблицу добавляются белки иных функций (P0AD49.2, P71346.3). Не факт, что они не гомологичны исходному.
Таким образом, факторы димеризации рибосом образуют хорошо идентифицируемое вероятностными методами семейство.
Задание 4. Поиск в выборке поледовательностей с доменом из SwissProt de novo мотивов с помощью MEME
Для этого задания я скачала 165 последовательностей белков с выбранным доменом из SwissProt. Из этого числа выбрала 75 последовательностей и запустила поиск мотивов с помощью программы MEME в этих последовательностях:
meme PF00320_seq75.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4
Далее запустила программу FIMO, чтобы провести поиск найденных мотивов в изначальном наборе из 165 последовательностей.
fimo meme_out/meme.txt protein-matching-PF00320.fasta
Выдача FIMO: html
Было найдено 819 вхождений мотивов в 165 последовательностях. Это значит, что некоторые мотивы встречаются в последовательностях несколько раз.
Задание 5. Оценка представленности сайта GATC в геноме бактерии
Моей бактерией является Coxiella burnetii RSA 493. Для проведения оценки представленности всех 24 четырехнуклеотидных сайтов без повторений нуклеотидов в геноме моей бактерии была использована команда:
cbcalc -s sites.txt -M -o result.tsv GCF_000007765.2_ASM776v2_genomic.fna
-s sites.txt - файл с четырехнуклеотидными сайтами
-М - метод Карлина
-o result.tsv - имя выходного файла
По результатам команды была построена гистограмма (Рис. 5.)
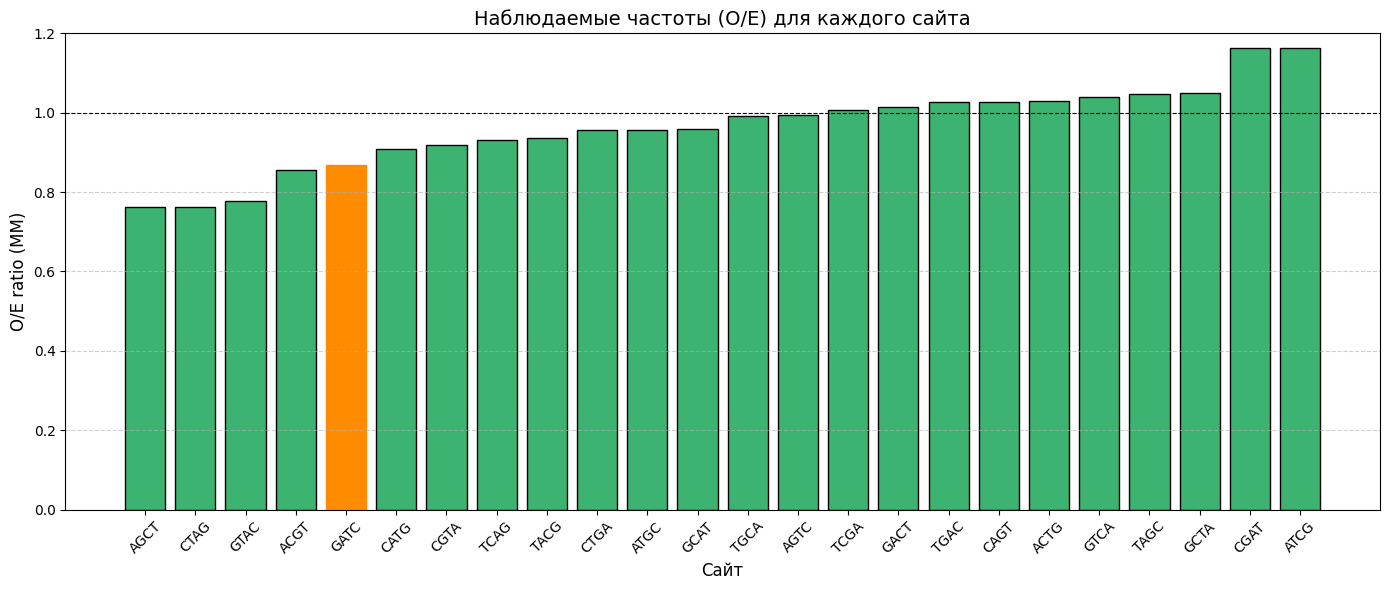
Исходя из результатов и гистограммы видно, что относительная представленность для сайта GATC равна 0,867. Учитывая тот факт, что это один из самых низких значений относительной представленности по сравнению с другими сайтами, можно сделать вывод, что метилирование данного сайта не играет важную роль в клеточных процессах в моей бактерии.