Практикум 2. Алгоритмы филогенетической реконструкции.
В данном практическом задании необходимо построить филогенетические деревья двумя разными программами, анализирующие последовательности цитохромов B, выровненные друг с другом.
На kodomo было построено выравнивание цитохром В с помощью программы muscle. Далее я построила деревья с помощью двух программ: fastme (с оценкой расстояний по MtREV и p-distance) и iqtree. После построения я визуализировала деревья в iTol.
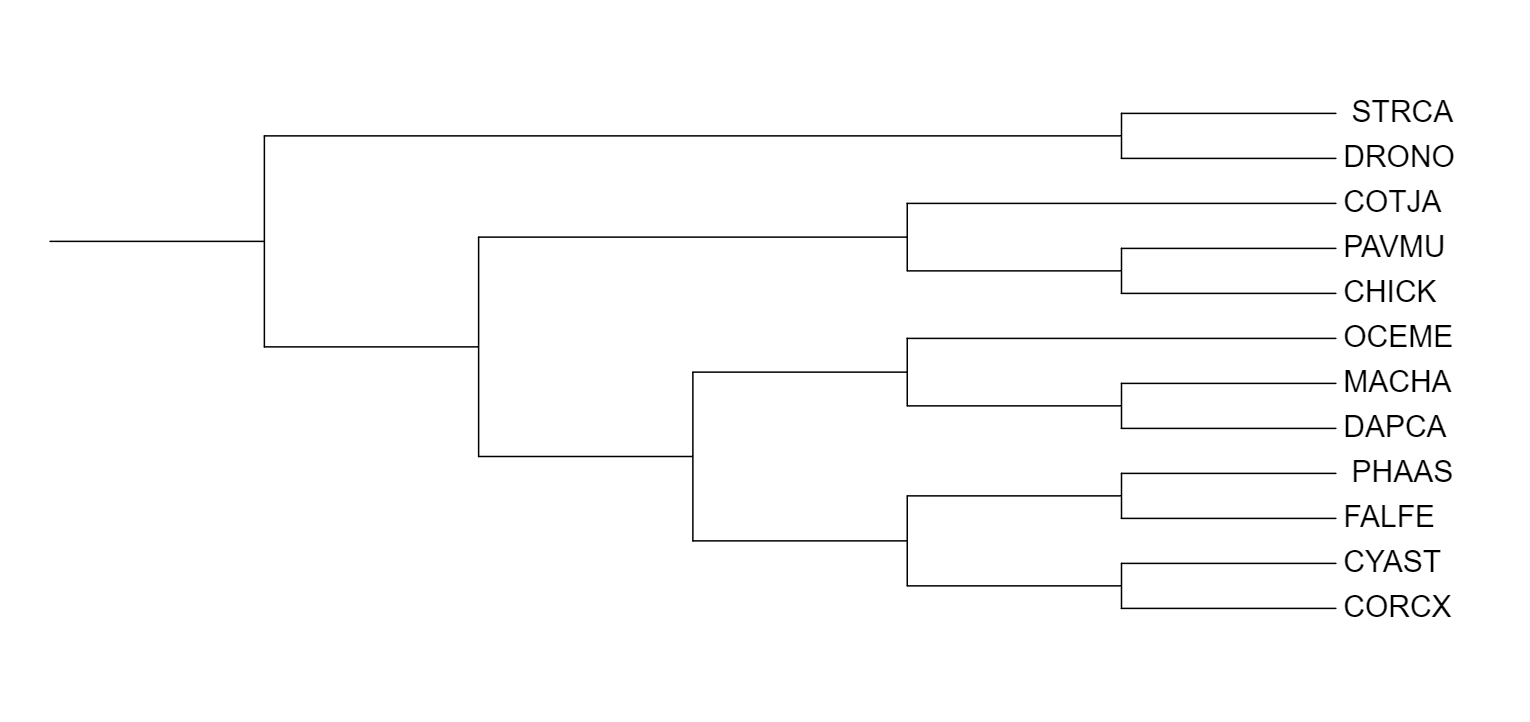
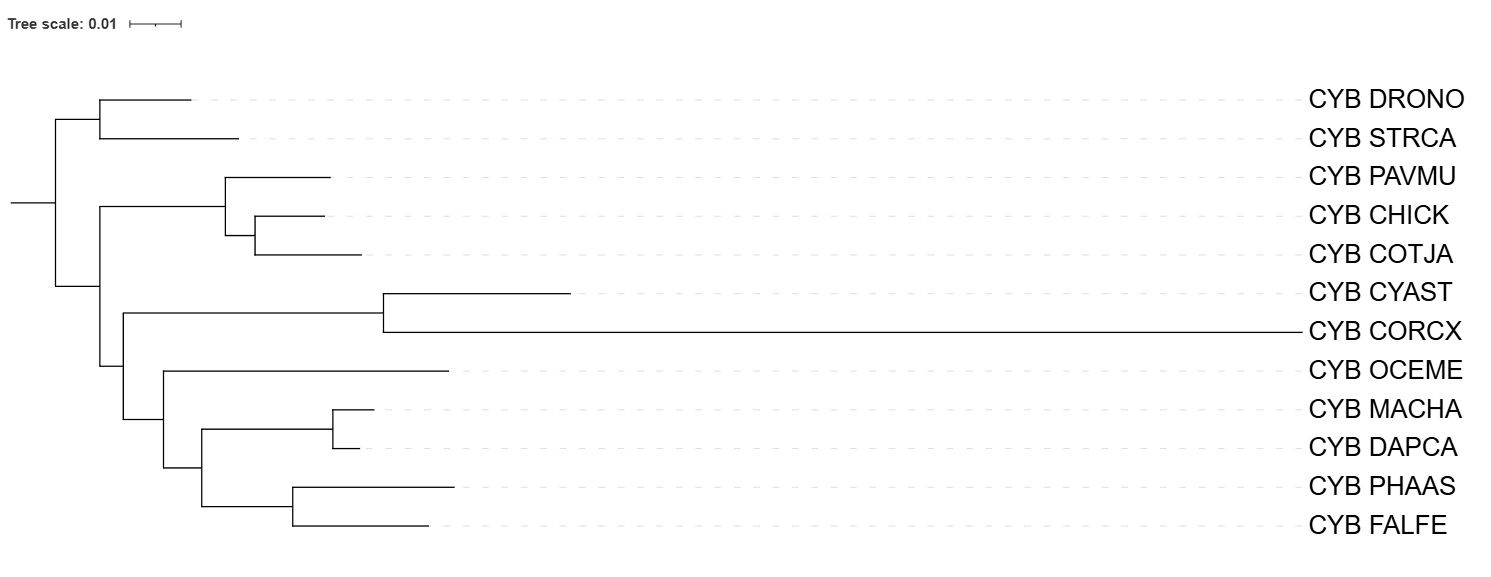
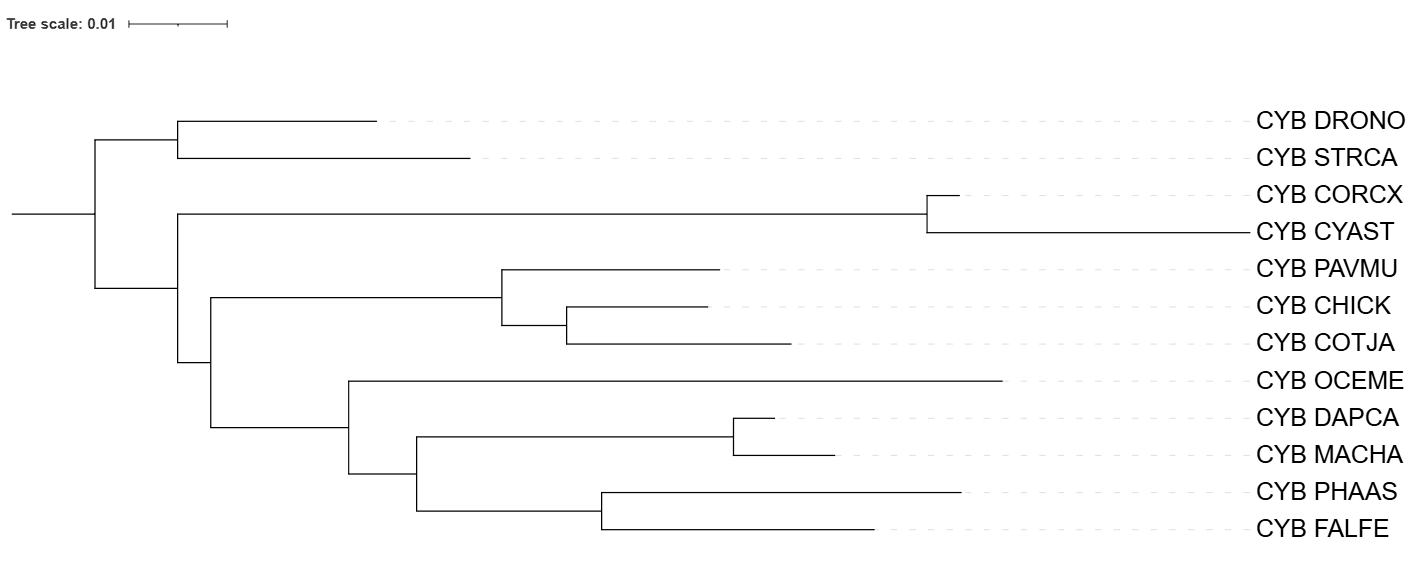
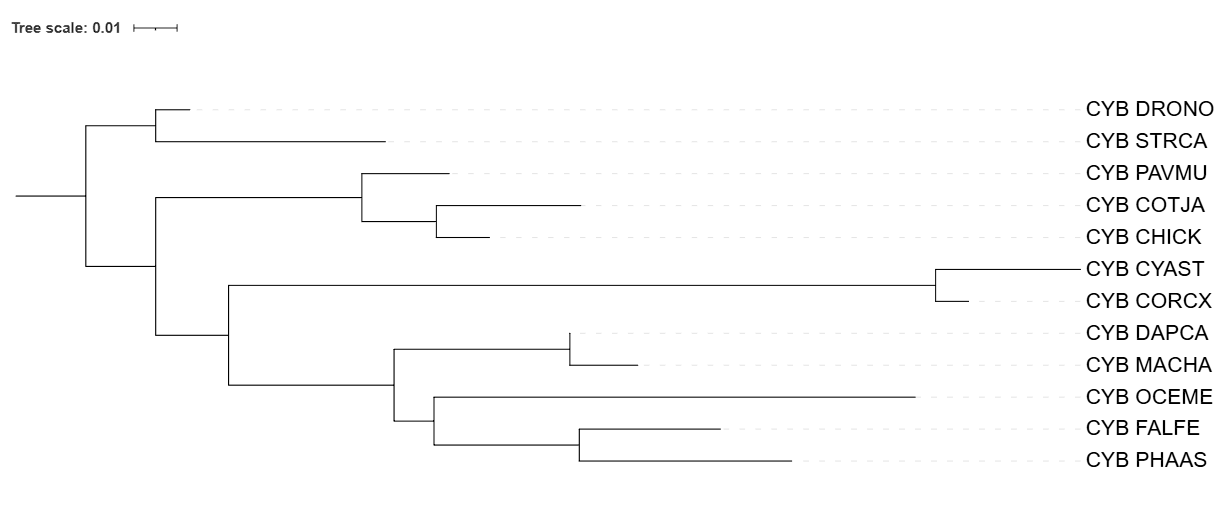
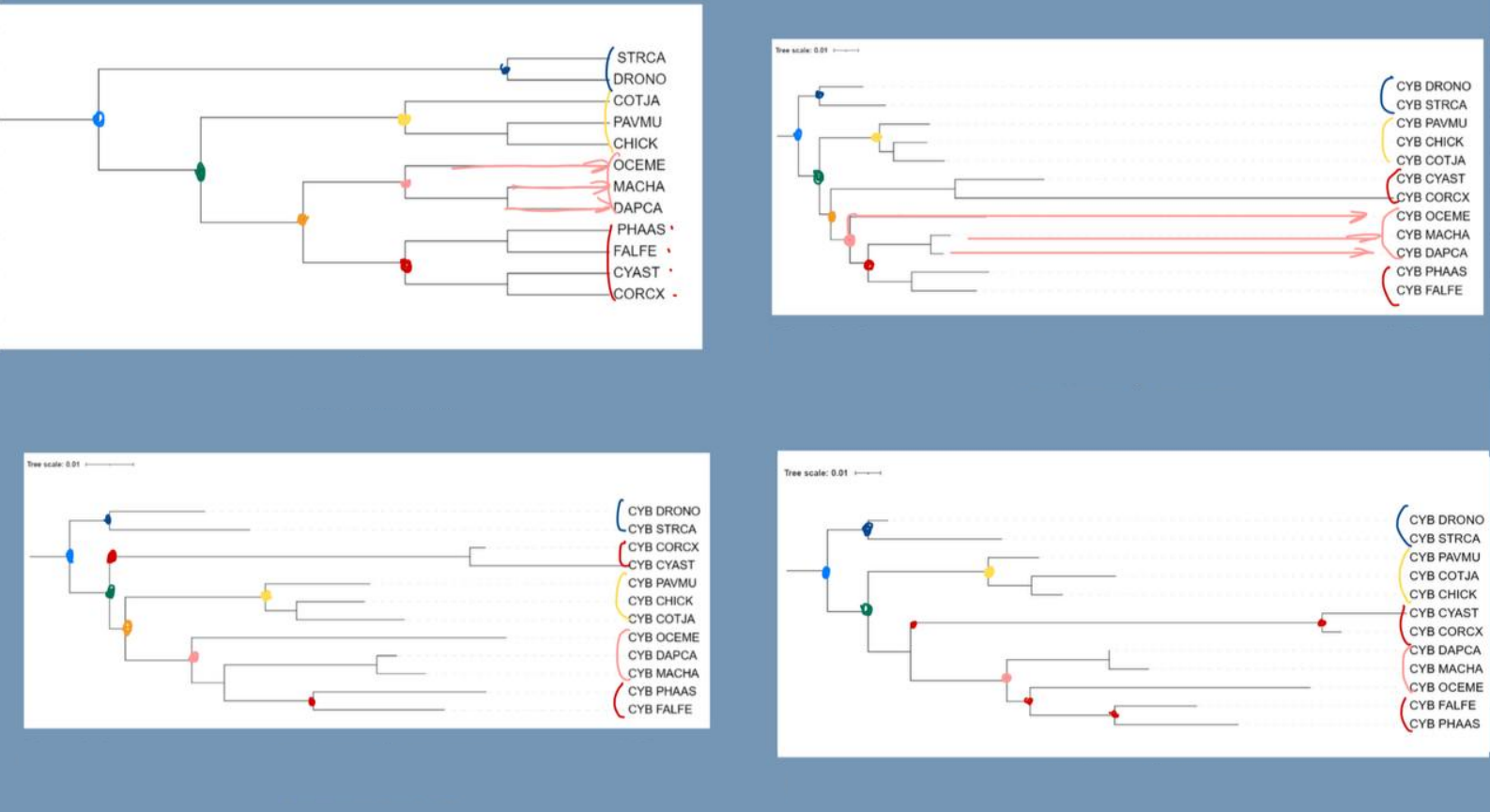
На рис. 1. изображено филогенетическое дерево, построенное на основе данных NCBI Taxonomy. На рис. 2. - дерево, построенное программой Fastme с моделью p-distance. На рис. 3. - дерево, построенное программой Fastme с моделью MtREV. На рисю 4. - дерево, построенное программой IQtree.
Сравнение деревьев
Теперь сравню полученные программами филогенетические деревья с эталонным из NCBI Taxonomy. В таксономии наблюдаются 4 клады: (STRCA, DRONO), (COTJA, PAVMU, CHICK), (OCEME, MACHA, DAPCA), (PHAAS, FALFE, CYAST, CORCX). Стоит отметить что все программы правильно расположили верхнюю ветвь (STRCA, DRONO). Также на всех рисунках можно заметить, что программы объединили GOTJA и CHICK в сестринские группы, хотя на самом деле по таксономии GOTJA является ветвью к кладе где сестринские PAVMU и CHICK.
По таксономии у MACHA DAPCHA и OCEME один ближайший общий предок, а по дереву, построенному программой fastme с моделью p-distance, предок другой от OCEME. Данная ошибка прослеживается во всех деревьях, построенными программами. На Рис. 3. CORCX, CYAST поменяли ближайшего предка на более дальнего чем в таксономии. На Рис. 4,2 та же история повторяется. (OCEME, MACHA, DAPCA) стала предком для (PHAAS, FALFE).