- Cyanocitta stelleri (CYAST) -> Aphelocoma coerulescens(APHCE)
- Falco femoralis (FALFE) -> Falco peregrinus (FALPE)
- Oceanodroma melania (OCEME) -> Oceanodroma tethys (OCETE)
- Macronectes halli (MACHA) -> Macronectes giganteus (MACGA)
Практикум 3. Реконструкция дерева по нуклеотидным последовательностям. Бутстреп
1. Реконструкция дерева по нуклеотидным последовательностям
При поиске последовательностей митохондриальной рибосомальной рнк возникли проблемы. Не для всех животных существуют необходимые записи. По этой причине я решила заменить их на близкородственные, для которых нужные последовательности имеются. Ниже представлен список произведенных замен:
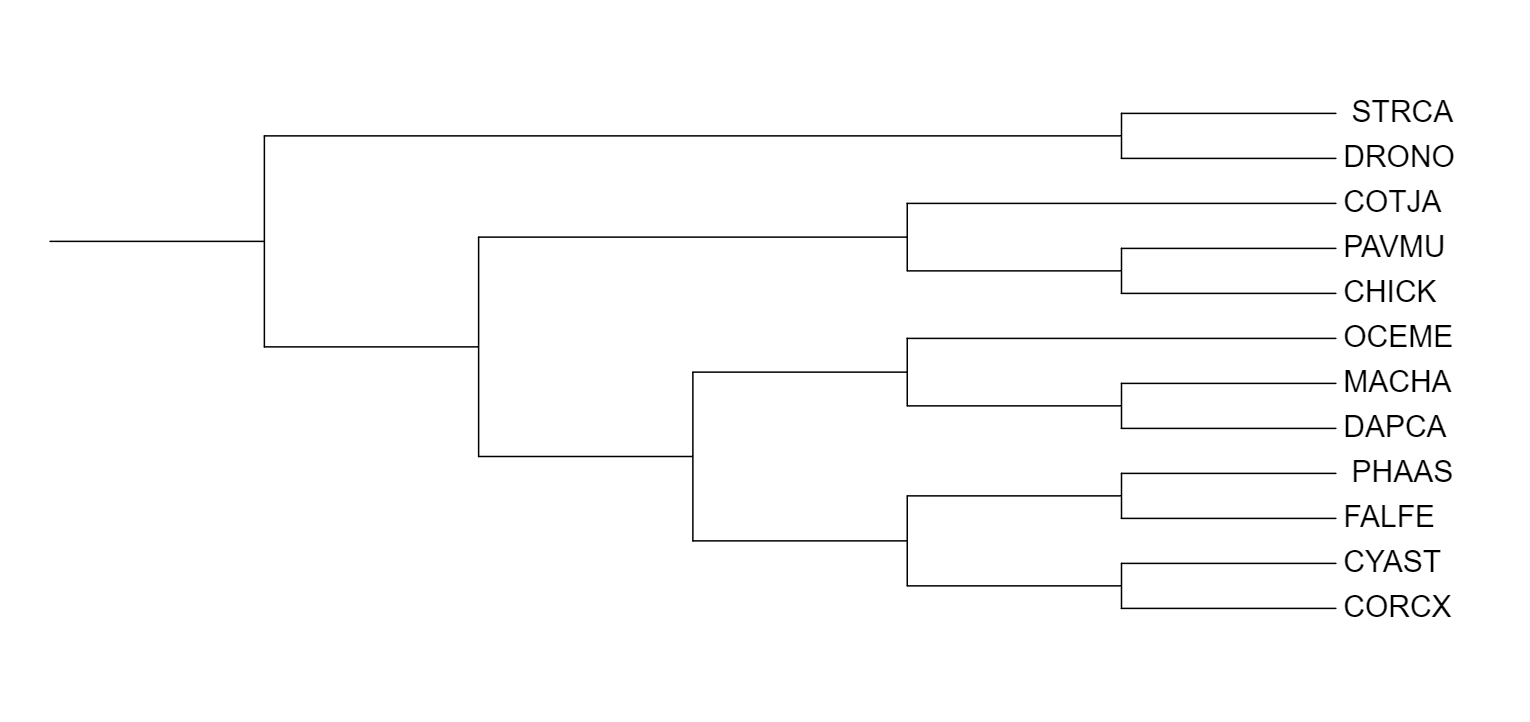
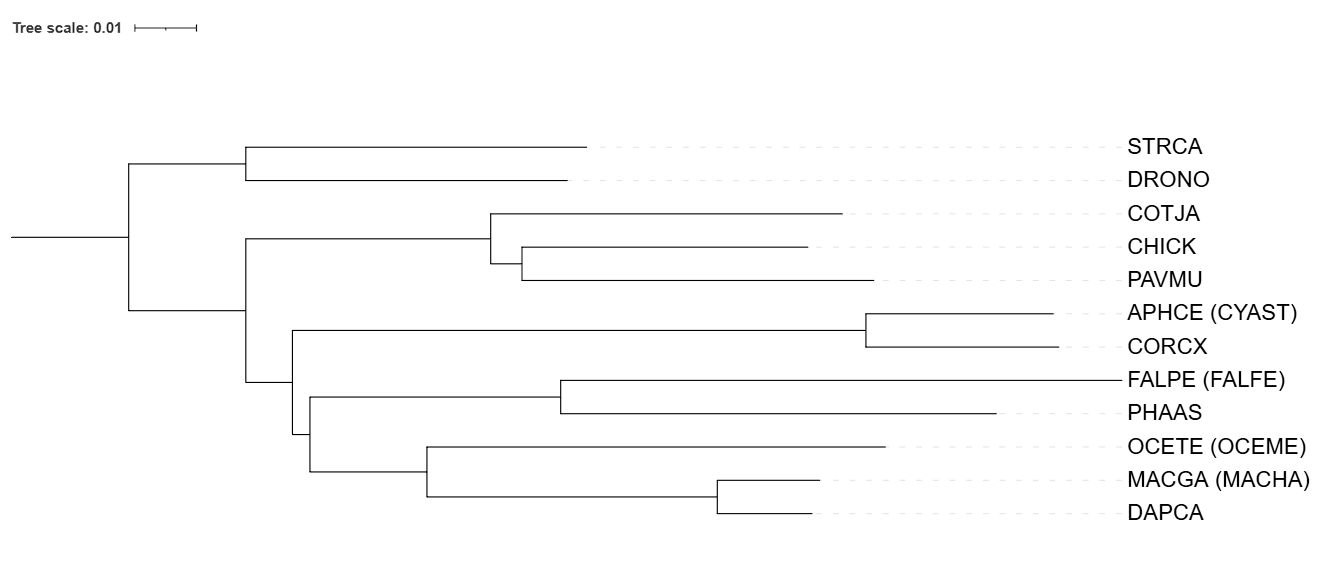
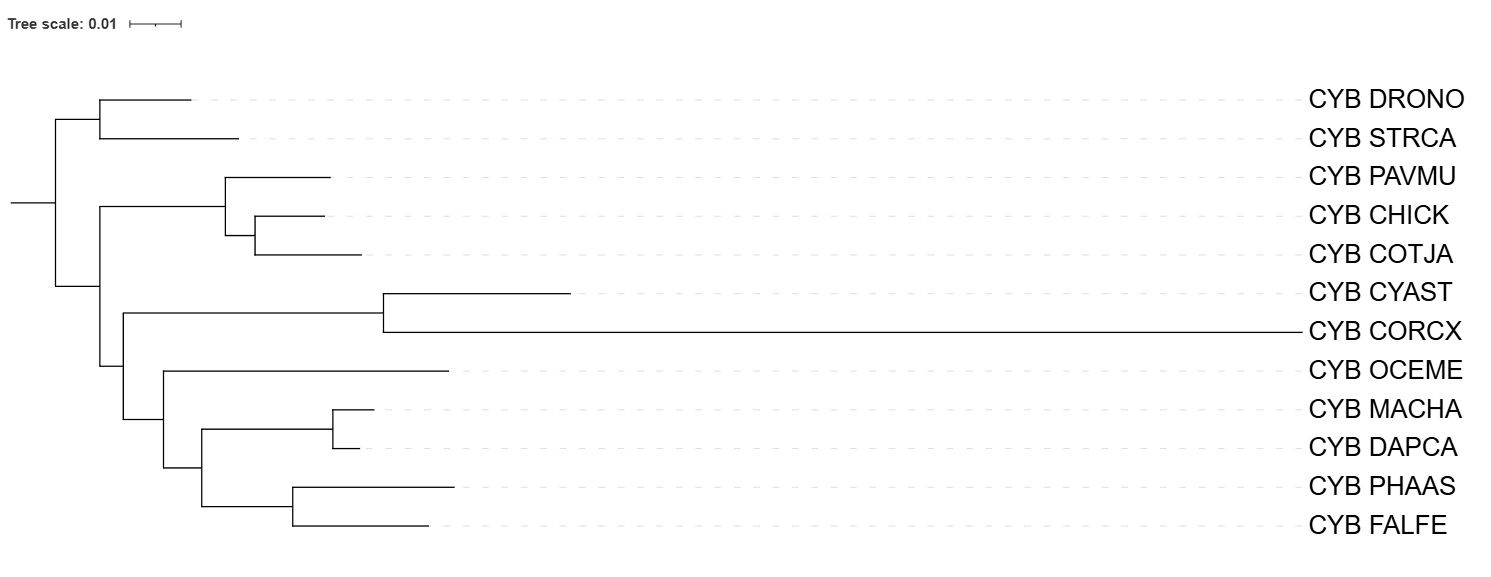
Сравню Рис. 1 и Рис. 2. В целом деревья очень схожи, но есть одно отличие. На дереве, построенным по выравниванию 12S rRNA, ветвь (APHCE, CORCX) отходит раньше, чем ветвь (OCETE, (MACGA, DAPCA)).
Учитывая тот факт, что отличий я нашла всего в одном числе, можно сказать, что дерево, построенное на основе последовательностей 12s rRNA оказалось ближе к дереву, построенному по NCBI по сравнению с деревом, построенным на основе последовательностей цитохрома b.
2. Укоренение во внешнюю группу
В качестве внешней группы я выбрала организм расположенный далеко относительно всех описываемых организмов. Все выбранные организмы относятся к классу Птицы, поэтому внешней группой стал представитель класса Крокодилы - Caiman crocodilus (CAICR) - Крокодиловый кайман. После реконструкции дерево было укоренено в ветвь, отделяющую каймана от всех остальных и получилось верно укорененное дерево, относительно исследуемых видов.
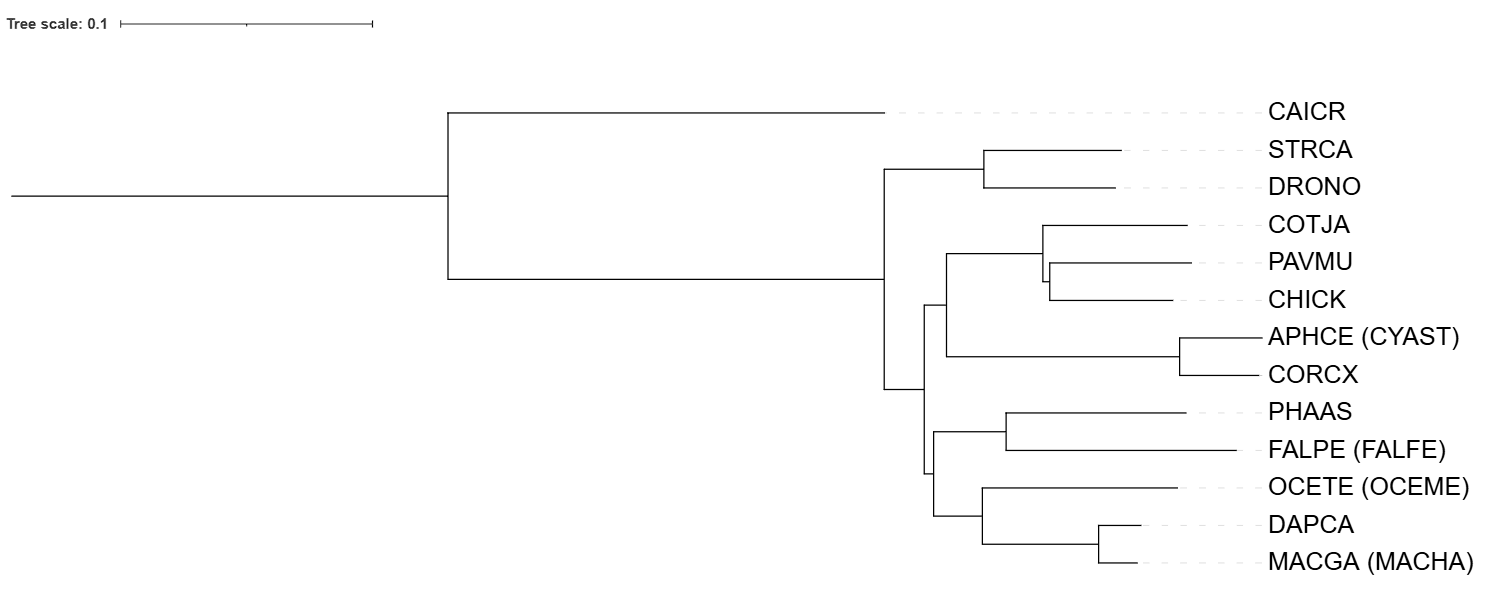
Внешняя группа правильно расположилась на дереве. Само дерево схоже с деревом, построенным по NCBI.
3. Бутстреп
Для оценки достоверности построенных ветвей филогенетического дерева используется метод бутстрепа. Этот метод показывает, насколько устойчива каждая ветвь: чем чаще конкретная ветвь появляется в репликах (повторных выборках), тем выше её поддержка. В данном случае я использовала 100 реплик бутстрепа, что означает, что максимально возможная поддержка для ветви составляет 100. Чем ближе значение поддержки к 100, тем более надёжной считается соответствующая ветвь.
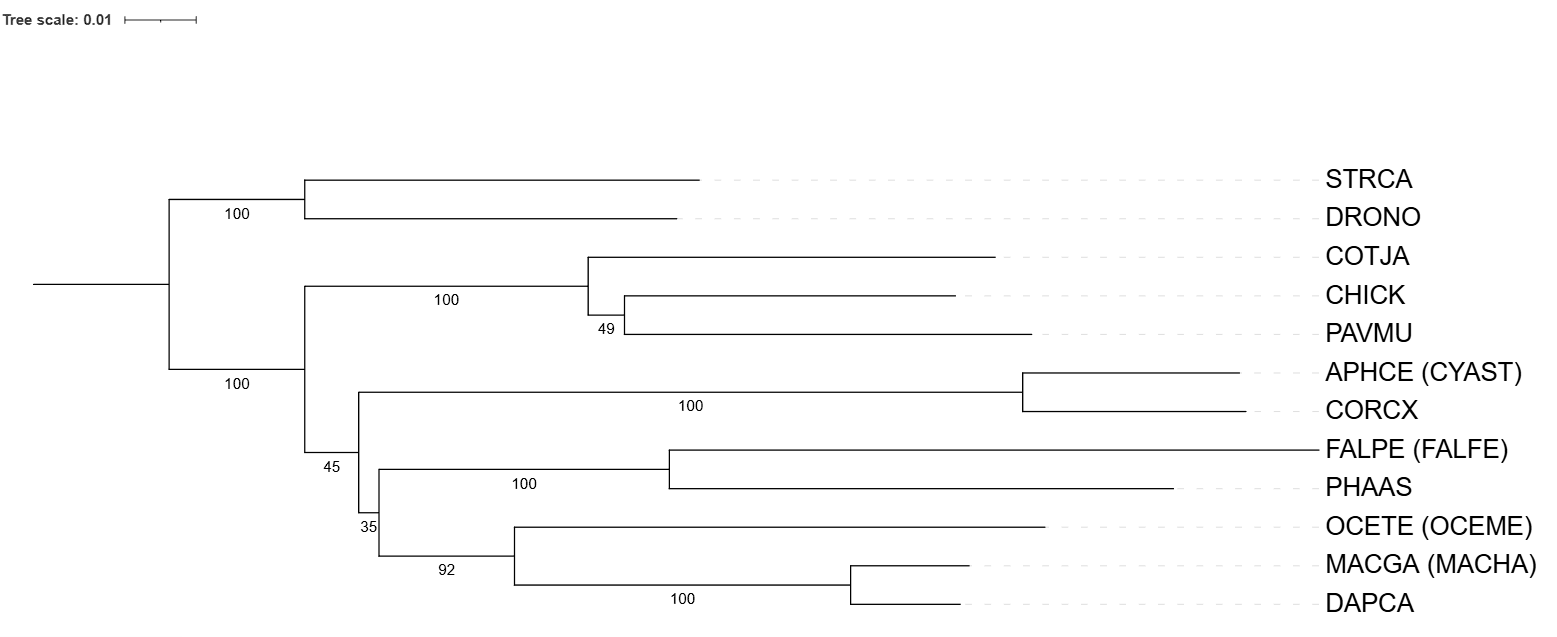
После настройки инструмента itol стали видны числа, которые показывают поддержку каждых ветвей. Ранее я говорила, что ветвь (APHCE, CORCX) отходит раньше, чем ветвь (OCETE, (MACGA, DAPCA)). И если взглянуть на числа поддержки в этих местах (45 и 35), то становится очевидным тот факт, что неправильно реконструированные ветви имеют меньшую поддержку.