- sp|P0A910|OMPA_ECOLI Beta sheet 28 35
- sp|P0A910|OMPA_ECOLI Beta sheet 58 65
- sp|P0A910|OMPA_ECOLI Beta sheet 70 76
- sp|P0A910|OMPA_ECOLI Beta sheet 99 106
- sp|P0A910|OMPA_ECOLI Beta sheet 113 120
- sp|P0A910|OMPA_ECOLI Beta sheet 143 150
- sp|P0A910|OMPA_ECOLI Beta sheet 156 163
- sp|P0A910|OMPA_ECOLI Beta sheet 183 190
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 12 16
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 38 45
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 50 56
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 79 86
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 93 100
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 123 130
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 136 143
- sp|P0A910|OMPA_ECOLI/21-346 Beta sheet 163 170
- 6B87_1|Chains TMhelix 18 35
- 6B87_1|Chains TMhelix 75 90
Практикум 7. Трансмембранные белки
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения данного задания я выбрала белок Outer membrane protein A. Ниже представлена информация о белке:
| Название белка (русский) | Белок внешней мембраны A |
| Название белка (английский) | Outer membrane protein A (OMPA) |
| Идентификатор PDB | 1bxw |
| Идентификатор UniProt | OMPA_ECOLI |
| Организм | Escherichia coli (strain K12) |
| Мембрана | Внешняя мембрана грамотрицательных бактерий |
| Функция белка | Поддерживает целостность наружной мембраны и положение пептидогликана в периплазме. Участвует в формировании клеточной формы и устойчивости к стрессам. Слабо проницаемый порин: пропускает малые молекулы (например, L-арабинозу) в редкой "открытой" конформации (3% молекул). Образует ионные каналы с двумя состояниями проводимости (50-80 pS и 260-320 pS). |
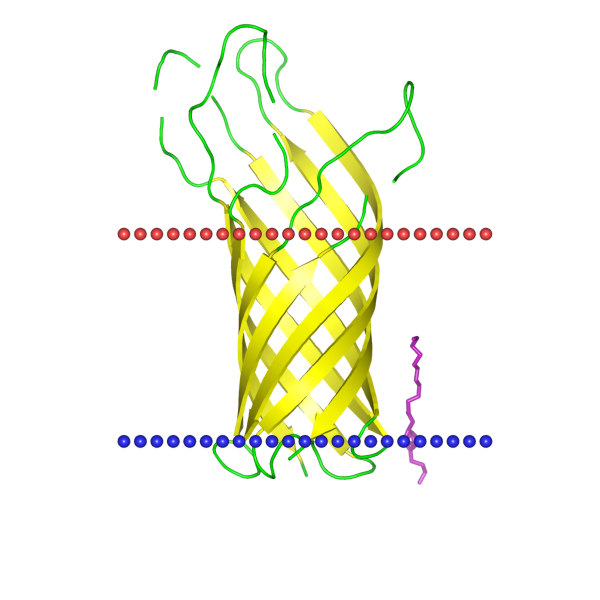
Координаты трансмембранных участков: 1(7 - 15), 2(35 - 44), 3(51 - 58), 4(76 - 85), 5(93 - 101), 6(120 - 130), 7(137 - 145), 8(160 - 169)
Для проведения сравнения я взяла fasta последовательность моего трансмембранного белка с UniProt. Ссылка на последовательность
Файл с результатами предсказания.
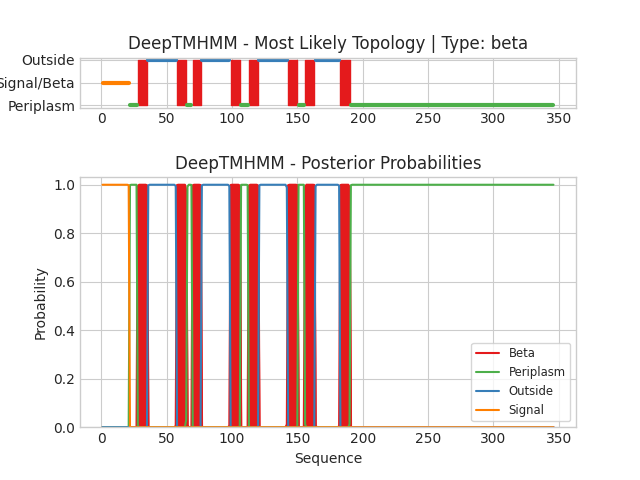
Предсказанные программой DeepTMHMM координаты трансмембранных бета-листов:
В таблице представлены результаты предсказания трансмембранных доменов, где оба метода обнаруживают по 8 участков. Однако никакой из них хорошо не перекрывается, среднее отклонение ~20АК, что критично для участков средней длины 10–12 АК. При этом исходная последовательность из UniProt содержит 346 АК, тогда как в ОРМ и PDB-структуре их всего 172.
Помимо этого наблюдается смещение координат сегментов между предсказаниями (в среднем на 20 аминокислот), так как последовательность с Uniprot включает N-концевую сигнальную последовательность (примерно 20 АК).
Из-за этого мною было принято решение сделать выравнивание двух последовательностей белка из UniProt и PDB.
После анализа выравнивания я решила удалить 20 аминокислот с начала последовательности белка из UniProt как сигнальная последовательность (вырезанная последовательность белка). Далее я запустила снова DeepTMHMM для новой последовательности белка (новая выдача DeepTMHMM).
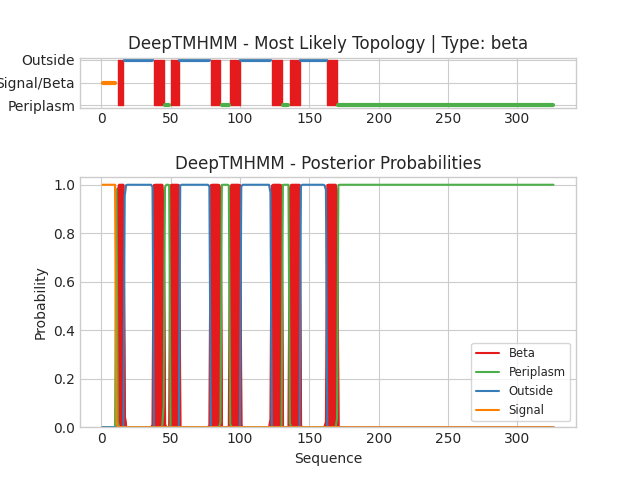
Предсказанные программой DeepTMHMM координаты трансмембранных бета-листов:
Теперь результаты предсказаний совпадают отлично, 8 участков и все перекрываются, границы выходят в среднем на 2-3 АК. Однако все равно программа DeepTMHMM делает косяки (например, первые 10 АК она определяет как сигнальную последовательность, что можно увидеть в текстовой выдаче, хотя истинная сигнальная последовательность была удалена).
Чтобы глубже покапаться в причине того, почему такая разница в длине последовательностей из UniProt и PDB я решила воспользоваться инструментом для поиска и визуализации белковых доменов (ссылка на ресурс)

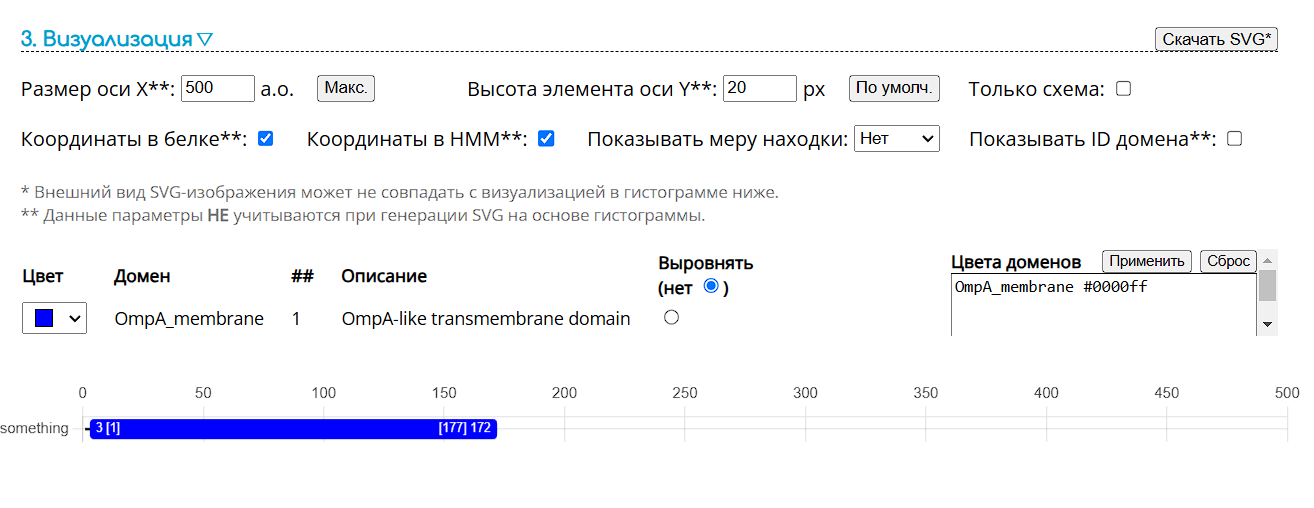
В последовательности из Uniprot вставлен домен OmpA family (отмечен зеленым на рисунке 4). Этим можно объяснить сильные различия в длине последовательностей Uniprot 346 АК и Pdb 172 АК.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для выполнения этого задания мне был выдан белок Protein TMHC2_E. Информация о белке представлена ниже:
| Название белка (русский) | Белок TMHC2_E |
| Название белка (английский) | Protein TMHC2_E |
| Идентификатор PDB | 6b87 |
| Идентификатор UniProt | Нет записи |
| Организм | Нет записи (synthetic construct) |
| Мембрана | Нет записи |
| Функция белка | TMHC2_E — искусственный белок без биологических функций, созданный как модель для изучения трансмембранных α-спиралей. Отсутствие природного аналога и экспериментальных данных о его активности исключает какую-либо функциональную роль |
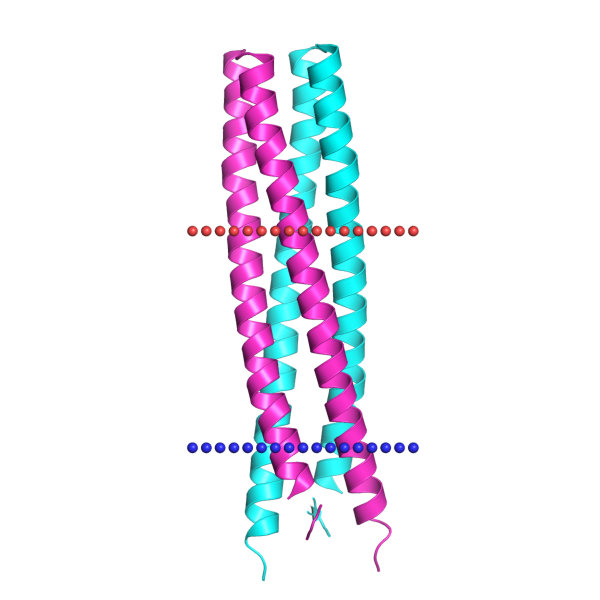
Координаты трансмембранных участков: 1(12 - 34), 2(70 - 92)
Для проведения сравнения я взяла fasta последовательность моего трансмембранного белка с RCSB PDB. Ссылка на последовательность
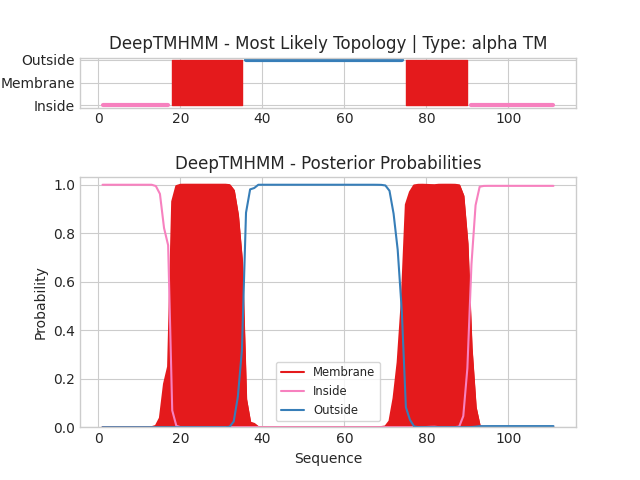
Файл с результатами предсказания.
Предсказанные программой DeepTMHMM координаты трансмембранных бета-листов:
Как видно предсказания совпали успешно: в обоих случаях по два участка, причем везде наблюдается перекрытие. Границы участков различаются несущественно - в пределах 5-7 АК при средней длине участка около 22 АК. Следует отметить, что DeepTMHMM не обнаружил наличия сигнальных пептидов в исследуемых последовательностях.