Сборка De novo
0. Скачивание архива
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/008/SRR4240378/SRR4240378.fastq.gz
Создание объединенного файла с адаптерами было сделано с помощью команды
cat /mnt/scratch/NGS/adapters/*.fa > adapters.fa
1. Подготовка чтений программой trimmomatic
Удаление адаптеров
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240378.fastq.gz trimadapt_SRR4240378.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7 2> trimadapt.err
Отчет об изначальной последовательности и воследовательности без адаптеров
fastqc SRR4240378.fastq.gz
fastqc SRR4240378.fastq.gz
Удаление чтений, длина которых меньше 32 нуклеотидов, а качество ниже 20
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 trimadapt_SRR4240378.fastq.gz trim_SRR4240378.fastq.gz TRAILING:20 MINLEN:32 2> trim.err
Качество полученных данных было проверено с помощью программы
fastqc trim_SRR4240378.fastq.gz
2.Создание k-меров
Разбиение данных на к-меры было сделано с помощью следующей программы:
velveth velvet/ 31 -fastq.gz -short trim_SRR4240378.fastq.gz
3.Сборка контигов на основе полученных k-меров
velvetg velvet/
Анализ полученных данных:
N50 = 7028
| ID | Длина | Покрытие |
|---|---|---|
| 8 | 36746 | 20.017199 |
| 57 | 19371 | 20.546642 |
| 15 | 16745 | 20.901762 |
4. Анализ полученных данных
| # | Score | E-value | Identities | Выровненный участок контига | Выровненный участок хромосомы | Gaps |
|---|---|---|---|---|---|---|
| 1 | 3949 bits | 0.0 | 6516/8617(76%) | 8431...16876 | 508806...500370 | 351/8617(4%) |
| 2 | 3932 bits | 0.0 | 4897/6234(79%) | 562...6740 | 516539..510438 | 187/6234(2%) |
| 3 | 2278 bits | 0.0 | 4621/6238(74%) | 29537...35594 | 488106...481997 | 308/6238(4%) |
| 4 | 1921 bits | 0.0 | 3255/4324(75%) | 16992...21270 | 500325...496111 | 154/4324(3%) |
| 5 | 1020 bits | 0.0 | 1109/1384(80%) | 22688...24064 | 494864...493487 | 13/1384(0%) |
| 6 | 573 bits | 5e-163 | 564/686(82%) | 36068...36747 | 481545...480874 | 20/686(2%) |
| 7 | 150 bits | 1e-35 | 108/120(90%) | 22436...22554 | 495148 ...495033 | 5/120(4%) |
| # | Score | E-value | Identities | Выровненный участок контига | Выровненный участок хромосомы | Gaps |
|---|---|---|---|---|---|---|
| 1 | 3253 bits | 0.0 | 7212/9822(73%) | 9754...19341 | 582686...573092 | 461/9822(4%) |
| 2 | 1279 bits | 0.0 | 2100/2777(76%) | 5348...8066 | 587055...584329 | 108/2777(3%) |

| # | Score | E-value | Identities | Выровненный участок контига | Выровненный участок хромосомы | Gaps |
|---|---|---|---|---|---|---|
| 1 | 4423 bits | 0.0 | 5863/7536(78%) | 6309...13708 | 151796...144368 | 243/7536(3%) |
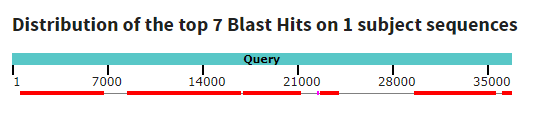
Чтобы лучше понять, как накладываются контиги на банковский геном, посмотрим на карты локального сходства:

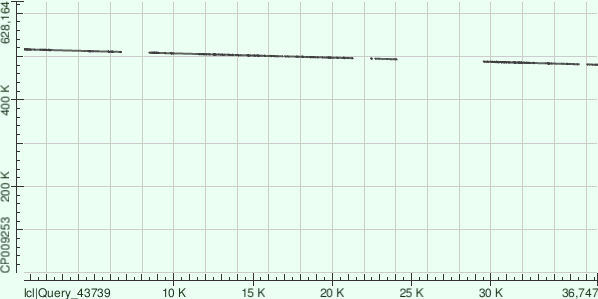
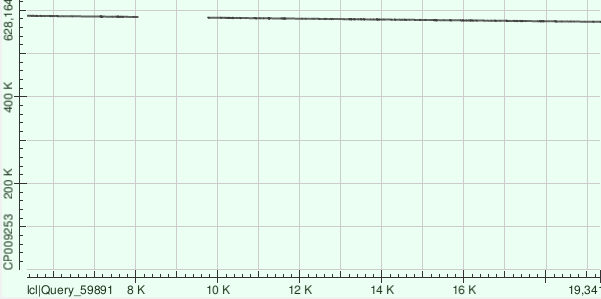
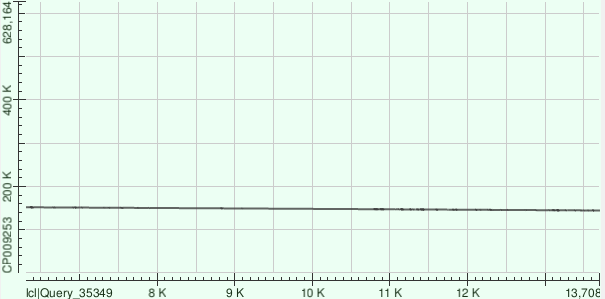
Контиги легли довольно неплохо. Прерывистость во всех картах можно объяснить вариабельными участками.