Практикум 8
Задание 1. Консервативный мотив в выравнивании последовательностей гомологичных белков
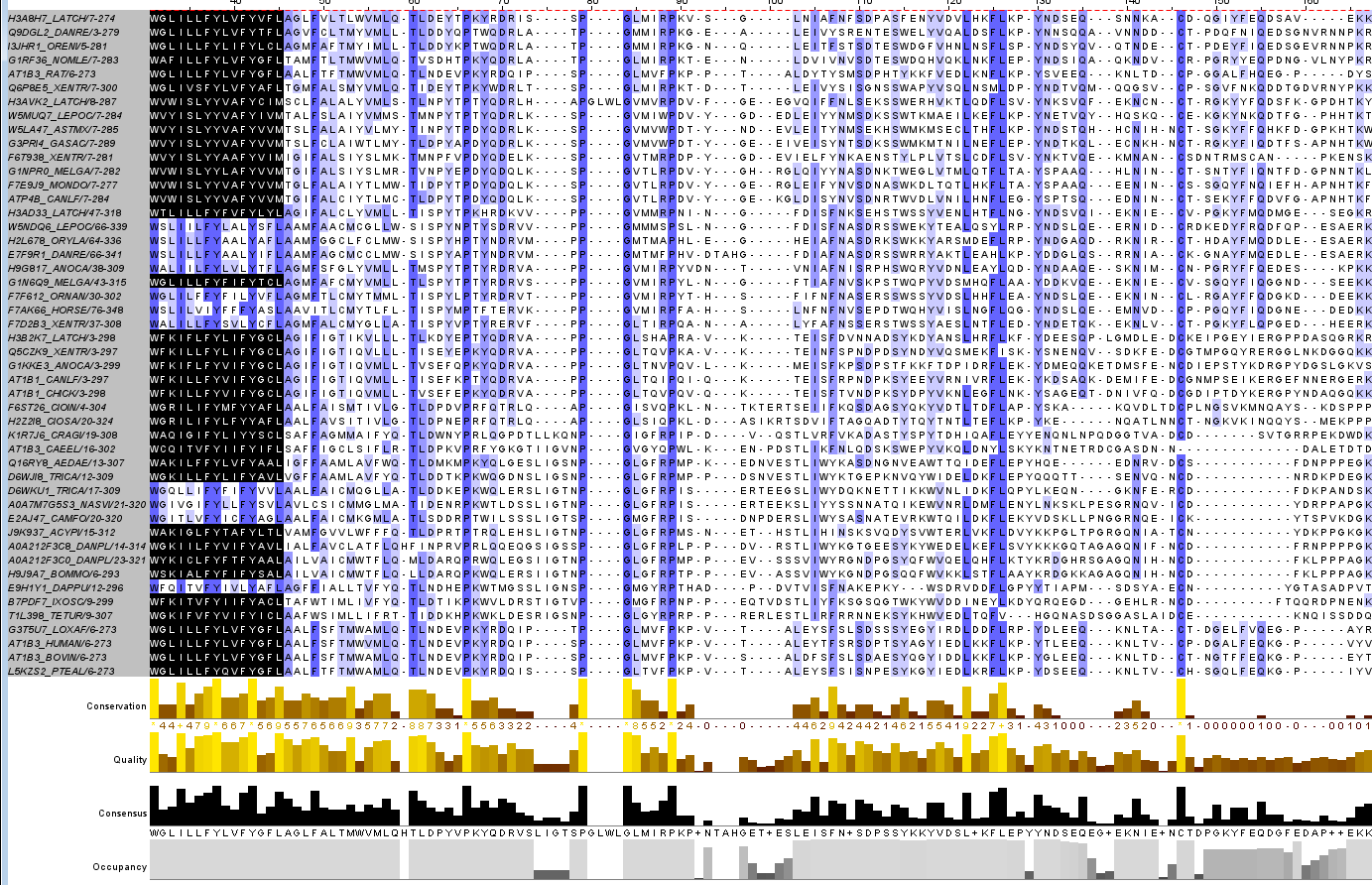
Для выполнения задания было выбрано семейство Na_K-ATPase (PF00287)
seed: 59
Паттерн выбранного мотива: W-X-X-I-X-X-[FY]-Y-X-X-[FY]-Y-X-X-[LM]
Данный мотив содержится в 48 последовательностях, поэтому можно говорить, что его информационное содержание достаточно высокое.
Далее с помощью сервиса MyHints был выполнен поиск данного мотива в базе данных Swiss-prot. Получено 44 находки, все они принадлежат бэта-субъединицам АТФаз. Таким образом, можно сделать вывод, что мотив является характерным для выбранного семейства.
Поиск мотива, специфичного для клады
С использованием метода NJ было построено филогенетическое дерево и выделена клада c 13 последовательностями.
В качестве консервативного был выбран мотив: PC[IV]..[KS]LN[KR][IL]..W.P
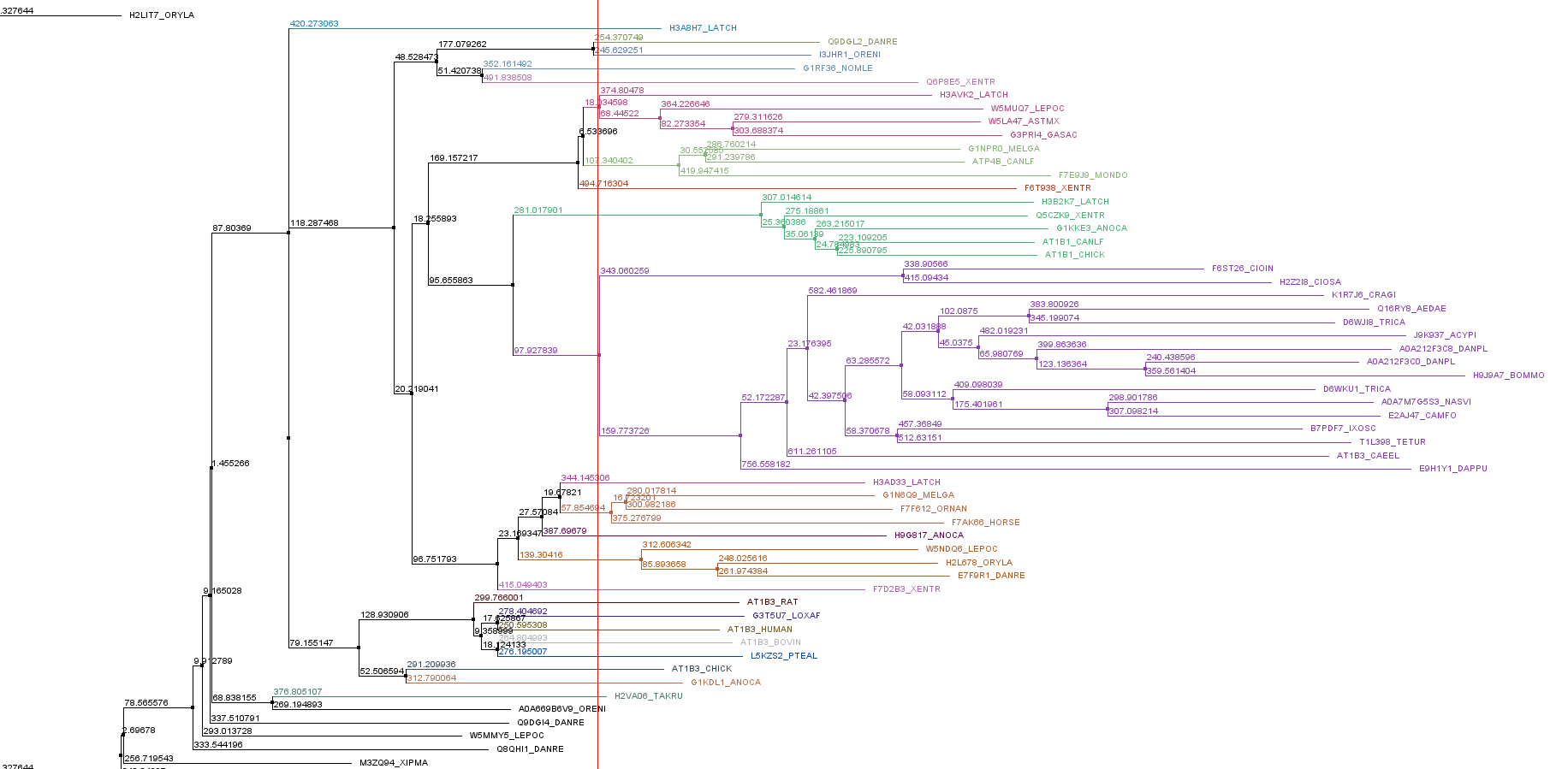
Поиск мотива был выполнен внутри клады, показано, что он встречается во всех 13 последовательностях. Также поиск среди всех последовательностей показал, что данный мотив встречаетмя в 13 последовательностях из общего числа (т.е. не встречается больше нигде в выравнивании). Это говорит о том, что выбранный мотив хорошо характеризует данную кладу.
PSI-BLAST
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 162 | Q2P036.1 | \(\begin{equation}0,004\end{equation}\) | Q5F5V4.1 | 0.005 |
| 2 | 188 | O25693.2 | \(\begin{equation}2e-09\end{equation}\) | - | - |
| 3 | 188 | Q9ZM51.1 | \(\begin{equation}1e-11\end{equation}\) | A7H8E6.1 | 0.012 |