Практикум 9
Домены и профили
Для данного задания был взят домен Peptide-N-glycosidase F, N terminal (ссылка на страницу в Pfam).

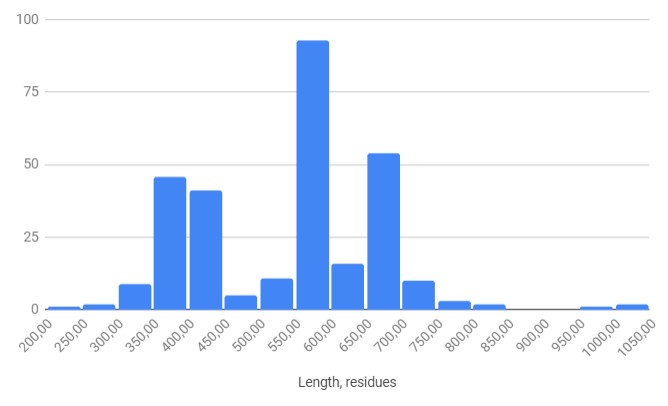
Затем была составлена таблица с информацией о всех белках с выбранной доменной архитектурой из Uniprot. Поиск проводился по запросу: database:(type:pfam pf09112) database:(type:pfam pf09113). Затем была составлена диаграмма с длинами этих белков (рис. 2). Значения для диаграммы были получены из исходного файла с помощью команды awk -F";" '{for(i=1;i<=NF;i++){if ($i ~ /residues/){print $i}}}' table.txt > outfile.txt. Как мы видим, характерные длины белков лежат в интервале от 550 до 600 нуклеотидов.
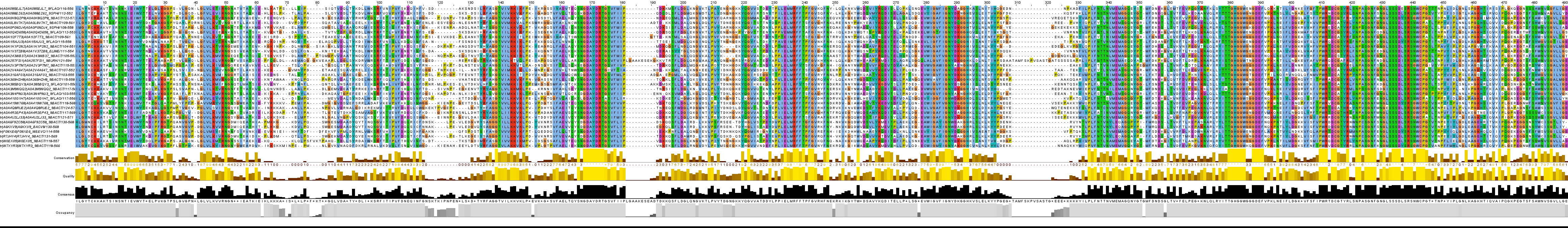
Были получены 40 файлов с последовательностями белков выборки. Затем с помощью команды cat все последовательности были записаны в один общий файл. Затем этот файл был загружен в Jailview и все последовательности были выравнены с помощью Mafft with Deffaults. Была также проведена небольшая ревизия, чтобы удалить участки перед первым консервативным блоком и после последнего, а также удалить последовательности, очевидно выбивающиеся из выборки. Окончательный вариант выравнивания представлен на рисунке 3. Его также можно скачать в fasta-формате.
По полученному выравниванию с помощью команды hmm2build -g profile.hmm alignment.fa был создан hmm-профиль. Затем этот профиль был откалиброван с помощью следующей команды: hmm2calibrate profile.hmm. Конечный варинат профиля доступен по ссылке.
Для начала из Uniprot были скачаны в один файл все найденные последовательности с выбранным доменом. Затем с помощью команды hmm2search --cpu=1 -E 0.1 profile.hmm seqs.fa > hmm.txt была проведена проверка hmm-профиля, результаты которой приведены в файле hmm.txt. По сравнению с данными, полученными до этого, количество находок уменьшилось ровно на 300 (1076). В эти находки входят только те, которые были найдены ранее. На рисунке 4 можно видеть распределение весов получившихся находок.
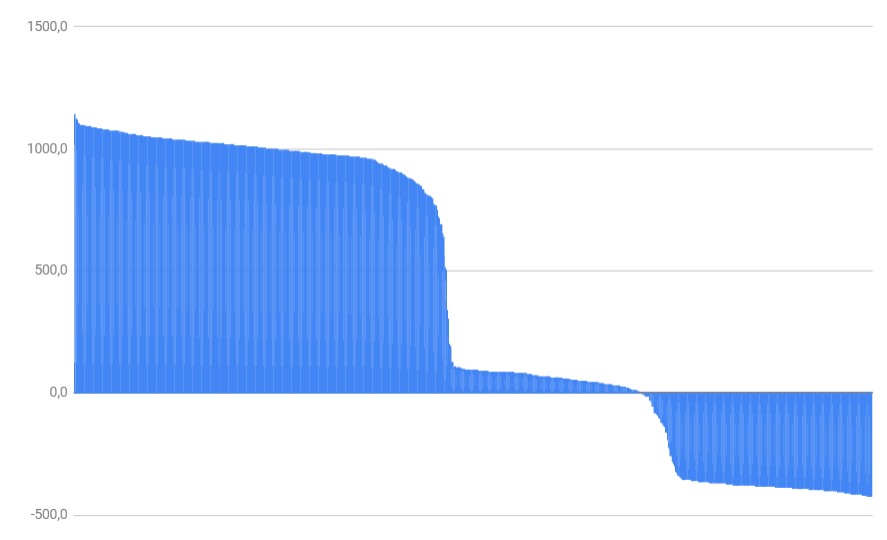
Видно, что в точке в районе 770 есть резкий скачок вниз. Возьму точку со значением 770,4 в качестве порогового значения. Затем с помощью посчитанных прежде значений 1-specifiry и Sencivity для этого значения была построена Таблица 2х2.
| uniprot+ | uniprot- | |
|---|---|---|
| hmm+ | 486 | 0 |
| hmm- | 590 | 0 |
ROC-кривая не получилсь, поскольку деальная специфичность равна единице.