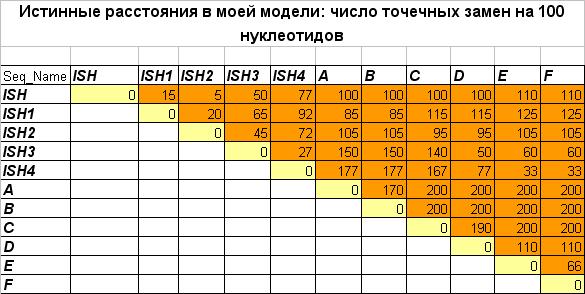
Обозначения последовательностей:
ISH — исходная последовательность гена;
ISH1...ISH4 — последовательности, находящиеся в узлах 1, 2, 3, 4
соответственно;
A...F — последовательности в соответствующих листьях.

Далее с помощью программы emma было построено множественное выравнивание. У
исполнения программы была одна ма-аленькая особенность: были использованы
параметры -gapopen и -gapextend со значениями 100.0 и 10.0 соответственно.
Эти значения практически запрещают гэпы в выравнивании, а они нам НЕ НУЖНЫ,
потому что эволюционная модель НЕ ПРЕДУСМАТРИВАЕТ ни вставок, ни делеций.
Множественное выравнивание использовалось для построений двух матриц: попарных
различий (D) и попарных расстояний (вычисленных по формуле Джукса-Кантора).
Матрицы были построены с помощью программы distmat пакета EMBOSS. Правда, для
первой матрицы был использован пункт меню 0 (расстояние как среднее число
несовпадающих нуклеотидов), а для второй — пункт 1 (вычисление расстояний по
методу Джукса-Кантора). Матрицы приведены ниже.
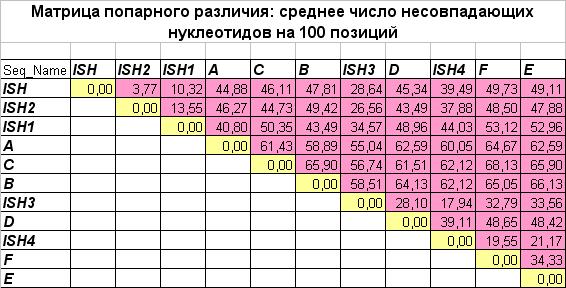
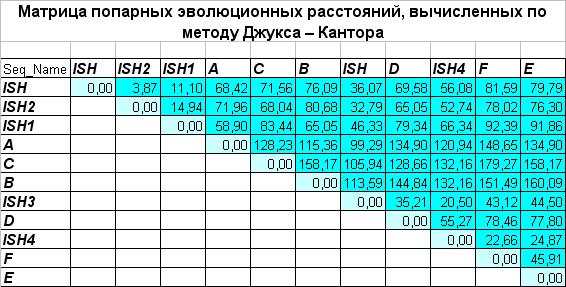
Стоит заметить, что порядок последовательностей в этих двух матрицах немного
отличается от такового в исходной матрице. Видимо, это связано с особенностями
работы программы distmat...
Следующим шагом стало преобразование всех трёх матриц в таблицы вида "Имя
пары" - "Расстояние" и объединение получившихся таблиц в одну. На основе этой
общей таблицы (отсортированной по убыванию "истинных" расстояний) был построен
график, демонстрирующий зависимость двух оценок расстояния от величины
"истинного" расстояния.
 |
На получившемся графике видно, что два способа оценки расстояния, указанных в
начале, выдают результаты меньше "истинного" расстояния. Правда, модель Джукса-
Кантора лучше считает истинное расстояние, чем обычный подсчёт числа
несовпадающих нуклеотидов. При значениях расстояния, близких к 0, результаты примерно одинаковы (то есть получившиеся оценки расстояний близки к "истинным"). Далее идёт расхождение в сторону уменьшения расстояний. Любопытно, что разница между результатами применения двух способов, названных в начале, выше, чем разница между "истинными" расстояниями и результатами применения модели Джукса-Кантора. Более того, при больших значениях "истинного" расстояния линия, показывающая величину несовпадений (т.е. попарных различий), стремится к горизонтальному положению. |