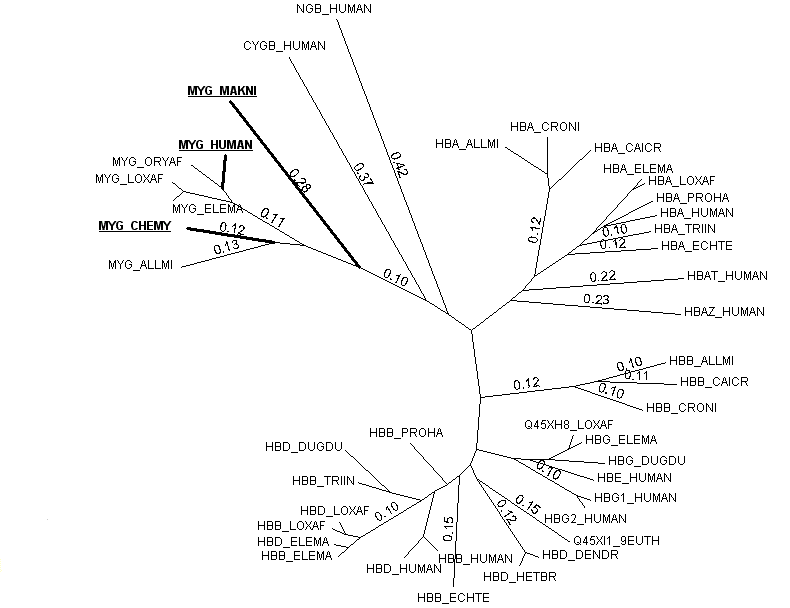

На дереве показаны названия белков, а также длины основных ветвей. 3 белка
внешней группы (MYG_HUMAN, MYG_CHEMY и MYG_MAKNI) выделены жирным шрифтом и
подчёркнуты, а ветви, идущие к ним, имеют бОльшую толщину.
Сделано это было следующим образом: опция "Properties" --> "Text" (для изменения
подписей) либо "Font" (для изменения шрифта).
Euteleostomi + Sarcopterygii + + Amniota + + + Sauropsida + + + + Sauria + + + + + Crocodylidae + + + + + + Alligatorinae + + + + + + + Alligator + + + + + + + + Alligator mississippiensis + + + + + + + Caiman + + + + + + + + Caiman crocodilus + + + + + + Crocodylinae + + + + + + + Crocodylus niloticus + + + + Testudines + + + + + Chelonia mydas + + + Mammalia + + + + Eutheria + + + + + Afrotheria + + + + + + Hyracoidea + + + + + + + Procaviidae + + + + + + + + Dendrohyrax + + + + + + + + + Dendrohyrax dorsalis + + + + + + + + Heterohyrax + + + + + + + + + Heterohyrax brucei + + + + + + + + Procavia + + + + + + + + + Procavia capensis habessinica + + + + + + Sirenia + + + + + + + Dugongidae + + + + + + + + Dugong dugon + + + + + + + Trichechidae + + + + + + + + Trichechus inunguis + + + + + + Tenrecidae + + + + + + + Echinops telfairi + + + + + + Macroscelidea + + + + + + + Elephantulus intufi + + + + + + Proboscidea + + + + + + + Elephantidae + + + + + + + + Elephas + + + + + + + + + Elephas maximus + + + + + + + + Loxodonta + + + + + + + + + Loxodonta africana + + + + + + Tubulidentata + + + + + + + Orycteropus afer + + + + + Euarchontoglires + + + + + + Homo sapiens + Actinopterygii + + Makaira nigricans
Дерево было также сохранено в виде скобочной структуры формата phylip. После
некоторого редактирования данная структура была отображена в GeneMaster.
Ниже можно эту картинку увидеть.
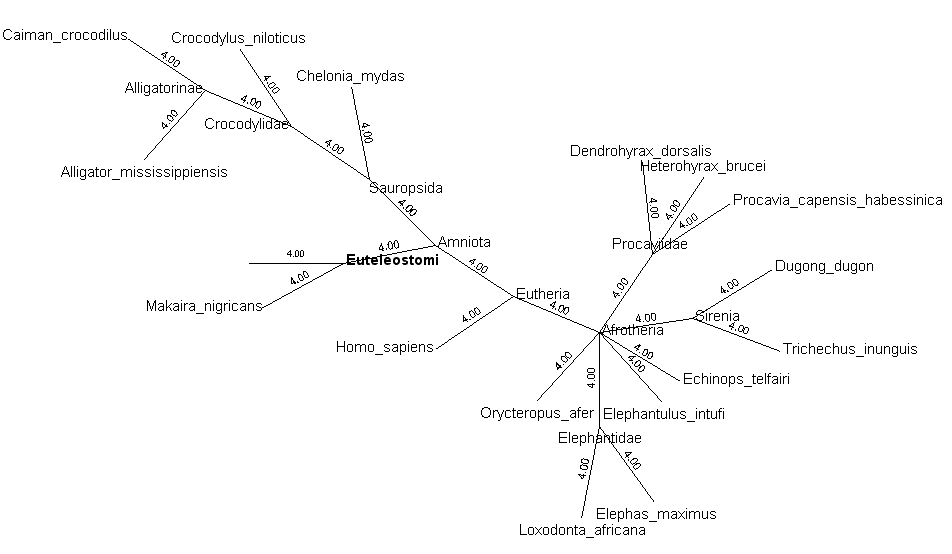
Жирным шрифтом показан родительский таксон — Euteleostomi. От него
отходят три ветви: ветвь Amniota (из таксона Sarcopterygii), ветвь с организмом
Macaria nigricans (который относится к таксону Actinopterygii) и ещё одна ветвь
(корневая), которая ведёт из началу дерева. Дерево, значит, построено по
следующему принципу: из точки, являющейся корнем, выходит ветвь к исходному
таксону, от которого расходятся ветви к дочерним таксонам.
Стоит обратить внимание на тот факт, что длины всех ветвей одинаковы. Это
говорит о том, что дерево, полученное с помощью списка организмов, не
показывает эволюционных расстояний между отдельнми таксонами, а всего лишь
отображает иерархию таксонов.
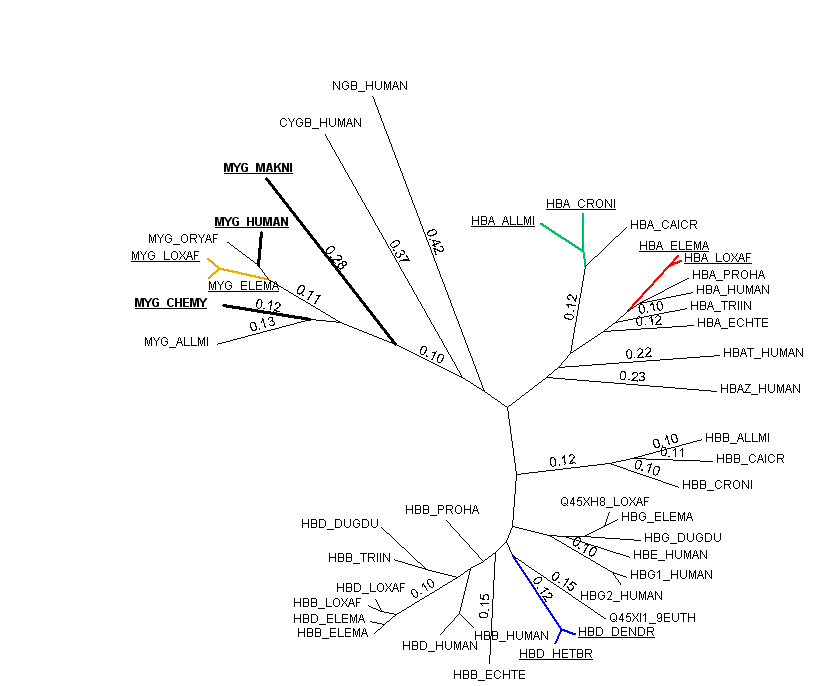
Как известно, ортологами называют белки, имеющие общее происхождение и
схожую функцию, но находящиеся в разных организмах. На картинке показаны
оранжевым, красным, зелёным и синим 4 пары явных ортологов (MYG_LOXAF и
MYG_ELEMA, HBA_ELEMA и HBA_LOXAF, HBA_ALLMI и HBA_CRONI, HBD_DENDR и HBD_HETBR
соответственно). В каждой из этих пар белки располагаются рядом на дереве и
имеют небольшие (согласно длинам ветвей) различия. Ортологичность
вышеперечисленных пар подтверждается тем, что организмы, в которых находятся
эти белки, располагаются рядом на таксономическом дереве.
Опишем способ раскраски ветвей. Выделяем ветвь, общую для обоих ортологов,
заходим в "Properties" --> "Color" и изменяем цвет. По умолчанию стоит опция
"Childs" (то есть изменения для этой ветви и для дочерних ветвей).
Так как три белка-миоглобина, выделенных на дереве, даны нам как outgroup
(внешняя группа), то корень полученного дерева находится примерно рядом с этой
группой, отделяя её и ещё часть белков от остальных белков. Вероятнее всего,
корень находится на ветви с подписью "0.10" рядом с ветвью, ведущей к белку
MYG_MAKNI.
На дереве имеется ещё несколько групп ортологов. Это, например, 3
HBA-ортолога (два из них раскрашены зелёным), к которым ведёт ветка с подписью
"0.12". Дальше можно выделить 3 явных HBB-ортолога в правой части дерева. В
левой нижней части имеются 4 ортолога, к которым ведёт ветка с подписью "0.10".
Ортологичность можно проверить, обратившись к таксономическому дереву.
Конечно, хотелось бы считать ортологами всю группу MYG-белков в левой верхней
части дерева, но ортологами эти белки не являются. По простой причине:
организмы, в которых располагаются белки, очень далеко отстоят друг от друга на
таксономическом дереве.
Паралоги, в свою очередь, — это белки одного организма, получившиеся в
результате дублирования одного гена. Они, очевидно, должны иметь похожее
происхождение и располагаться близко друг от друга на филогенетическом дереве.
Выделим пары паралогов на дереве: HBD_LOXAF и HBB_LOXAF, HBB_ELEMA и HBD_ELEMA,
HBD_HUMAN и HBB_HUMAN, HBG1_HUMAN и HBG2_HUMAN, HBAT_HUMAN и HBAZ_HUMAN. Белки
из каждой пары, по дереву, имеют очень близкое происхождение.
Также паралогами, скорее всего, являются белки CYGB_HUMAN и NGB_HUMAN, потому
что они располагаются рядом друг с другом и также рядом с предполагаемым
корнем. Хотя, по дереву, эволюционное расстояние между ними достаточно
большое...