Трансмембранные белки
1. Описание структуры 3х альфа-спиральных ТМ белков и 3х ТМ бета-баррелей
Таблица 1. Описание трансмембранных белков с известной 3D структурой |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PDB код | Тип
(спираль, баррель) |
Число цепей,
образующих одну ТМ единицу (баррель или набор спиралей) |
Число транс мембранных участков в ТМ единице | Число остатков в одном трансмембранном участке
(типичное, минимальное, максимальное) |
Толщина мембраны в ангстремах | Наклон спиралей/ тяжей к нормали | Какая мембрана, организм, "код транспортера", функция белка |
| 3d9s | спираль | 4 A[8] B[8] C[8] D[8] ( не совпадает с данными сайта http://pdbtm.enzim.hu/?m=search, там 6,6,6,6,6,6) |
32 | 18.5; 10; 23 | 30.3 ± 0.9 A | 0 ± 0° | Membrane (apical plasma membrane ) Homo sapiens (Human) 1.A.8.8.9 water channel activity ;protein binding |
| 1b9u | спираль | 1 A[1] |
1 | 27; 27; 27 | 29.8 ± 4.0 A | 41 ± 2° | Cell inner membrane Escherichia coli 3.A.2.1.1 hydrogen ion transporting ATP synthase activity, rotational mechanism |
| 2uws | спираль | 3 H[1] L[5] M[5] |
11 | 23.6; 22; 27 | 31.6 ± 0.8 A | 3 ± 0° | Cellular chromatophore membrane Rhodobacter sphaeroides (Rhodopseudomonas sphaeroides) 3.E.2.1.1 electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity ;metal ion binding |
| 1g90 | баррель | 1 A[8] |
8 | 11; 9; 13 | 25.4 ± 1.5 A | 11 ± 1° | Cell outer membrane Escherichia coli 1.B.6.1.1 porin activity ;protein binding ;structural molecule activity |
| 1tqq | баррель | 3 A[4] B[4] C[4] |
12 | 11; 9; 13 | 24.6 ± 1.4 A | 0 ± 2° | Cell outer membrane Escherichia coli 1.B.17.1.1 transporter activity |
| 1uun | баррель | 8 A[2] B[2] C[2] D[2] E[2] F[2] G[2] H[2] |
16 | 9.5; 9;10 | 40.7 ± 2.1 A | 0 ± 0° | Endoplasmic reticulum membrane Mycobacterium smegmatis 1.B.24.1.1 take part in cytolysis of cells of another organism |
-
Все рассмотренные здесь мембраны имеют в среднем примерно одинаковую толщину, около 30 A ( если быть точным, то это ширина гидрофобного слоя, а не мембраны). В среднем на трансмембранный бета-тяж приходится в 2 - 3 раза меньше остатков, чем на альфа-спираль , что вполне логично, т.к. структура альфа-спирали более сложная.
2.Аннотирование трансмембранных участков белка P19492
По данным записи Uniprot: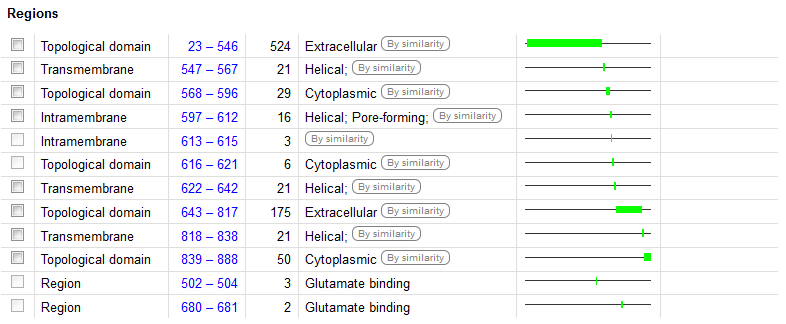
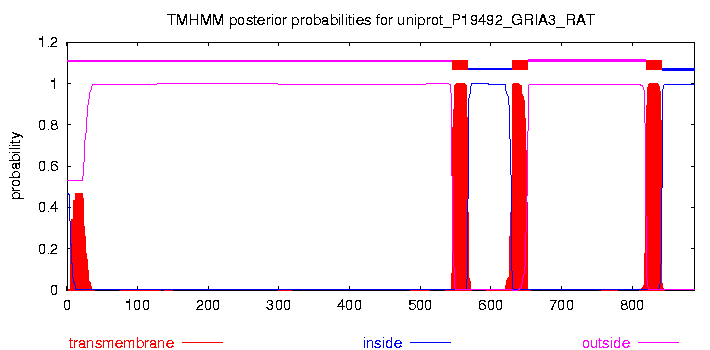
Выдача TMHMM:
# uniprot_P19492_GRIA3_RAT Length: 888 # uniprot_P19492_GRIA3_RAT Number of predicted TMHs: 3 # uniprot_P19492_GRIA3_RAT Exp number of AAs in TMHs: 78.03965 # uniprot_P19492_GRIA3_RAT Exp number, first 60 AAs: 9.96831 # uniprot_P19492_GRIA3_RAT Total prob of N-in: 0.46812 uniprot_P19492_GRIA3_RAT TMHMM2.0 outside 1 544 uniprot_P19492_GRIA3_RAT TMHMM2.0 TMhelix 545 567 uniprot_P19492_GRIA3_RAT TMHMM2.0 inside 568 629 uniprot_P19492_GRIA3_RAT TMHMM2.0 TMhelix 630 652 uniprot_P19492_GRIA3_RAT TMHMM2.0 outside 653 819 uniprot_P19492_GRIA3_RAT TMHMM2.0 TMhelix 820 842 uniprot_P19492_GRIA3_RAT TMHMM2.0 inside 843 888
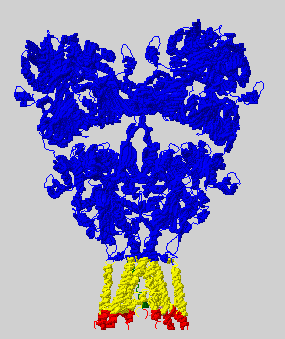
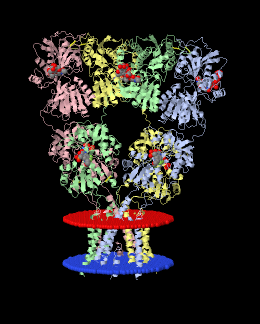
Характеристика и разметка гомолога
1.1.16.01. Glutamate-gated Ion Channel (GIC) Glutamate receptor 2 по 4 трансмембранные альфа-спирали в каждой их 4-х цепей: A - Tilt: 20° - Segments: 1(523-543), 2(572-583), 3(596-617), 4(791-813) B - Tilt: 19° - Segments: 1(523-543), 2(572-583), 3(596-617), 4(791-813) C - Tilt: 20° - Segments: 1(523-543), 2(572-583), 3(596-617), 4(791-813) D - Tilt: 20° - Segments: 1(523-543), 2(572-583), 3(596-617), 4(791-813)
Выравнивание в msf формате
Выравнивание в jar формате
Для исследуемого белка TMHMM предсказывает 3 спирали ( при этом на картинке виден как бы фрагмент 4-ой, который на самом деле является сигнальной последовательностью),при этом TMHMM не предсказал 4-ую альфа-спираль,находящуюся на участке 600-630 ( примерно). Uniprot и сравнение с гомологом предсказали все 4 трансмембранные альфа-спирали.
В целом, все предсказания довольно похожи : 3 из 4-х альфа-спиралей лишь незначительно отличаются координатами соответствующих участков во всех предсказаниях, а 4-ая в предсказаниях Uniprot и при сравнении с гомологом .
© Алиса Муравьева. Все права защищены.