Алгоритмы реконструкции деревьев
* Почти на всех деревьях фиолетовой стрелочкой отмечено место правильного укоренения : тривиальная ветвь
{ CLOB1 } vs { BACSU, GEOKA, STAES, LACLM, STRPN, ENTFA, LACAC } .
Укоренение в среднюю точку
С помощью программы retree было укоренено в среднюю точку дерево, построенное по алгоритму neighbor-joining на предыдущем занятии.Укоренение в среднюю точку нельзя сделать с деревьями, построенными методом максимальной экономии, т.к. этот метод не выдает длин ветвей => нельзя найти среднюю точку. А с деревом, построенным алгоритмом UPGMA не имеет смысла это делать, т.к. программа выдает уже укорененное дерево.
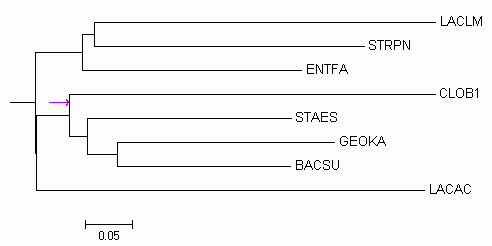
Укоренение произошло в ветвь {BACSU, GEOKA, STAES, CLOB1, LACAC } vs { LACLM, STRPN, ENTFA }.Это неправильное укоренение .Кроме того не очень понятен смысл утолщения на самом левом узле.
Использование внешней группы
.gif)
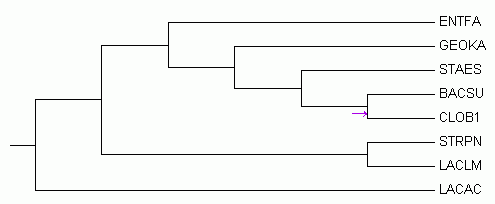
Укоренение произошло в тривиальную ветвь { LACAC } vs { LACLM, STRPN, ENTFA, BACSU, GEOKA, STAES, CLOB1 }.
Это неправильное укоренение.
Бутстрэп
+---------------LACAC
+---------100.0-|
| | +-------LACLM
| +-100.0-|
+-------| +-------STRPN
| |
| | +-----------------------GEOKA
| | |
| +-100.0-| +-------CLOB1
| | +--79.0-|
| +--99.0-| +-------BACSU
| |
| +---------------STAES
|
+---------------------------------------ENTFA
Species in order:
1. ENTFA
2. GEOKA
3. STAES
4. BACSU
5. CLOB1
6. STRPN
7. LACLM
8. LACAC
Sets included in the consensus tree ( Ветви, включенные в консенсусное дерево)
Set (species in order) How many times out of 100.00
.....*** 100.00
.....**. 100.00
.****... 100.00
..***... 99.00
...**... 79.00
Sets NOT included in consensus tree: ( Ветви, НЕ включенные в консенсусное дерево)
Set (species in order) How many times out of 100.00
..*.*... 17.00
..**.... 3.00
.*.*.... 1.00
.***.... 1.00
Комментарии:
Реконструкция филогении по сравнению с результатом fprotpars на исходном выравнивании не улучшилась (они полностью совпадают).
Топология этого дерева совпалает с правильной только по двум ветвям:
{ BACSU, GEOKA, STAES, CLOB1 } vs { LACLM, STRPN, ENTFA, LACAC }
{ BACSU, GEOKA, STAES, CLOB1, LACAC, ENTFA } vs { LACLM, STRPN }
Ветви, которые есть в дереве, построенном fprotpars, но отсутствуют в правильном:
{ BACSU, CLOB1 } vs { STAES, GEOKA, LACLM, STRPN, ENTFA, LACAC }
{ BACSU, CLOB1, STAES } vs { GEOKA, LACLM, STRPN, ENTFA, LACAC }
{ BACSU, GEOKA, STAES, CLOB1, ENTFA } vs { LACLM, STRPN, LACAC }
Ветви, которые есть в правильном дереве, но отсутствуют в дереве, построенном fprotpars:
{ BACSU, GEOKA } vs { STAES, CLOB1, LACLM, STRPN, ENTFA, LACAC }
{ BACSU, GEOKA, STAES } vs { CLOB1, LACLM, STRPN, ENTFA, LACAC }
{ BACSU, GEOKA, STAES, CLOB1, LACAC } vs { LACLM, STRPN, ENTFA }
*Рассматривались только нетривиальные ветви.
Ветви, не получившие большинства:
{ STAES, CLOB1 } vs { BACSU, GEOKA, LACLM, STRPN, ENTFA, LACAC } ( поддержка 17.00) - ветвь не верна
{ BACSU, STAES } vs { LACLM, STRPN, ENTFA, LACAC, CLOB1, GEOKA } ( поддержка 3.00) - ветвь не верна
{ BACSU, GEOKA } vs { STAES, CLOB1, LACLM, STRPN, ENTFA, LACAC } ( поддержка 1.00) - ветвь верна!
{ BACSU, GEOKA, STAES } vs { CLOB1, LACLM, STRPN, ENTFA, LACAC } ( поддержка 1.00) - ветвь верна!
Выводы: Разные методы предсказания филогенетического дерева по семейству белков IF2 выдают различные результаты, отличные от правильного ( за исключением неукорененного дерева , построенного алгоритмом Neighbor-Joining). Следовательно данное семейство белков не очень подходит для предсказания филогенетического дерева данных видов.