Реконструкция деревьев по нуклеотидным последовательностям. Анализ деревьев, содержащих паралоги.
Построение дерева по нуклеотидным последовательностям
- Последовательности 16S rRNA были получены из банка EMBL с помощью команды seqret embl:AC -sask, где АС - acception number записи EMBL, описывающий полный геном нужной бактерии.| Название | Мнемоника | AC записей EMBL | Координаты rRNA 16S |
| Bacillus subtilis | BACSU | AL009126 | 9810-11364 |
| Clostridium botulinum | CLOB1 | CP000726 | 9282-10783 |
| Enterococcus faecalis | ENTFA | AE016830 | 248466-249987 |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421-11973 |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255-60826 |
| Lactococcus lactis | LACLM | AM406671 | 511423-512971 |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006-1599559(complement) |
| Streptococcus pneumoniae | STRPI* | CP000936 | 15372-16805 |
- Положила все последовательности в единый файл 16S_rRNA.fasta и сделала выравнивание программой muscle.
- Воспользовалась для построения дерева программой fdnaml из пакета PHYLIP.
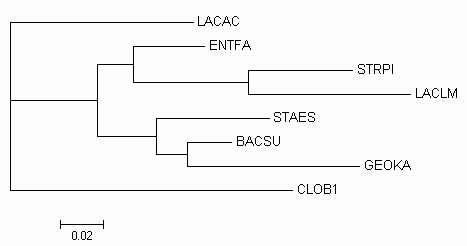
Дерево, полученное с помощью программы fdnaml
Правильное дерево
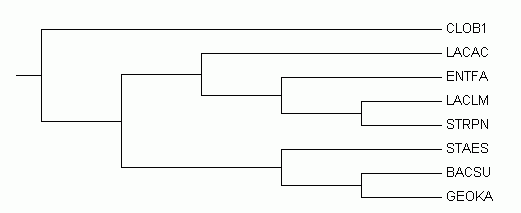
Полученное дерево совпадает с правильным 4-мя ветвями:
{BACSU, GEOKA } vs { STAES, CLOB1, LACLM, STRPN, ENTFA, LACAC }
{BACSU, GEOKA, STAES } vs { CLOB1, LACLM, STRPN, ENTFA, LACAC }
{BACSU, GEOKA, STAES, CLOB1, LACAC } vs { LACLM, STRPN, ENTFA }
{BACSU, GEOKA, STAES, CLOB1, LACAC, ENTFA } vs { LACLM, STRPN }
В целом качество реконструкции лучше по сравнению с деревьями, построенным по белкам:у всех программ сопадает с правильным деревом 3-4 ветви.Но деревьев. полностью совпавших с правильным, нет.
Построение и анализ дерева, содержащего паралоги
Была создана база данных для BLAST'а из файла proteo.fasta, затем в ней были найдены гомологи белка CPLX_BACSU (evalue - 0.001) Затем были отобраны белки, принадлежащие выбранным ранее бактериям. Дерево построено программой fneighbor (алгоритм Neighbor-joining )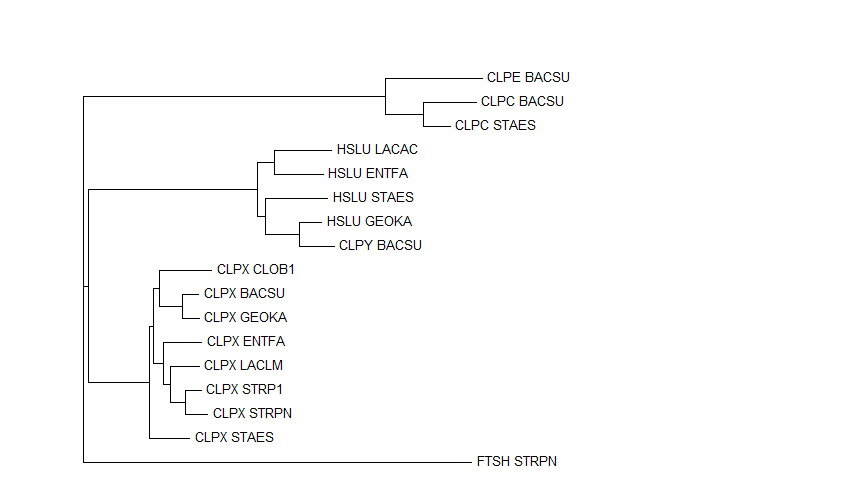
| Примеры ортологов:
CLPC_BACSU & CLPC_STAES HSLU_ENTFA & HSLU_LACAC CLPX_BACSU & CLPX_GEOKA |
Примеры паралогов:
HSLU_ENTFA & CLPX_ENTFA CLPC_STAES & CLPX_STAES HSLU_GEOKA & CLPX_GEOKA |