Max target sequences - устанавливает максимальное количество искомых последовательностей.
Short queries - в случае поиска коротких последовательностей, в отличие от длинных, следует использовать другие параметры по умолчанию. Эта кнопка устанавливает данные параметры.
Expect threshold - верхний порог E-value. Последовательности с большим, чем установленное E-value, не показываются.
Word size - размер слов, по которым производится поиск последовательностей. Чем бельше длина слов, тем быстрее работает алгоритм.
Max matches in a query range - задает ограничение по максимальному числу совпадений между введённой последовательностью и последовательностями в базе данных
Matrix - устанавливает вес для выровненных пар.
Gap Costs - устанавливается штраф за появление нового гэпа.
Compositional adjustments - позволяет избежать ложных результатов, связанных с участками малой сложности
Filters and Masking- дополнительные фильтры
В параметрах поиска были изменены "Max target sequences" до 20000 и "Word size" до 2 (из-за малого количества последовательностей), остальные пункты по умолчанию.
В результате работы Blast получена таблица.
Из списка последовательностей были выбраны 7 гомологичных, для них получено множественное выравнивание и найдены участки гомологии (рис.1). Имеются 100% консервативные столбцы, между которыми высокая плотность консервативных позиций и нет гэпов.
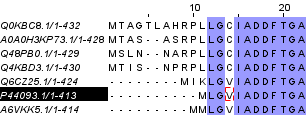
рис.1
Были выбраны 2 белка: FOL1_DICDI(Folic acid synthesis protein FOL1) и U1LPR7_9MICO(7,8-dihydroneopterin aldolase), для них построена карта сходств (рис.2).
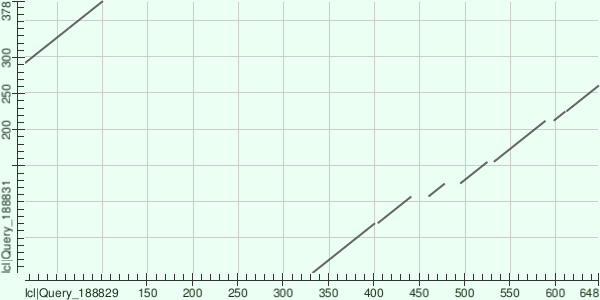
рис.2
Видно, что у белков произошла транслокация.
Поисковой запрос: "I want to sleep"
В параметрах поиска были изменены "Max target sequences" до 20000 и "Word size" до 2, остальные пункты ео умолчанию. Результаты приведёны ниже (рис.3).
рис.3