| Командная строка | Функция |
cp Human/chr16.fasta fp.delta/chr16.fasta |
Копиравание chr16.fasta |
hisat2-build chr16.fasta task2.fasta |
Индексация референса |
cp ../Human/reads/chr16.fastq chr16.fastq |
Копирование ридов |
fastqc chr16.fastq |
Обработка FastQC |
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr16.fastq chr16_trim.fastq TRAILING:20 MINLEN:50 |
Обрезание концов |
hisat2 -x task2.fasta -U chr16_trim.fastq -S chr16_aligntoref.sam --no-softclip --no-spliced-alignment |
Картирование чтений из fastq (по индексированной последовательности) |
samtools view -b chr16_aligntoref.sam -o chr16_align.bam |
Конвертнация в .bam |
samtools sort chr16_align.bam chr16_align_sorted |
Сортировка по координате в начале чтения |
samtools index chr16_align_sorted.bam |
Индексация отсортированного .bam |
samtools mpileup -u -f chr16.fasta -o chr16_poly.bcf chr16_align_sorted.bam |
Создание файла с полиморфизмами |
bcftools call -cv -o chr16_poly.vcf chr16_poly.bcf |
Создание файла со списком отличий между референсом и чтениями в формате .vcf |
vcftools --vcf chr16_poly.vcf --remove-indels --recode --out chr16_poly_niind |
Удаление инделей |
convert2annovar.pl -format vcf4 chr16_poly_niind.recode.vcf -outfile chr16_poly.avinput |
Конвертнация в .vcf4 (для annotate_variation.pl) |
annotate_variation.pl -out chr16_annotate -build hg19 -dbtype refGene chr16_poly.avinput /nfs/srv/databases/annovar/humandb.old/ |
Аннотация по refgene |
annotate_variation.pl -filter -out chr16_annotate_dbsnp -build hg19 -dbtype snp138 chr16_poly.avinput /nfs/srv/databases/annovar/humandb.old/ |
Аннотацтия по dbsnp |
annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr16_1000g chr16_poly.avinput /nfs/srv/databases/annovar/humandb.old/ |
Аннотацтия по 1000 genomes |
annotate_variation.pl -regionanno -build hg19 -out chr16_gwas -dbtype gwasCatalog chr16_poly.avinput /nfs/srv/databases/annovar/humandb.old/ |
Аннотацтия по GWAS |
annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 -out chr16_clinvar chr16_poly.avinput /nfs/srv/databases/annovar/humandb.old/ |
Аннотацтия по Clinvar |
Обрезали нуклеотиды с качеством меньше 20 (вероятность ошибки чтения больше 0,01) с концов и убрали чтения длиной меньше 50 нуклеотидов
Команда, с помощью которой чтения были обрезаны:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr16.fastq chr16_trim.fastq TRAILING:20 MINLEN:50
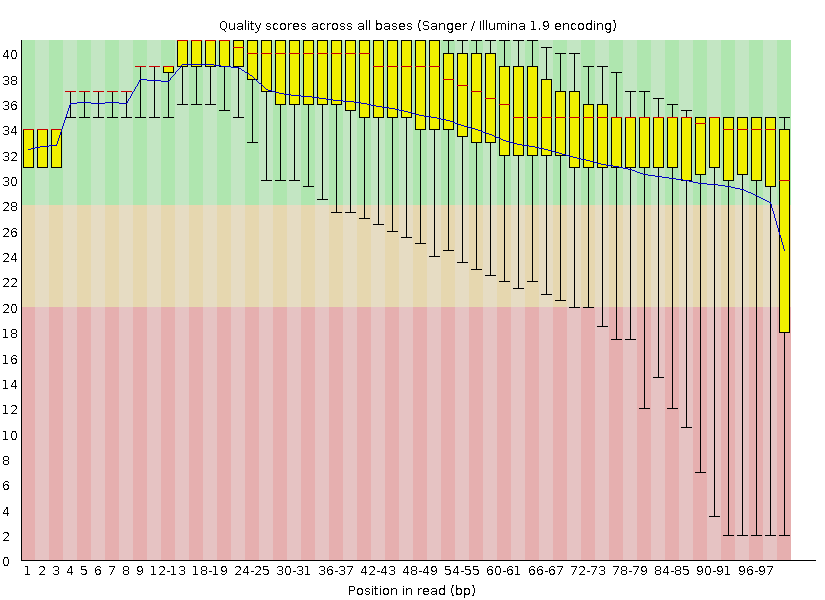
До обрезки:

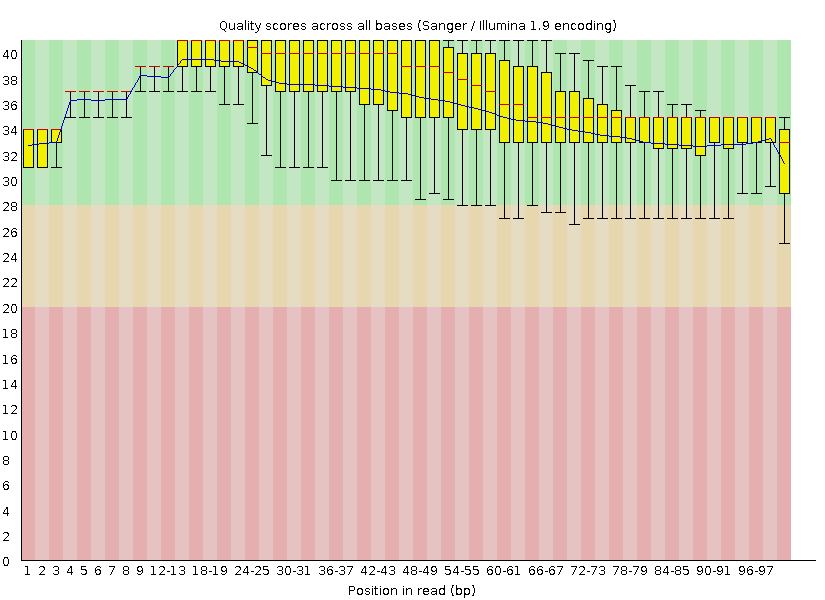
После обрезки:
Результаты:
Input Reads: 3965 Surviving: 3798 (95,79%) Dropped: 167 (4,21%)
В результате ~96% сохранились, что не особо отличаетя от изначальных данных
99.08% чтений были картированы на хромосому, качество можно считать довольно высоким
3798 reads; of these:
3798 (100.00%) were unpaired; of these:
32 (0.84%) aligned 0 times
3763 (99.08%) aligned exactly 1 time
3 (0.08%) aligned >1 times
99.16% overall alignment rate
Всего 65 полиморфизмов, из них 63 SNP, 1 индель и 1 вставка
| Позиция | Тип | Референс | Чтение | Глубина покрытия | Качество чтений |
| 11348273 | Замена | T | A | 10 | 4.12853 |
| 11444454 | Делеция | gaaaaaaaaaaa | gaaaaaaaaaa,gaaaaaaaaa | INDEL | 9.03477 |
| 31095171 | Замена | C | T | 53 | 221.999 |
*К сожалению, вставки не было, поэтому пришлось описать 2 замены. Делеция только с инделем
База данных refseq в annovar распределяет SNP по их расположению в последовательности
| Тип SNP | Количество SNP |
| intronic | 23 |
| exonic | 9 |
| intergenic | 19 |
| UTR3 | 3 |
| UTR5 | 3 |
| upstream | 5 |
| downstream | 1 |
Расположение экзонных мутаций: TNR2, PRM3, PRM2, PRM1, RMI2, PRSS53, HERPUD1
В базе данных 1000genomes нашлось 58 SNP, так же как и в базе dbsnp
По базе данных 1000genomes: медиана 0,4715455, среднее арифметическое 0,464222799
По базе данных dbsnp: всего SNP, имеющих rs, оказалось 58, а без - 6
Два SNP ассоциированы с ожирением и метаболический синдром, третий - с болезнью Паркинсона
gwasCatalog Name=Obesity-related traits chr16 11374866 11374866 G T hom 221.999 . gwasCatalog Name=Parkinson's disease chr16 31095171 31095171 C T hom 221.999 . gwasCatalog Name=Metabolic syndrome chr16 56969148 56969148 G A het 225.009 .
Отсутствуют результаты по Clinvar