Попробовал. Оценил. Очень понравилось, удалось неплохо поиграться :-).
Чтобы выполнить эту задачу, я предварительно оценил зону контакта лиганда с белком.
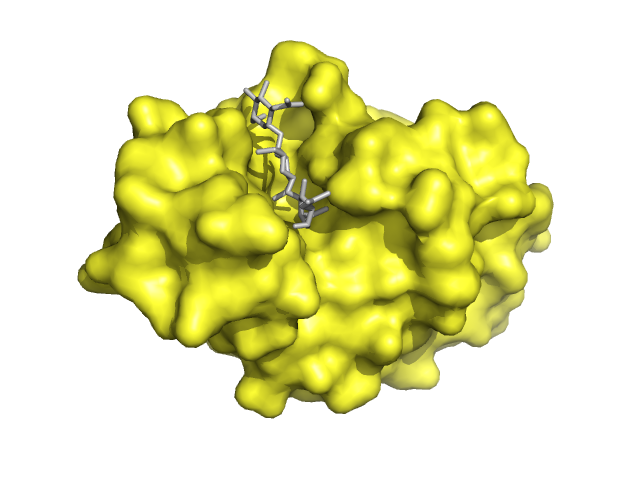
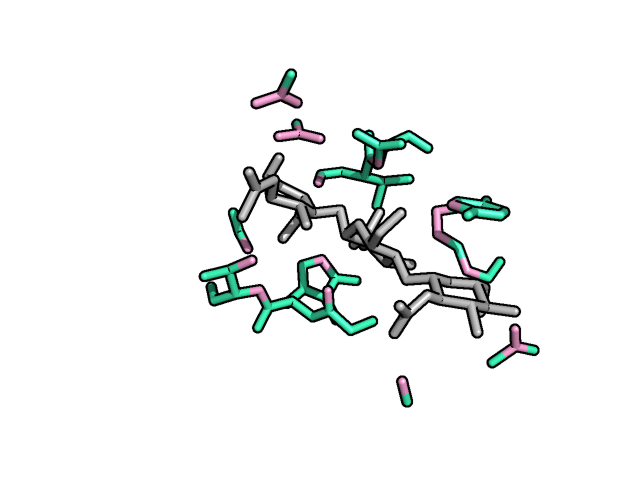
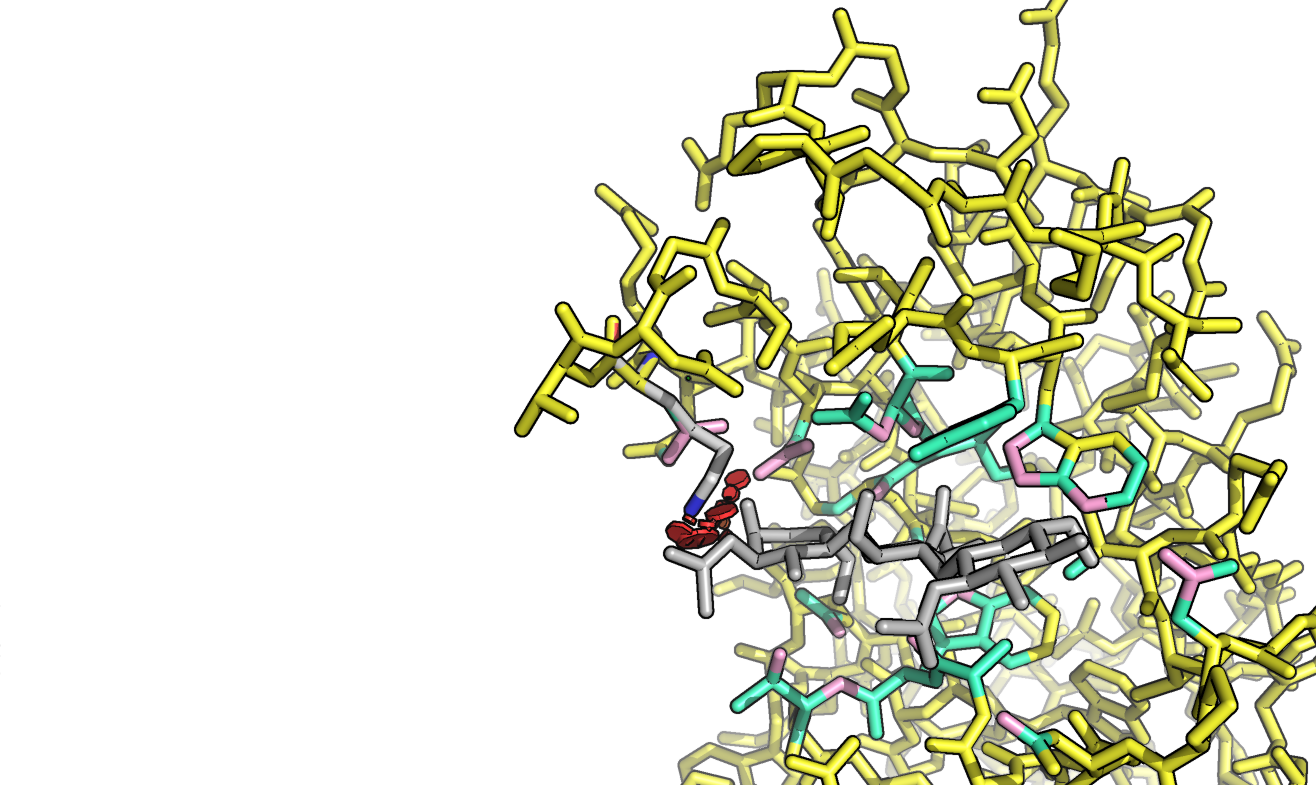
На картинке снизу желтым представлен целый белок – 1LMP, тогда как серым представлена молекула его лиганда, состоящая (по сути) из двух частей: NAG и NDG.
№2 Проведите мутацию в белке, которая по вашему мнению должна привести к потере связывания с лигандом

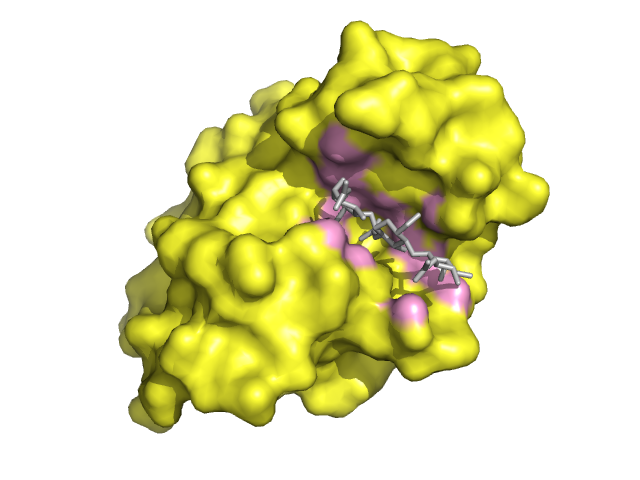
Далее, я окрасил в розовый цвет те атомы белка, которые находятся на расстоянии не более чем 3.5 ангстрем, чтобы оценить зону контакта (на таком расстоянии возможны самые разные нековалентные взаимодействия). Вот, что получилось:
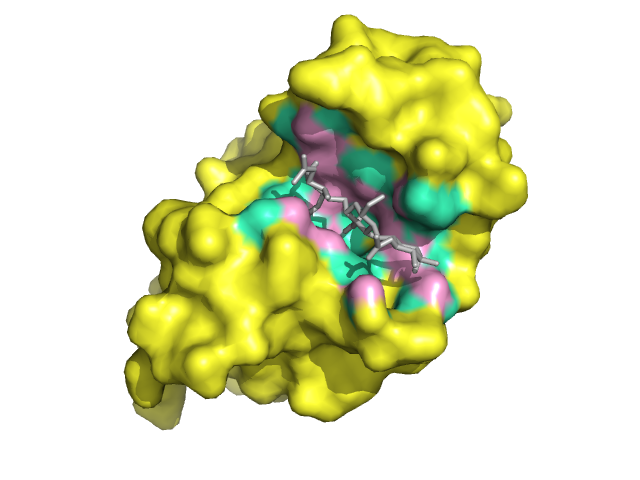
На лекциях по структурной биоинформатике нам говорили, что гидрофобный контакт можно оценить на расстоянии не более чем 5 ангстрем. Внизу, по сути, представлена уже вышеописанная картинка, только докрашенная светло-зеленым цветом (этим цветом покрашены те атомы, что находятся на расстоянии от лиганда 3.5-5.0 ангстрем).
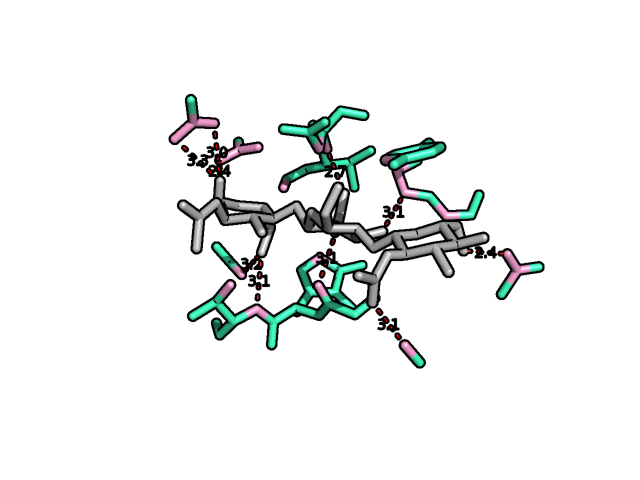
На картинке ниже, слева, можно увидеть только лиганд и окружающие его остатки, а справа – то же самое, только представлены водородные связи.
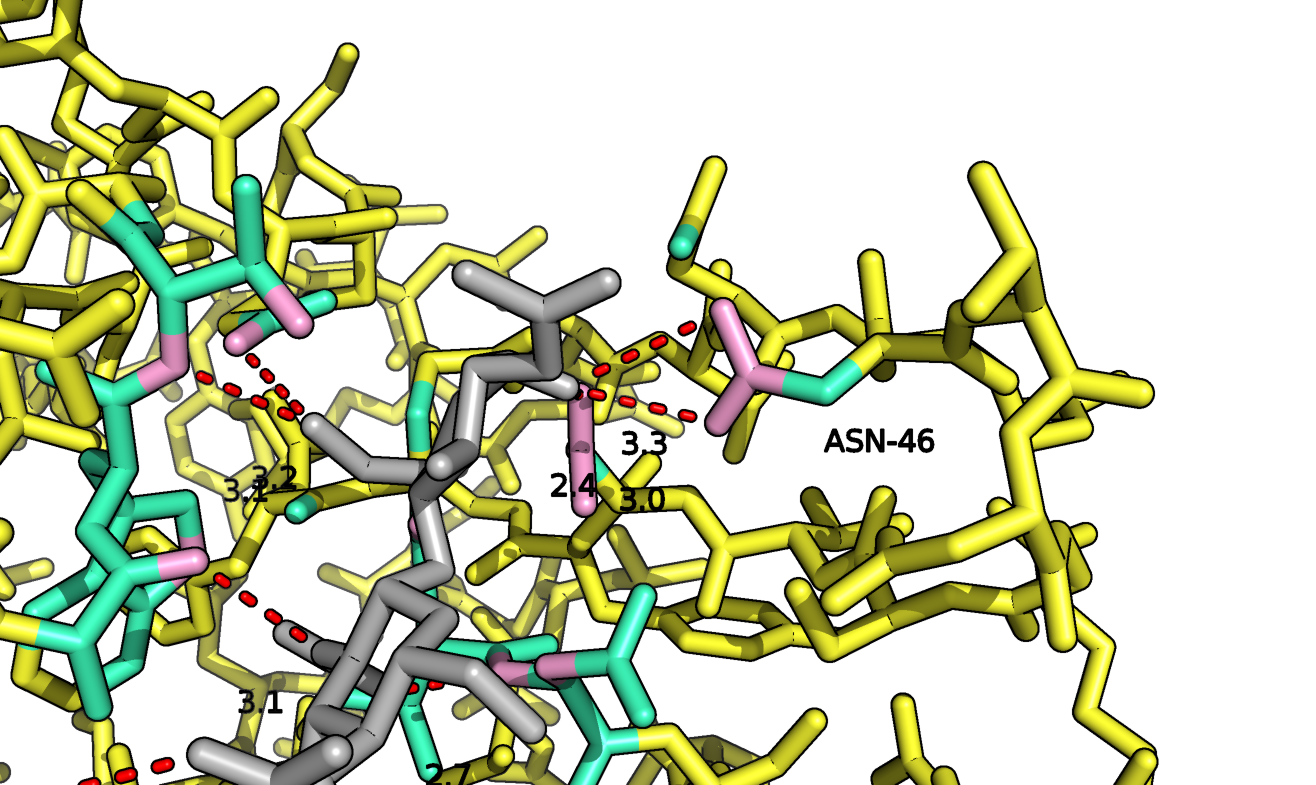
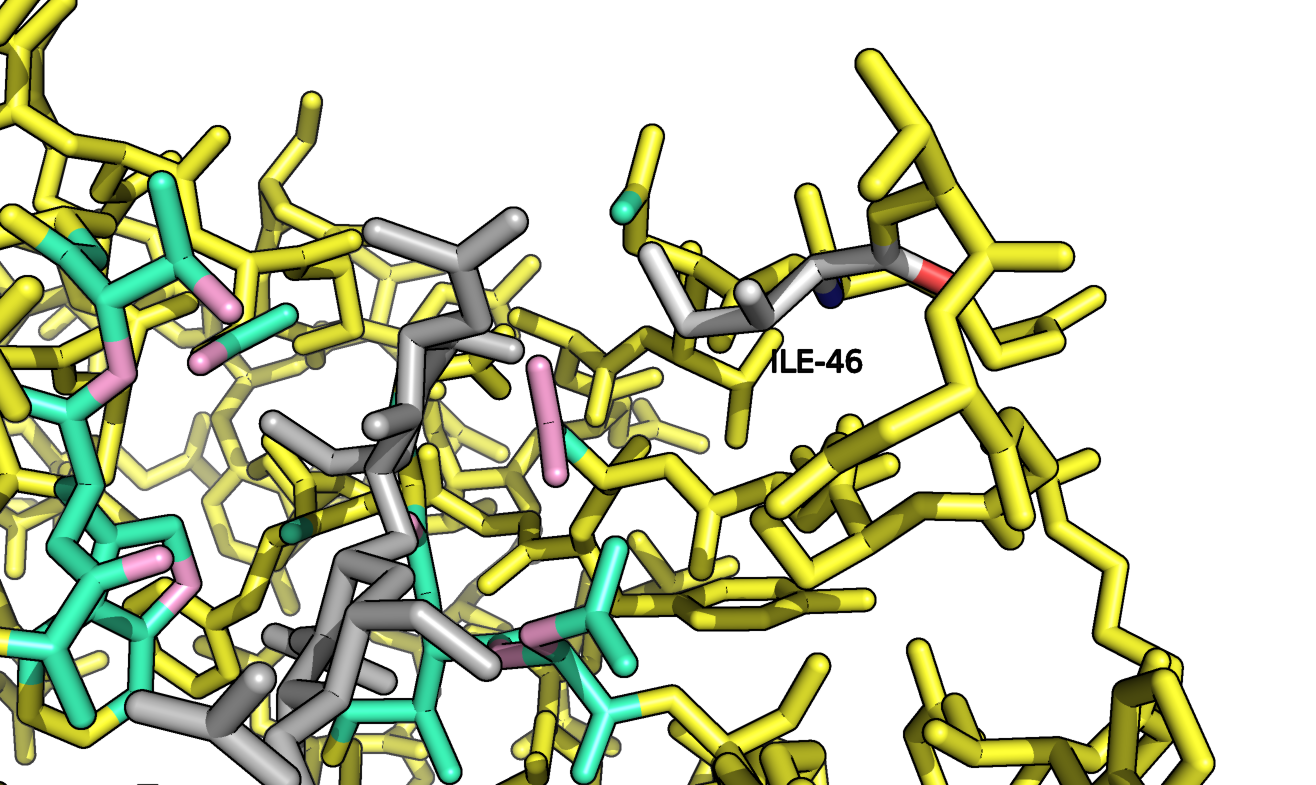
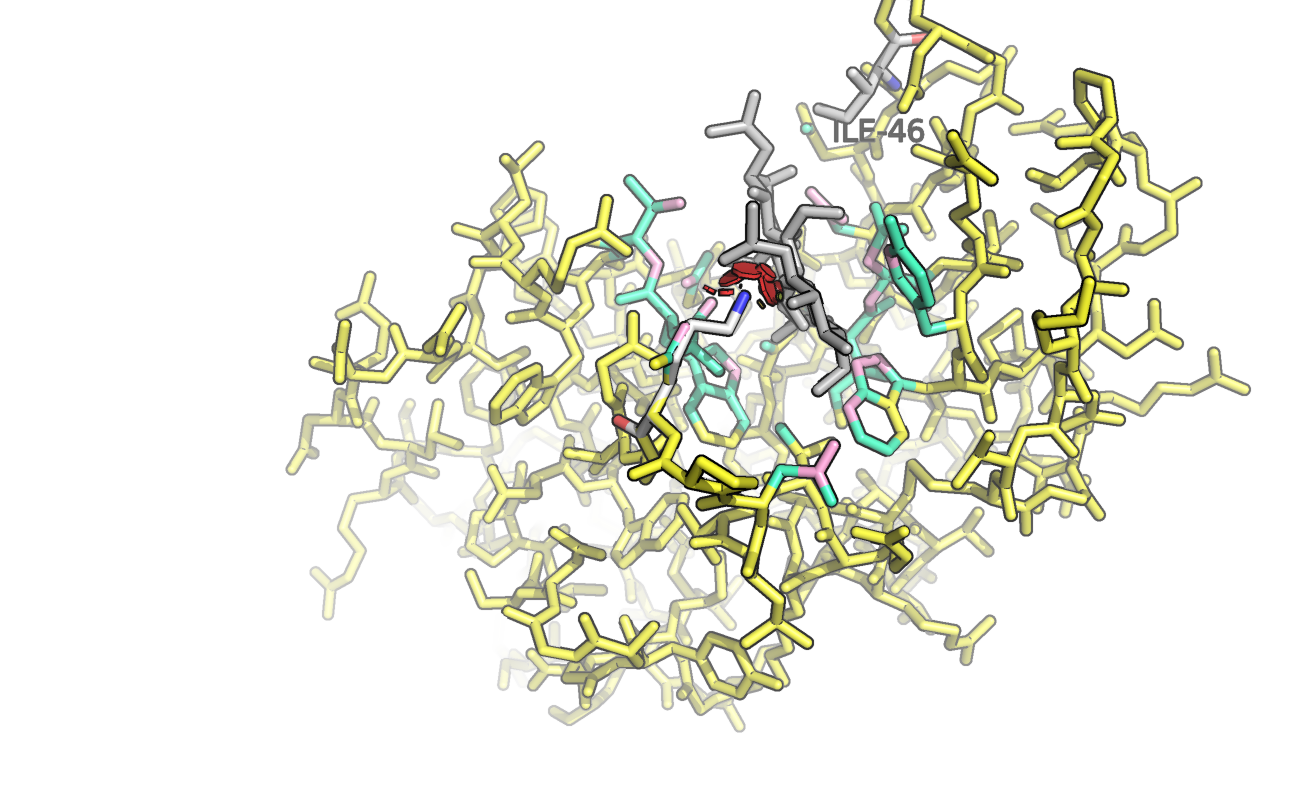
Таким образом, было решено сделать такую мутацию, которая затрагивала бы аминокислоту, образующую водородную связь с молекулой лиганда. Но поскольку таких (водородных) связей достаточно много, пришлось бы мутировать несколько остатков, чтобы белок прекратил прочно связывать лиганд. Было решено мутировать только одну аминокислоту для наглядности. Выбор пал на аспарагин 46 (ASN 46), который образует аж 2 водородные связи с молекулой лиганда. Заменена она была на один из ротамеров изолейцина (ILE 46). На рисунках внизу представлена структура белка с лигандом до (слева) и после (справа) мутации.
В последствии было решено изменить остаток не на ILE46, а на LYS46. Ротамер новой аминокислоты стерически мешает лиганду занять оптимальное место в соответствующем сайте белка.
Также было решено мутировать остаток ASN 103, поскольку он находится ближе всего к лиганду. Его я поменял на остаток лизина, причем на такой ротамер, который максимально стерически мешает лиганду. Это продемонстрировано на рисунке снизу. Такая мутация наверняка будет препятствовать связыванию лиганда в активном центре белка.
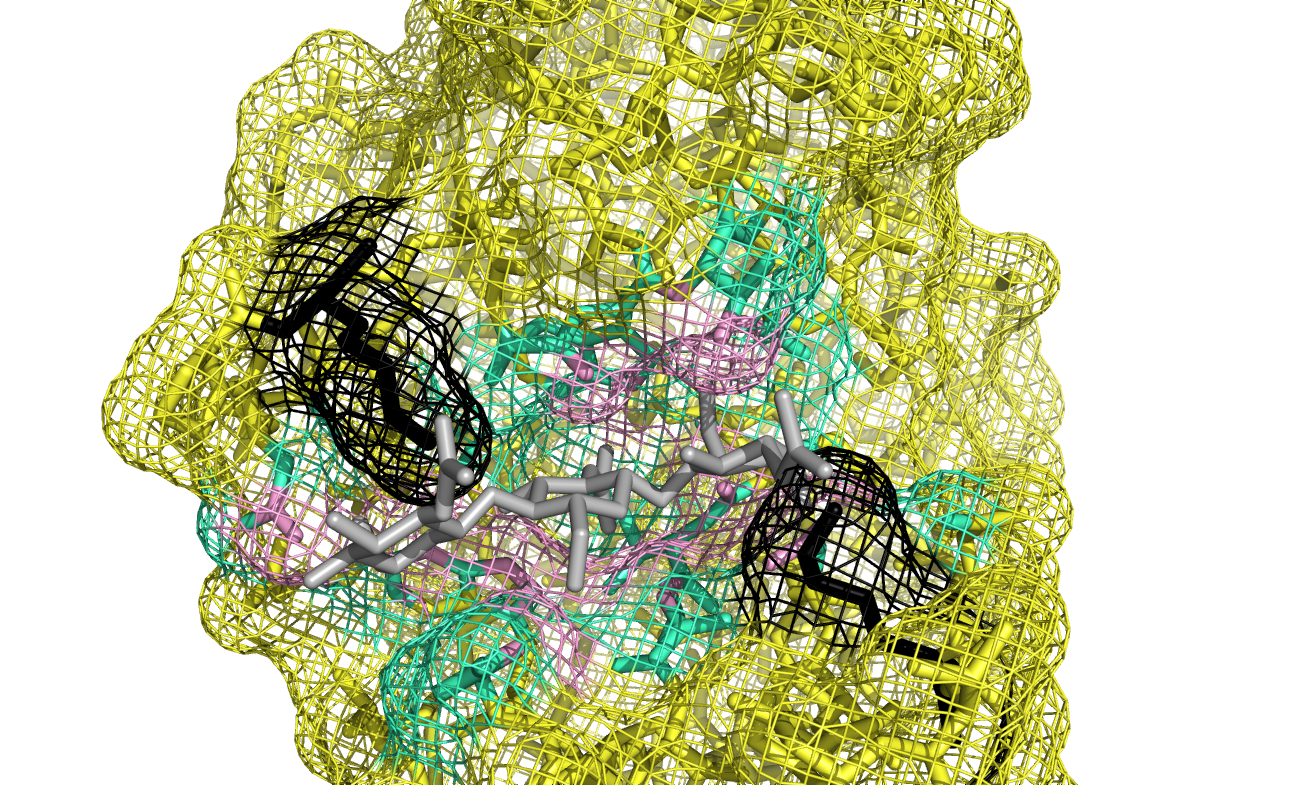
Чтобы подвести итог показываю картинку, на которой изображено, как две мутации накладываются на лиганд (мутированные остатки показаны черным цветом).
Ниже представлен анимационный ролик, на котором совмещаются структура с двумя мутациями (см. выше) со структурой, у которой мутаций нет. Первая представлена циановым цветом, а мутированные остатки – желтые; вторая – красная, остатки (исходные, не мутированные) – синие.
Показываю на картинке.
Привожу изображение полилейциновой спирали и скрипт.

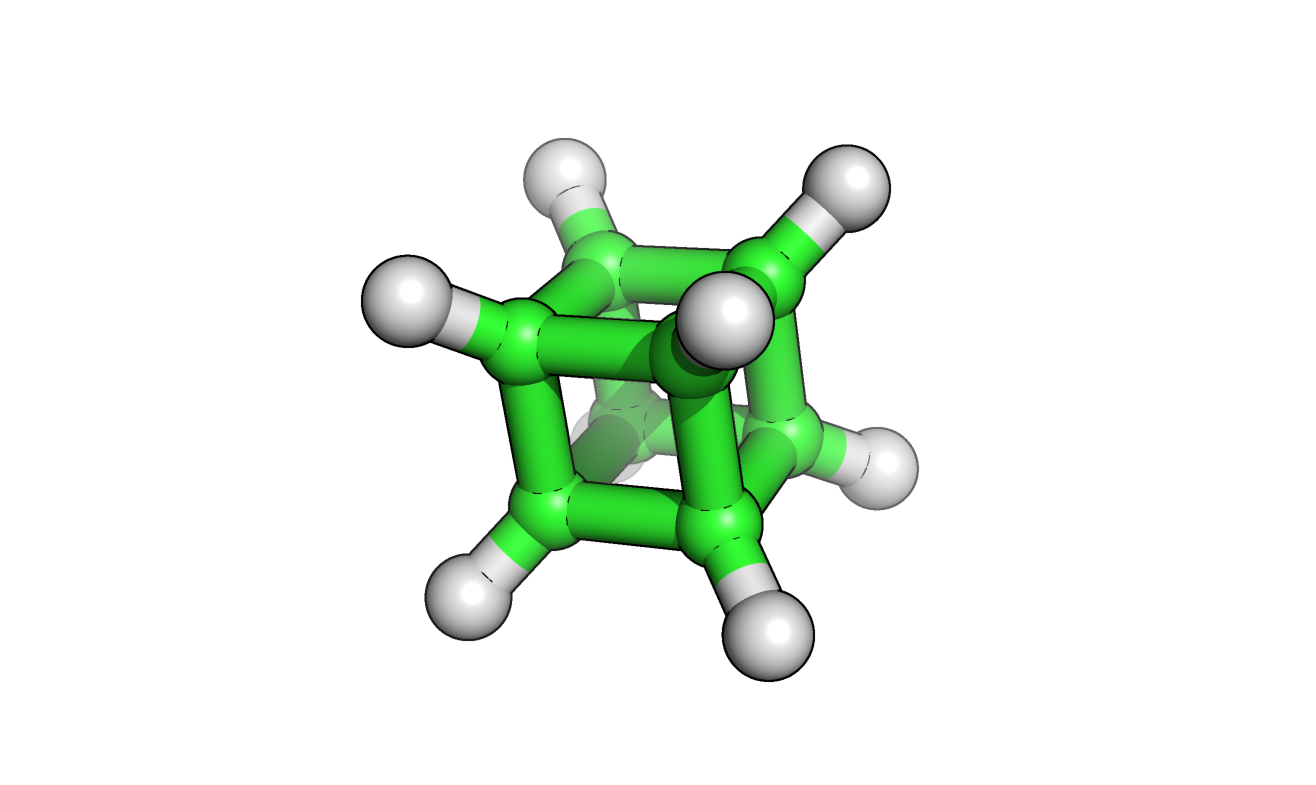
Привожу изображение кубана.