Будем изучать как реализован контроль температуры в молекулярной динамике на примере GROMACS. Объект исследования это одна молекула этана.
А начнем с того, что подготовим файл координат и файл топологии.
make_ndx -f box_38.gro -o 1.ndx
editconf -f box_38.gro -o et1.gro -n 1.ndx
editconf -f et1.gro -o et.gro -d 2 -c
В итоге получился следующий файл: et.gro. А файл топологии был исправлен: et.top.
После этого были скачены для работы следующие файлы:
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Для получения файлов на вход молекулярно-динамическому движку mdrun с помощью grompp
был использован следующий скрипт:
#!/bin/bash
for i in {be,vr,nh,an,sd}; do
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
done
Получилось, как и ожидалось, 5 trp файлов. Подаем их на вход mdrun, добавив в скрипт следующую строчку:
mdrun -deffnm et_${i} -v -nt 1
Теперь переходим к анализу результатов. Начнем с визуального анализа. Для каждой из 5 систем проведем конвертацию в pdb и просмотрим в PyMol.
Добавим в скрипт такую строчку:
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
А теперь опишем полученные pdb файлы!
et_an.pdb - молекула колеблется возле одного положения, нет вращений, есть слабые колебания длин связей и углов.
et_be.pdb - в самом начале колеблятся только длины связей и углов.
et_nh.pdb - тут наблюдается вращение метильных групп относительно С-С связи, а также небольшие колебания валентных углов.
et_sd.pdb - молекула меняет конформацию и положение в пространстве (довольно хаотично).
et_vr.pdb - наблюдаются смены конформаций и колебания длин связей и валентных углов; молекула большую часть времени находится в заторможенной конформации.
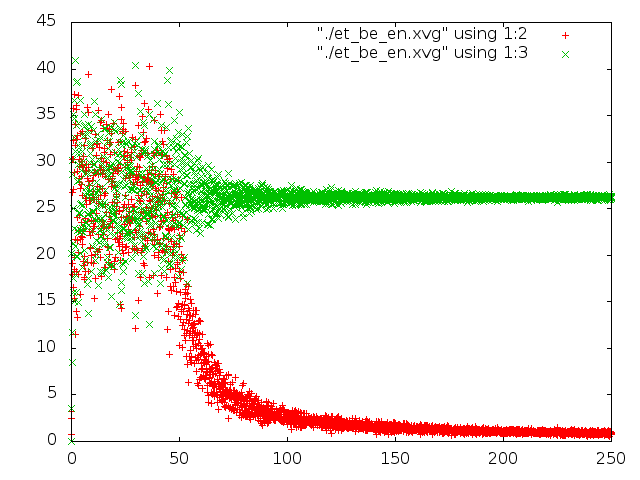
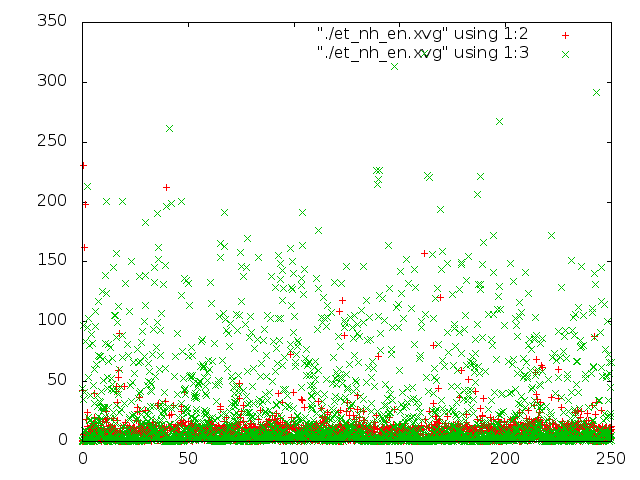
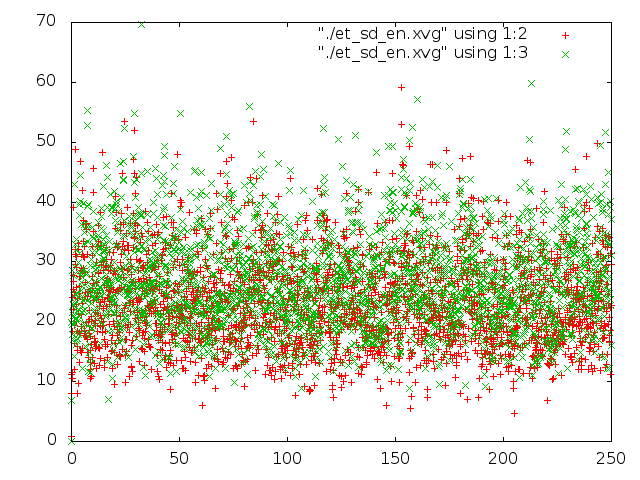
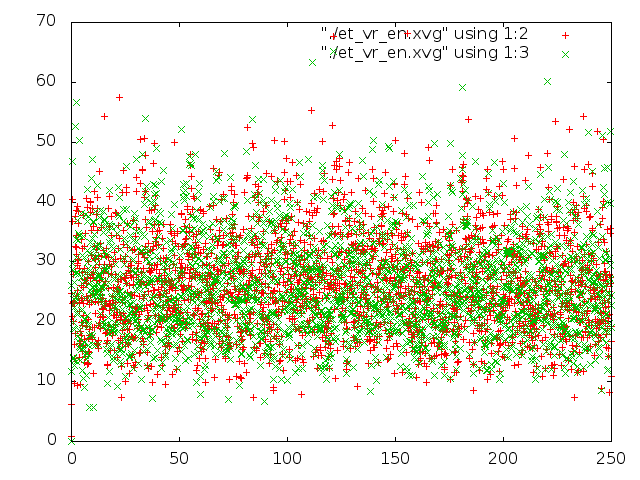
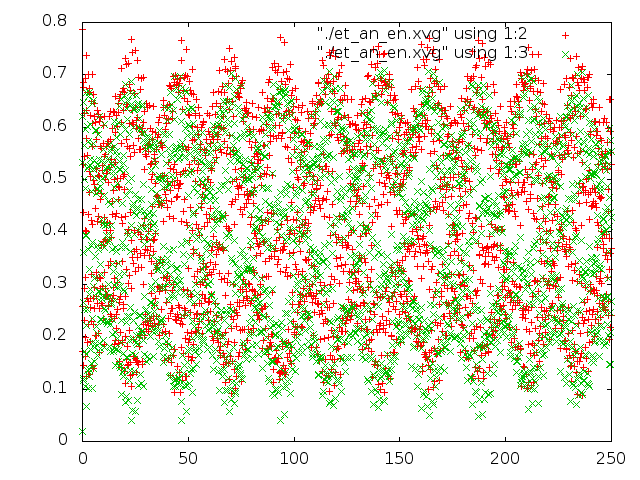
Далее, надо сравнить потенциальную энергию связи и кинетическую энергию для каждой из 5 систем. Добавим в скрипт следующую строчку:
g_energy -f et_${i}.edr -o et_${i}_en.xvg
А теперь построим графики изменения энергий с помощью gnuplot. Для этого был создан файл (для каждой из систем) такого содержания:
set terminal png
set output "et_vr_en.png"
plot "./et_vr_en.xvg" using 1:2, "./et_vr_en.xvg" using 1:3
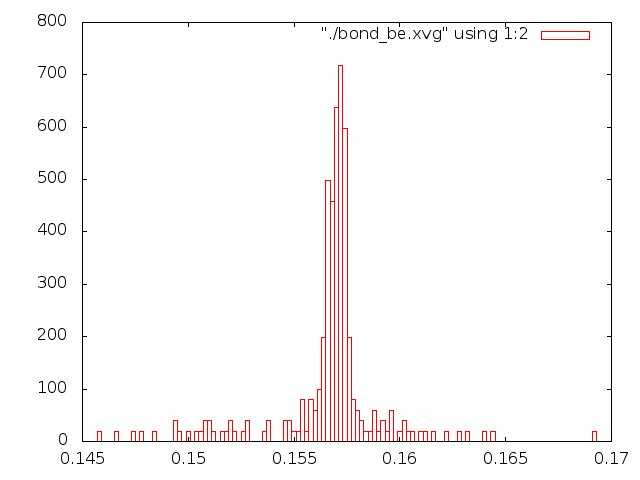
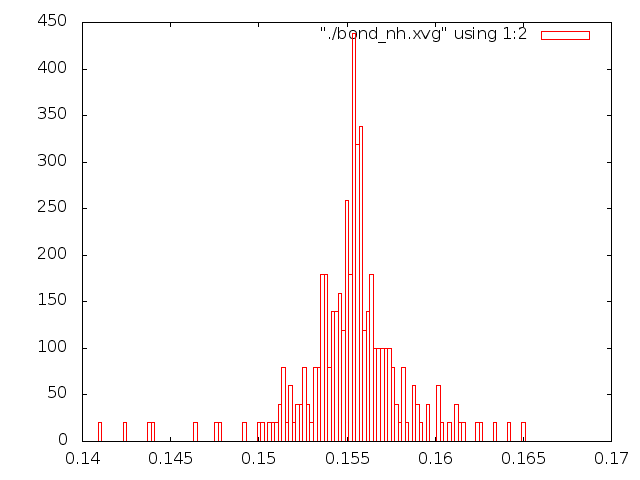
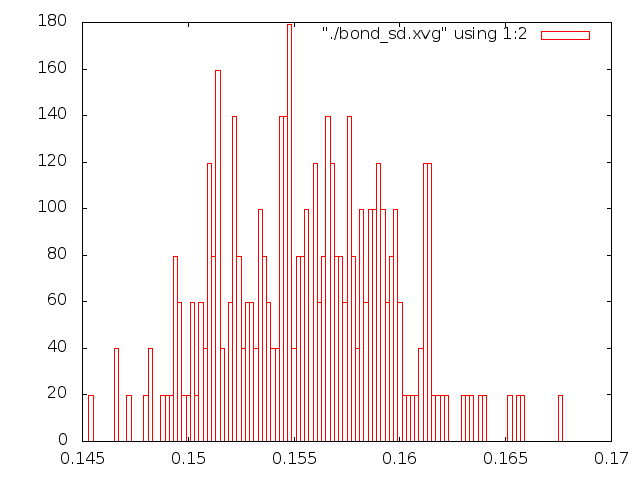
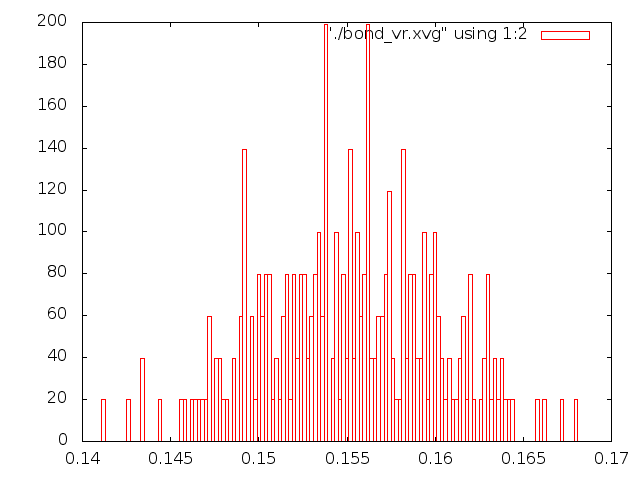
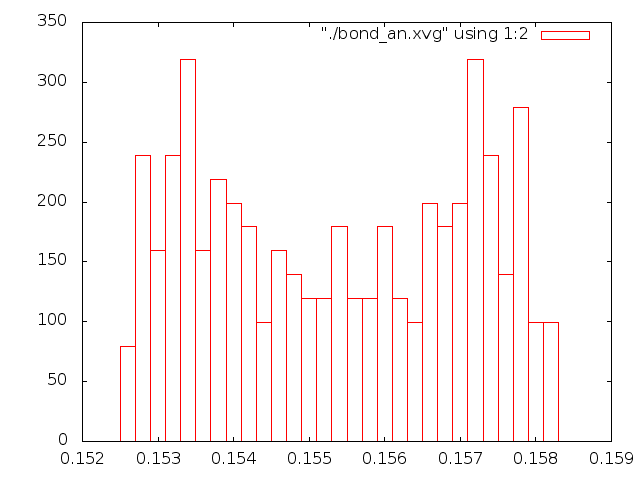
Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью.
Для этого в текстовом редакторе создадим файл b.ndx со следующим содержимым:
[ b ]
1 2
Запустим утилиту по анализу связей g_bond (добавив соответствующую строку в скрипт):
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Ну и построим графики распределения длинн связей (boxes в gnuplot).
Привожу все картинки (что за файлы и откуда, написано на графиках):