Молекулярная динамика биологических молекул в GROMACS
Изучение результатов работы на суперкомпьютере
Любой анализ начинают с визуального анализа движений молекул! Вводим следующую команду:
trjconv -f dna_md.xtc -s dna_md.tpr -o dna_pbc_1.pdb -skip 20 -pbc mol
Привожу полученный pdb файл.
Надо понять, на каком этапе происходит переход из А в В форму. Вообще обе они правозакручены, но А считается
более компактной. Если брать в расчет именно компактность, то уже на 5-6 модели ДНК переходит из А в B форму:
REMARK GENERATED BY TRJCONV
TITLE Protein in water t= 800.00000
REMARK THIS IS A SIMULATION BOX
CRYST1 50.833 48.976 54.527 90.00 90.00 90.00 P 1 1
MODEL 5
Видно, что время моделирования - 0.8 нс.
Теперь определим средне-квадратичное отколнение в ходе моделирования. Так как у нас происходит конформационный переход сначала расчитаем
отклонение в ходе всей симуляции относительно стартовой структуры.
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_1
И относительно каждой предидущей структуры на растоянии 400 кадров. Если ближе к концу закончился конформационный переход, то отколнение должно уменьшаться!
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_2 -prev 400
Привожу файлы pdb файл и rms_2
Для большей наглядности были созданы два графика:
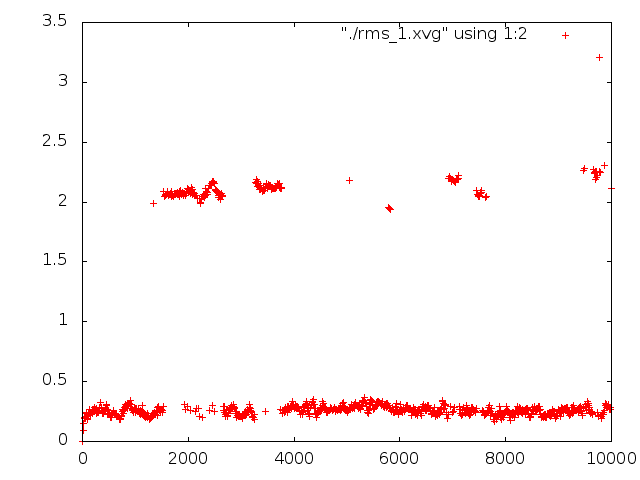
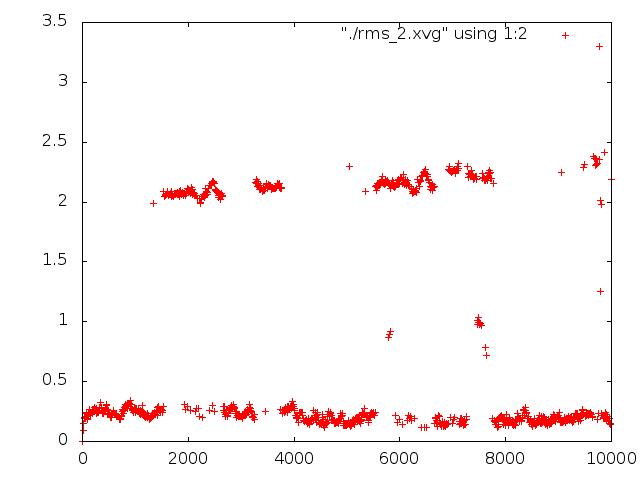 Вроде бы, ничем принципиальным графики не отличаются, по последнему графику видно, что квадратичное отклонение, действительно, становится меньше.
Теперь определим изменение гидрофобной и гидрофильной поверхности в ходе конформационного перехода.
Вроде бы, ничем принципиальным графики не отличаются, по последнему графику видно, что квадратичное отклонение, действительно, становится меньше.
Теперь определим изменение гидрофобной и гидрофильной поверхности в ходе конформационного перехода.
g_sas -f dna_md.xtc -s dna_md.tpr -o sas_dna.xvg
Получили файл sas_dna.xvg. И теперь построим зависимость изменения гидрофобной гидрофильной поверхностей доступных растворителю от времени.
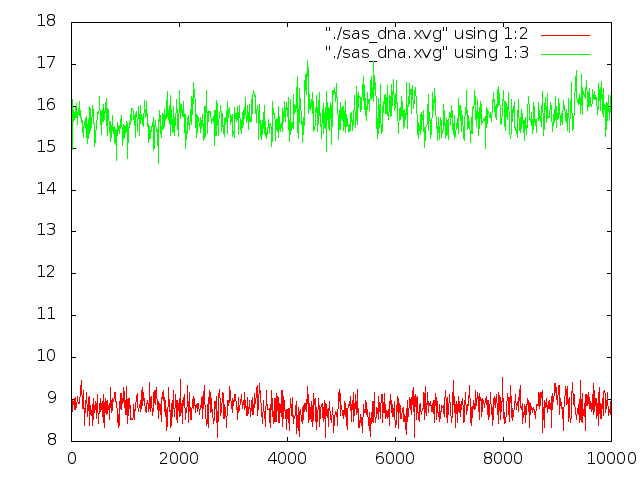 Зеленым цветом показана гидрофильность, а красным - гидрофобность. Отчетливо видно, что гидрофобность (поверхности) остается неизменной, тогда как
гидрофильность имеет пик ближе к концу.
Традиционным анализом для ДНК является расчёт колчества образуемых водородных связей.
Если мы будем исследовать связи между ДНК и ДНК, то это будут водородные связи между цепями ДНК. Для конца траектории:
Зеленым цветом показана гидрофильность, а красным - гидрофобность. Отчетливо видно, что гидрофобность (поверхности) остается неизменной, тогда как
гидрофильность имеет пик ближе к концу.
Традиционным анализом для ДНК является расчёт колчества образуемых водородных связей.
Если мы будем исследовать связи между ДНК и ДНК, то это будут водородные связи между цепями ДНК. Для конца траектории:
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_dna
Получился файл hbond_dna.xvg.
Вот какая получилась зависимость:
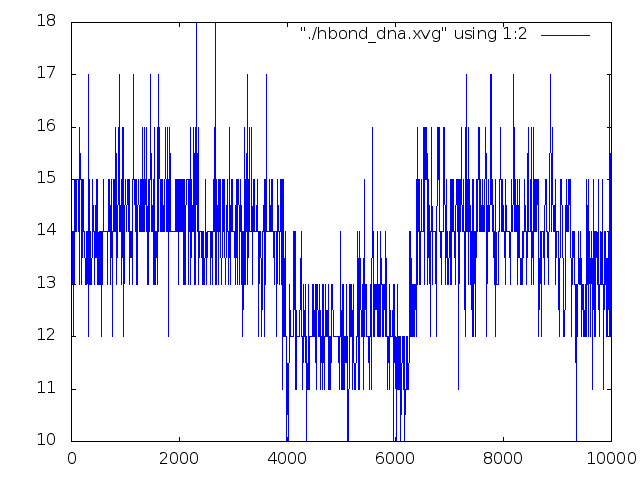 Вообще, исходно в дуплексе было 14 водородных свзяей (так как последовательность одной из цепочек - GATCTA). На графике
видно, что число водородных связей разбросано от 10 до 18, но это единичные выбросы, большая часть значений колеблется на уровне 12-15. Это близко к 14.
Изменение в числе водородных связей вижно где-то посередине, но ближе к концу это значение возвращается к норме. Можно сделать вывод, что принципиально число водородных связей не меняется.
Не менее интересно будет изучить количество вдородных связей ДНК-Вода!
Вообще, исходно в дуплексе было 14 водородных свзяей (так как последовательность одной из цепочек - GATCTA). На графике
видно, что число водородных связей разбросано от 10 до 18, но это единичные выбросы, большая часть значений колеблется на уровне 12-15. Это близко к 14.
Изменение в числе водородных связей вижно где-то посередине, но ближе к концу это значение возвращается к норме. Можно сделать вывод, что принципиально число водородных связей не меняется.
Не менее интересно будет изучить количество вдородных связей ДНК-Вода!
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_sol
Получился файл hbond_sol.xvg.
А вот какая получилась зависимость:
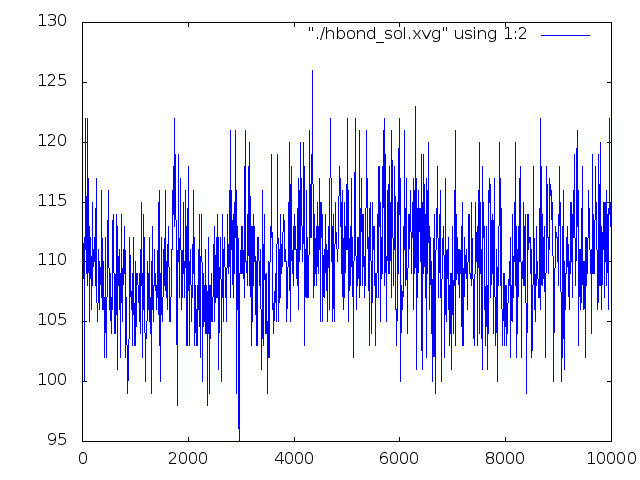 Ну, здесь видно, что в конце графика среднее число водородных связей такое же, как и в начале. Это число колеблется в пределах 95-125, однако в самом начале
и в конце это число приблизительно равно 110.
Из этого можно сделать вывод, что небольшие изменения в количестве водородных связей не мешают ДНК перейти из одной формы в другую.
Ну, здесь видно, что в конце графика среднее число водородных связей такое же, как и в начале. Это число колеблется в пределах 95-125, однако в самом начале
и в конце это число приблизительно равно 110.
Из этого можно сделать вывод, что небольшие изменения в количестве водородных связей не мешают ДНК перейти из одной формы в другую.






