Первый шаг - это построение выравнивания последовательности из структуры ID: 1lmp и выбранного мною белка (LYS_BPPS1). Выравнивание было строили в программе ClustalW и сохранили в формате PIR.
Далее, поменяем полученный файл выравнивания в соответствии с тем, как описано в задании. Получился такой файл.
Потом, после соответствующих манипуляций был получен следующий файл.
Далее, нужно дописать скрипт по данной заготовке. Необходимо отредактировать строчки, в которых указаны какие водородные связи белка с лигандом должны быть в будущей модели. Для этого нужно знать, какие водородные связи присутствуют в образце! Привожу картинку, на которой изображены водородные связи между белком и молекулой лиганда в структуре 1lmp:
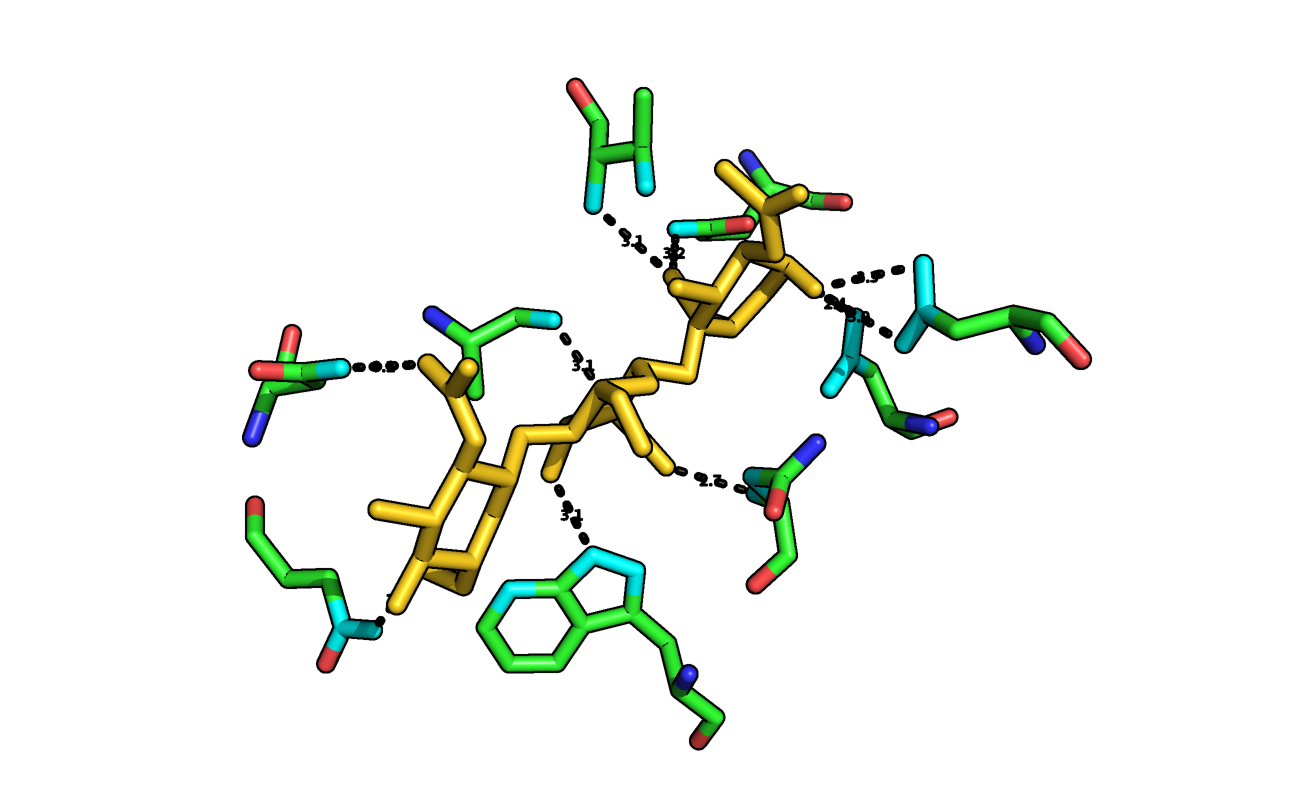
Запускаем скрипт:
mod9v7 lys_bpps1.py &
Привожу наложение получившихся пяти моделей. Видно, что они очень похожи! Правда, по непонятным мне причинам, у всех моделей есть большой неструктурированный участок на N-конце, а также большие петли. В структуре из PDB такого не было. На самом деле, я замечал, что если приходится иногда моделировать структуру по последовательности с помощью таких серверов, как LOMETS или I-TASSER, то на выходе можно получить неструктурированные части, причем иногда довольно протяженные.
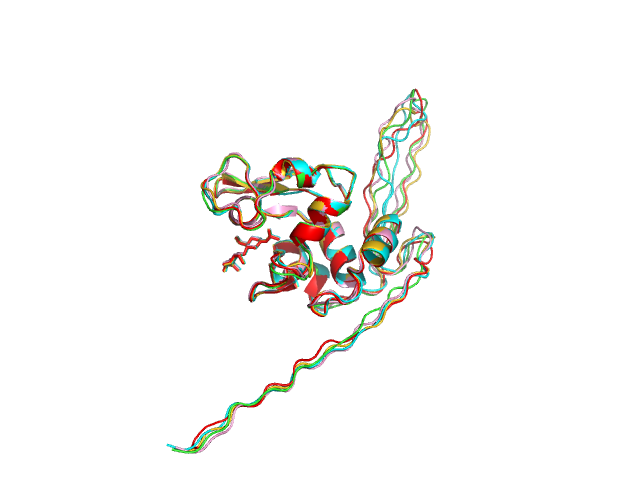
Проверим качество моделей и выберем лучшую. Инструменты для оценки качества структуры можно найти в веб интерфейсе WHATIF!
Первая модель. Omega average and std. deviation= 180.630 4.848 (должно быть среднее в районе +178) RMS Z-score for bond lengths: 1.007 (должно быть близко к 1) RMS Z-score for bond angles: 1.350 (должно быть близко к 1) Ramachandran Z-score : -1.372 (если положительное, то лучше среднего) Вторая модель. Omega average and std. deviation= 180.970 5.301 RMS Z-score for bond lengths: 1.004 RMS Z-score for bond angles: 1.323 Ramachandran Z-score : -1.490 Третья модель. Omega average and std. deviation= 180.740 5.504 RMS Z-score for bond lengths: 1.029 RMS Z-score for bond angles: 1.410 Ramachandran Z-score : -1.667 Четвертая модель. Omega average and std. deviation= 181.230 5.628 RMS Z-score for bond lengths: 1.004 RMS Z-score for bond angles: 1.393 Ramachandran Z-score : -1.431 Пятая модель. Omega average and std. deviation= 180.300 5.046 RMS Z-score for bond lengths: 1.000 RMS Z-score for bond angles: 1.336 Ramachandran Z-score : -1.522 Сравнение моделей (лучшие для соответствующих параметров). Omega average and std. deviation - четвертая RMS Z-score for bond lengths - пятая RMS Z-score for bond angles - вторая Ramachandran Z-score : - перваяОднозначно лучшей модели нет. Но самая выделяющаяся, на мой взгляд, это четвертая. Все файлы лежат здесь