
Я выбрал структуру 2MJW – NMR. В ней содержится 4 цепи (A,B,C,D) длиной 121, 94, 121 и 188 остатков соответственно. Какая-то необычная по размеру молекула белка для ЯМР. Все-таки, обычно длина белка, структура которого определяется этим методом не превышает 150-200 аминокислот… Однако строчка из PDB файла EXPDTA SOLUTION NMR доказывает, что структура определена с помощью ЯМР. В файле лежит 10 моделей!

Давайте попробуем посмотреть на водородную связь между 29 остатком аргинина (цепь C) и 32 остатком глутамата (тоже цепь C).
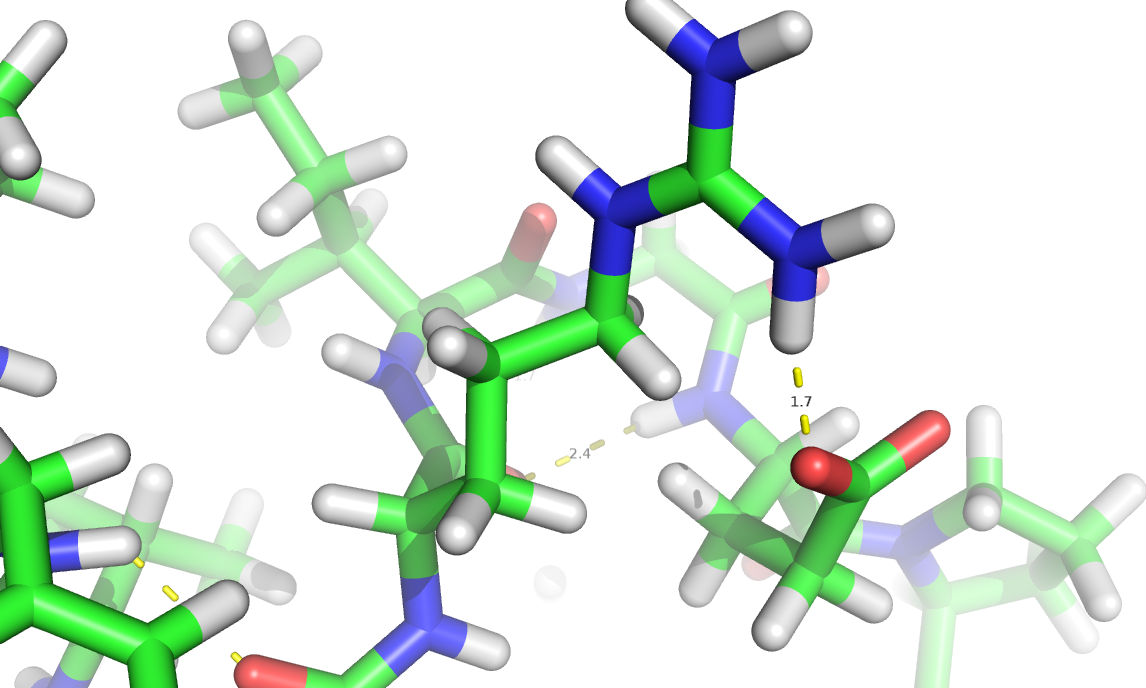
Обратите внимание, здесь мы видим атомы водорода (белые палочки). Это потому что мы смотрим на структуру, определенную с помощью ЯМР. А одна из особенностей этого метода – мы в состоянии увидеть атомы водорода. Это не удивительно, ведь по сути весь ЯМР зависит от этих атомов! Водородная связь изображена желтым пунктиром (1.7 – длина связи в данном случае). Это для 1 модели! Также стоит отметить, что водородная связь присутствует между атомами NH1 (аргинин 29) и OE2 (глутамат 32).
Посмотрев на остальные модели, я, с удивлением, обнаружил, что во всех остальных моделях, кроме 1 и 3, водородная связь образована между NH1 аргинина 29 и OE1 глутамата 32. Но самый особый случай (выделяющийся из всех остальных) можно увидеть на 6 модели. Привожу ее изображение.
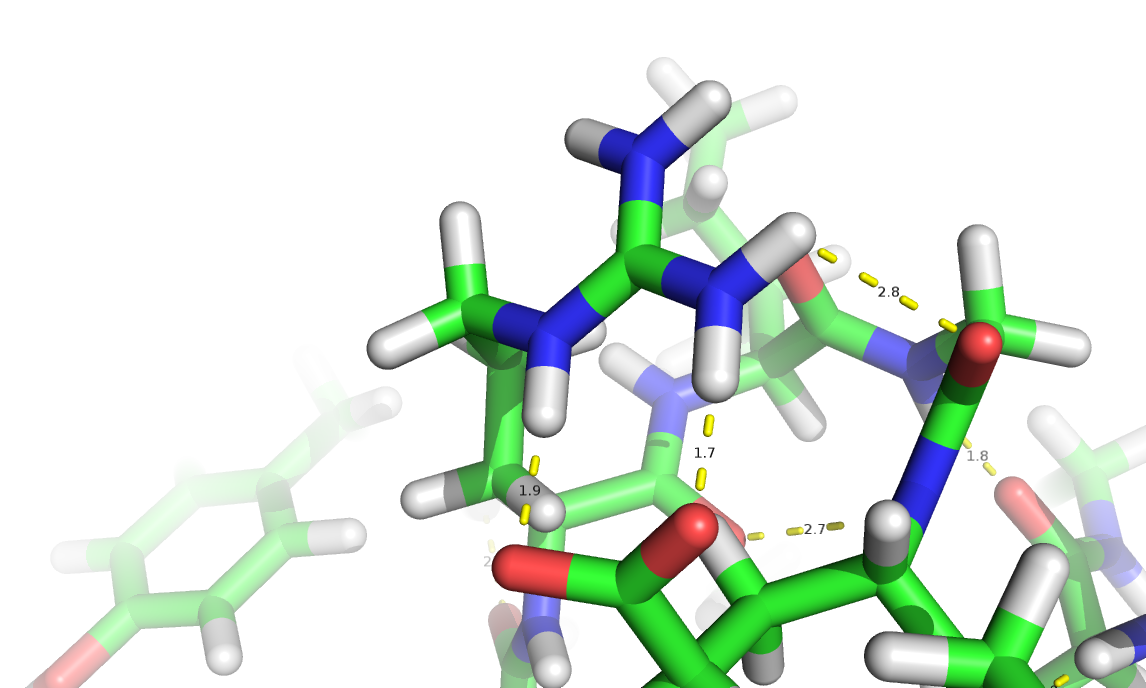
На этой картинке видно, что 29 остаток аргинина (да и остальные остатки) повернут таким образом, что он образует не одну водородную связь (как во всех моделях), а целых 3! Водородные связи образуют между собой следующие атомы: ARG29 NE – GLU32 OE2, ARG29 NH2 – GLU32 OE1 и ARG29 NE – GLY31 O (цепь C). Тут стоит отметить также такую особенность – во всех остальных моделях в образовании единственной водородной связи со стороны аргинина участвует атом NH1. Здесь же этот атом вообще повернут в другую сторону, и связь образует атом NH2. Вообще такой поворот (как, наверное, поворот и других остатков в модели) и привел к этим 3 водородным связям, вместо одной.
Я выбрал структуру 4CAZ – внизу представлена картинка асимметрической единицы (2 цепи).

А на рисунке ниже представлены 4 цепи - биологическая единица. Разные асимметрические единицы покрашены разными цветами!

Теперь перейдем к водородным связям между остатками из разных копий. Я нашел достаточно много таких связей и в качестве примера привожу одну из таких на рисунке ниже.
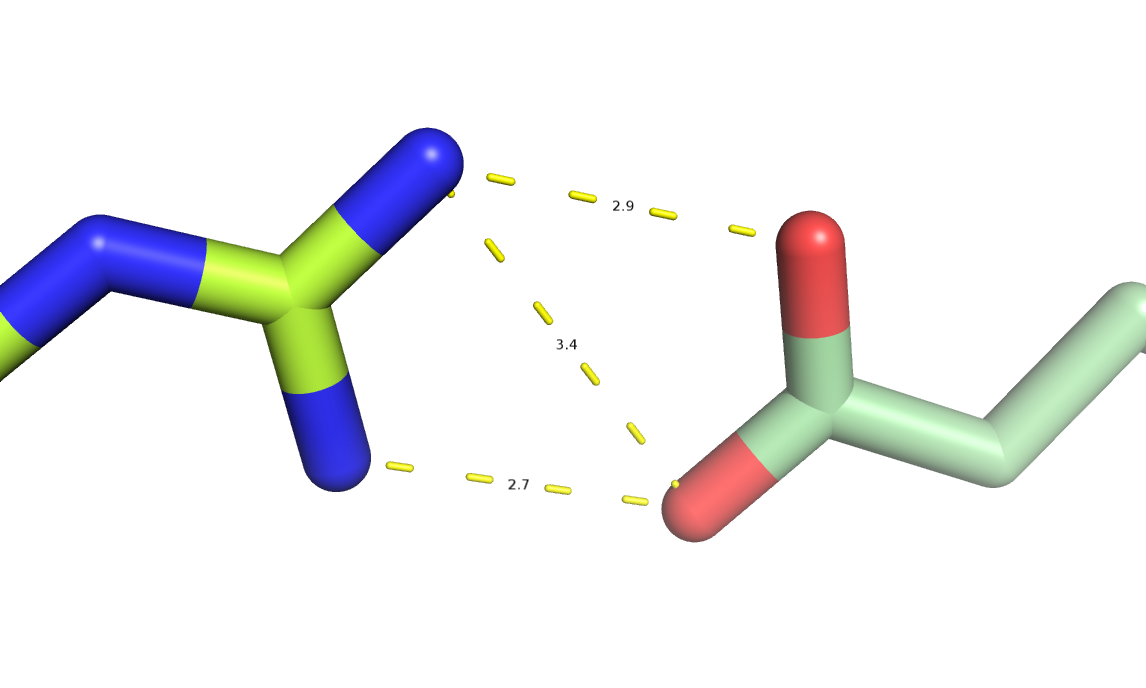
Справа на рисунке представлен глутамат цепи В одной асимметрической единицы, а слева – аргинин цепи В другой асимметрической единицы. Стоит отметить, что хотя тут и изображены 3 водородные связи, скорее всего, их 2, третья длиной 3.4 вряд ли такая уж настоящая водородная связь.
Я заинтересовался, что не так с этой структурой. Привожу ее изображение.
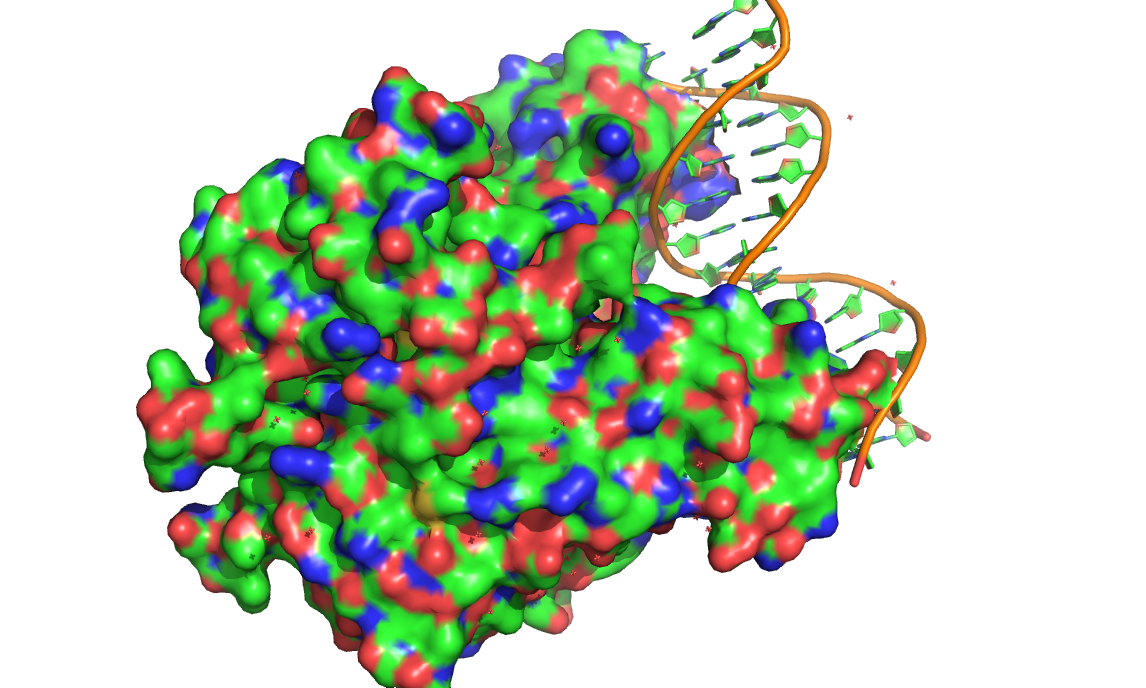
С виду никакой ошибки вроде бы нет. Может быть, что-то не так с pdb файлом? И на самом деле, если открыть этот файл и посмотреть на атомы цепей ДНК, то все они имеют occupancy 0.5! Самое интересное, в файле лежит две цепочки ДНК (как и должно быть) – цепь N и цепь M. И все атомы в этих цепях имеют occupancy 0.5. Но самое главное, что если в PyMol попробовать выделить каждую из этих цепочек по отдельности, то ничего не выйдет. Если вы забьете в командной строке следующую команду - select M, chain M – то PyMol выделит вам всю структуру ДНК! То есть, обе цепочки! Он напишет - selection "M" defined with 1228 atoms.
Давайте теперь попробуем выделить только цепочку N. И опять та же ситуация! PyMol выдаст - selection "N" defined with 1212 atoms. И окажется выделена вся структура ДНК. Причем, обратите внимание, обе команды выделения выделяют разное число атомов! Почему так, я не знаю, как объяснить...
Почему получается такая странная картина? Структура определена с разрешением в 2.5 ангстрем. Что не очень хорошо, на самом деле. На таком разрешении тяжело определить некие тонкости с occupancy (это было доказано в предыдущем практикуме), все стараются отразить в температурном факторе. И действительно, если посмотреть на температурные факторы тех странных атомов остатков ДНК (цепь М, у цепи N – тоже плохо, но не так сильно), то видно, что их температурные факторы зашкаливают. Как можно это интерпретировать (странное выделение и occupancy)? Вот мы видим на структуре две цепочки. При выборе цепи M (как и N) выделяются сразу две. Мне кажется, что в одной части ячеек (половине) цепь M находится в одном положении (когда в другом – цепь N), а в другой части – на месте цепи N (в то время, как цепь N занимает место M). Вот и получается такая странность. Конечно, я могу ошибаться.