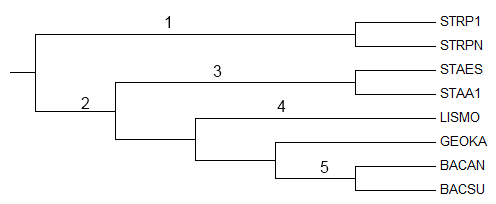

На изображенном дереве можно видеть бактерии, принадлежащие двум принципиально разным таксонам. Причём, если все они принадлежат таксону Firmicutes (и Bacilli), то ветвь под цифрой 1 выделяет таксон Lactobacillales (STRP1, STRPN), а ветвь 2 - таксон Bacillales. Если углубляться дальше, то можно заметить дальнейшее разделение по таксонам. Например, та же ветка 3 выделяет таксон Staphylococcaceae (в таксоне Bacillales). Ветка 4 указывает на Listeriaceae (также в Bacillales). Ветка 5 - на таксон Bacillaceae.
Я выбрал шаперонин с мнемоникой HSLO, а для получения выравнивания я пользовался программой Jalview.
Полная таксономия выбранных организмов:
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Bacillus anthracis | BACAN | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Bacillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Listeria monocytogenes | LISMO | Firmicutes | Bacilli | Bacillales | Listeriaceae |
| Staphylococcus aureus | STAA1 | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
| Staphylococcus epidermidis | STAES | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
| Streptococcus pneumoniae | STRPN | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
| Streptococcus pyogenes | STRP1 | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
С помощью программы Jalview я получил 4 разных дерева. Исходное и правильное дерево представлено выше.
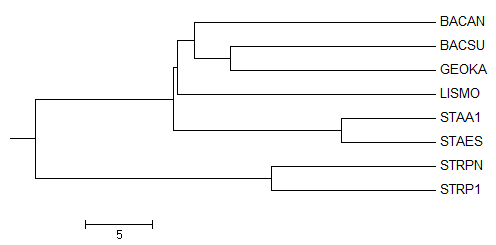
Первое дерево, полученное с помощью Calculate => Calculate Tree => Average Distance Using % identity. Видно, что оно очень похоже на правильное дерево, но есть принципиальная разница: организмы с мнемоникой GEOKA и BACSU помещены в один узел. С чем это может быть связано, не очень понятно, так как BACAN и BACSU явно имеют более близкое сродство друг с другом, чем те же GEOKA и BACSU. Таким образом, правильное дерево не содержит ветви {BACSU,GEOKA} против {BACAN, LISMO, STAA1, STAES, STRPN, STRP1}.
В свою очередь правильное дерево имеет все остальные ветви, которые имеет это дерево.
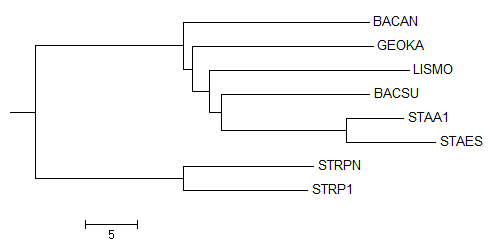
Первое дерево, полученное с помощью Calculate => Calculate Tree => Neighbour Joining Using % identity. В этом дереве больше тех ветвей, которые отсутствуют в правильном, причём разница тут сразу бросается в глаза (в отличии от первого). Ветки {BACAN} против {GEOKA ,BACSU, LISMO, STAA1, STAES, STRPN, STRP1}, {BACAN, GEOKA} против {BACSU, LISMO, STAA1, STAES, STRPN, STRP1} и {BACAN, GEOKA, LISMO} против {BACSU, STAA1, STAES, STRPN, STRP1} отсутствуют в правильном дереве.
Правильное дерево имеет ветви {BACAN, BACSU} против {GEOKA, LISMO, STAA1, STAES, STRPN, STRP1}, {BACSU, BACAN, GEOKA} против {LISMO, STAA1, STAES, STRPN, STRP1}, которых нет в этом дереве.
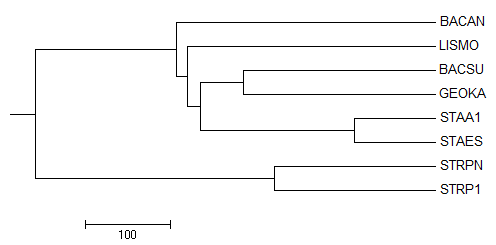
В этом дереве в отличии от правильного присутствуют следующие ветви: {BACAN} против {GEOKA ,BACSU, LISMO, STAA1, STAES, STRPN, STRP1}, {BACAN, LISMO, BACSU} против {GEOKA, STAA1, STAES, STRPN, STRP1} и {BACAN, LISMO, BACSU} против {STAA1, STAES, STRPN, STRP1}
В правильном дереве присутствуют ветви {BACAN, BACSU} против {GEOKA, LISMO, STAA1, STAES, STRPN, STRP1} и {BACSU, BACAN, GEOKA} против {LISMO, STAA1, STAES, STRPN, STRP1}, которых нет в этом дереве.
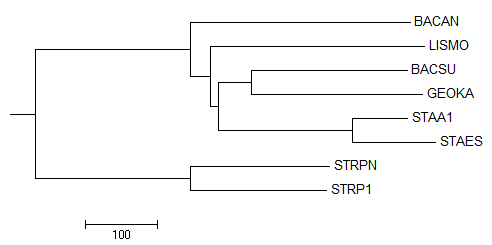
Это дерево получено 4 способом Calculate => Calculate Tree => Neighbour Joining Using BLOSUM62. В нём снова имеется узел с листьями BACSU и GEOKA. И снова бросается в глаза ветвь {BACAN} против {GEOKA ,BACSU, LISMO, STAA1, STAES, STRPN, STRP1}, которая отсутствует у правильного дерева.
И в также, в правильном дереве присутствуют ветви {BACSU, BACAN, GEOKA} против {LISMO, STAA1, STAES, STRPN, STRP1} и {BACAN, BACSU} против {GEOKA, LISMO, STAA1, STAES, STRPN, STRP1}, которых нет в этом дереве.
Таким образом, можно сделать вывод, что во всех четырёх деревьях и в правильном тоже имеются узлы STRPN, STRP1 и STAA, STAES. Почти у всех из 4 полученных деревьев (кроме 2) имеется узел GEOKA и BACSU. А родство BACAN и BACSU отсутствует у всех.
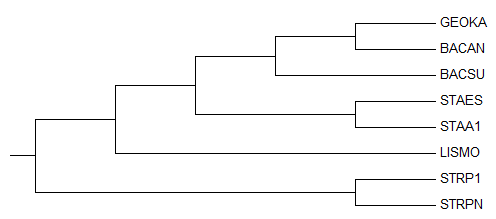
Здесь представлено дерево, которое построила MEGA. Оно похоже на правильное дерево и на дерево, построенное Jalview первым способом. Только, организм GEOKA опять оказался не на своём таксономическом месте. Он оказался больше сходен с BACAN, чем представитель рода Bacillus BACSU, что несколько странно. Быть может, я выбрал неудачный белок шаперонин-HSLO для бактерии BACSU, что и привело к подобным результатом, но в то же время это не объясняет, почему в одном случае GEOKA был больше схож с BACSU (деревья из Jalview), а в других - с BACAN (MEGA).
В правильном дереве присутствует ветвь {BACAN, BACSU} против {GEOKA, LISMO, STAA1, STAES, STRPN, STRP1}, которой нет в этом дереве, а отсутствует ветвь {BACAN, GEOKA} против {BACSU, LISMO, STAA1, STAES, STRPN, STRP1}, которая присутствует в этом дереве!
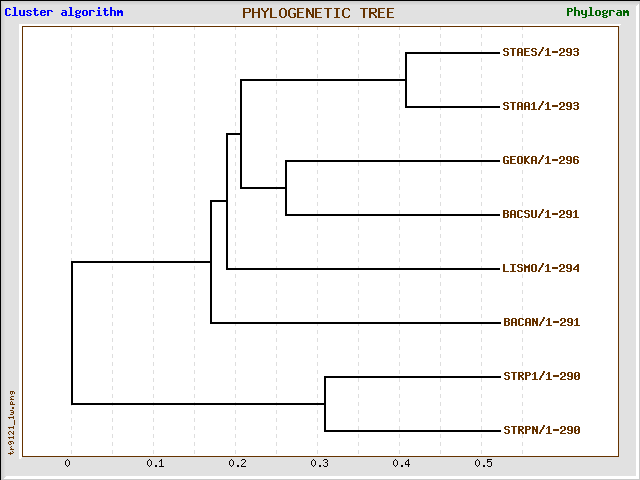
Ради интереса воспользовался сервером TreeTop, на мой взгляд, он не очень удобен для пользователя, который не так много имел дела с деревьями (неопытный, иначе говоря). Картинка, которая представлена выше, является результатом анализа выравнивания тех белков, с помощью которого я строил предыдущие деревья.