Пункт a.
С помощью BLAST на NCBI я получил 7 гомологов моего белка (GLSA1_BACSU). Привожу их список в виде ссылки:"список гомологов"
Пункт b.
Воспользовавшись программой JalView, я создал соответствующее множественное выравнивание. Построил его я следующим образом: WebService - Aligment - Tcoffee with Defaults
Пункт c.
Структура выравнивания.
| По столбцам выравнивания | По остаткам моего белка |
74-82 103-106 109-112 125-129 251-257 272-275 299-302 311-314 |
71-79 100-103 106-109 122-126 247-253 268-271 294-297 306-309 |
Участки с повышенной долей консервативных позиций я находил, настраивая conservation colour increment до 60 процентов. Соответственно, отбирал участки, где явно имеются функционально консервативные группы
В моём выравнивании имеются два участка с большим числом гэпов. Если считать, что это те самые участки, в которых выравнивание недостоверно, то вот их координаты: 5-16 столбцы выравнивания и 5-13 остатки моего белка, 287-297 столбцы выравнивания и 283-292 остатки моего белка.
Пункт d.
На самом деле, чаще всего среди функционально консервативных позиций у меня встречалась аминокислота глицин (G). Число этих групп равно 16. То есть, число столбцов в моём выравнивании, состоящих исключительно из глицина равно 16. Также часто встречаются группы NS (аспарагин и серин), KS (лизин и серин) и GI (глицин и изолейцин).

Пользуясь SRS я получил файл в fasta формате с последовательностями малых дельта-антигенов: Дельта-антигены.
Запрос, соответственно, выглядит следующим образом: (([swissprot-Taxonomy:Deltavirus*] & [swissprot-Description:delta*]) & [swissprot-Description:small*])
Далее я получил соответствующее выравнивание этих последовательностей с помощью программы muscle: Выравнивание
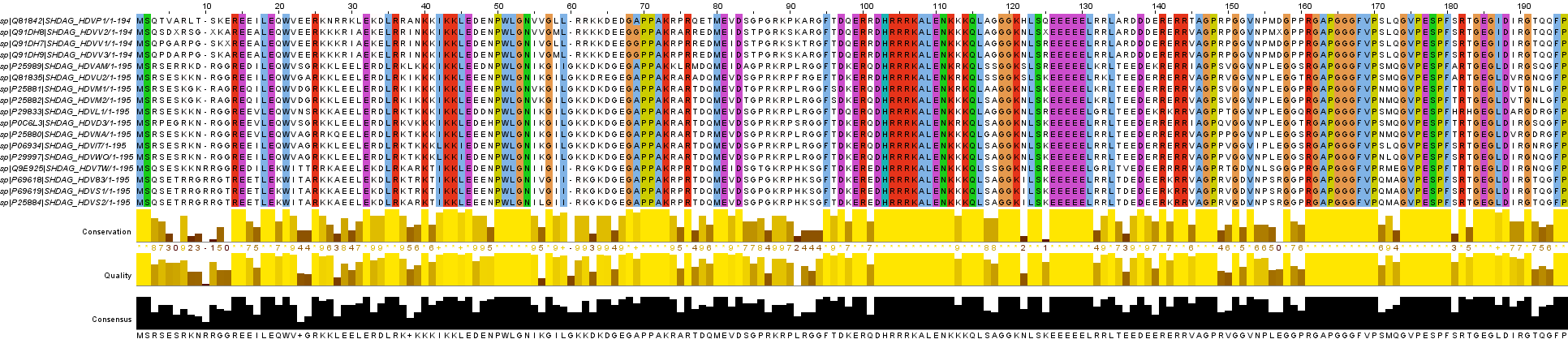
Раскраска произведена в Clustalx. Скорее всего, раскраска тут производится по принципу классификации аминокислот. Например, полярные отрицательно заряженные аспарагиновая и глутаминовая кислота здесь окрашены фиолетовым. Аминокислоты с гидрофобным радикалом окрашены синим. Ну и так далее.
А вообще хотелось бы отметить, что выравнивание нам показывает достаточно большое число функционально консервативных групп.
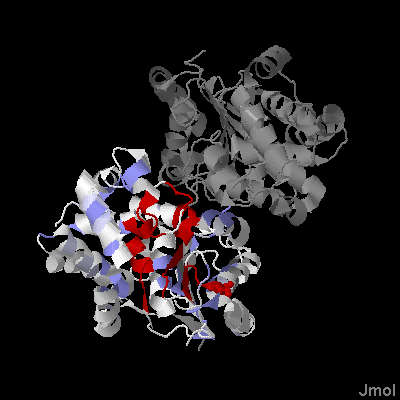
Привожу структуру своего белка. Выделена только одна цепь, что и понятно, поскольку имеем последовательность только для одной, а
вторая идентична ей.

Красным цветом я отметил те участки, координаты которых приведены в 1 задании. Они, на мой взгляд, наиболее важны в плане консервативности. Синим выделены консервативные остатки по BLOSUM62 (с учётом 60 процентной консервативности). Как видно, консервативные остатки спрятаны внутри глобулы. Выделенные красным группы расположены ближе к центру, и видимо принимают непосредственное участие в катализе, что и отражает их важность.
Вот такое выравнивание строит программа edialign: Edialign

А вот такое программа mafft (с помощью следующей команды: mafft "myproteins.fasta" > "pr.fasta"): Mafft
 Если рассмотреть консервативные участки, то можно увидеть, что они в основном одинаковы в обоих выравниваниях. Различие между ними имеется только в самом начале, где большое количество гэпов,
а также на участке выравнивания 290-300.
Но число гэпов одинаково.
Если рассмотреть консервативные участки, то можно увидеть, что они в основном одинаковы в обоих выравниваниях. Различие между ними имеется только в самом начале, где большое количество гэпов,
а также на участке выравнивания 290-300.
Но число гэпов одинаково.