Занятие 10-12. Эволюция белков митохондриальных рибосом
22 апреля - 12 мая
Поиск бактериальных гомологов белка S04 по нормированному профилю белков эукариотических митохондриальных рибосом
1. С помощью SRS были получены из UniProt последовательности белка S04 из митохондрий разных эукариот. Опции запроса:
A|||Ds: RT04
Description: S4,
Taxonomy: eukaryota
Общее число находок - 7. Найденные последовательности сохранены в формате FASTA в файле 7prots.fasta. Коды доступа сохранены в файле AC.txt, идентификаторы сохранены в файле ID.txt.
Выравнивание последовательностей получено с помощью алгоритма muscle: файл 7prots.html.
2. Все белки в выборке имеют следующий домен: PF01479 (для белка IMP3_HUMAN из митохондрий человека его положение 109-156 а.о.). Графическое представление:

Из PFAM получено выравнивание доменов с идентификатором PF01479 (всего 9352 последовательности), из полученного выравнивания извлечены варавнивания доменов всех найденных выше 7 белков. Стоит отметить, что для 2 записей выравнивание отсутствует. Выравнивание для 5 записей (файл GeneDoc.msf):

Сравнивая 2 выравнивая, можно сказать, что выравнивание белковых последовательностей не отличается наличием большого числа консервативных остатков относительно всей длины последовательности. Поэтому для получения лучшего результата, последовательности были выровнены "по профилю" (т.е в соответствии с доменным выравниванием).
3. С помощью программы muscle было построено выравнивание полных последовательностей найденных 7 белков: файл 7prots.html. С помощью программы pfm пакета PFTOOLS были рассчитаны веса последовательностей выборки: файл out.weighted.ali
4. При сравнении в GeneDoc выравниваний, полученный выше, можно сказать, что выравнивание с помощью алгоритма muscle менее удачное, в связи с чем была использована программа mafft для выравнивания полных последовательностей по профилю полученного выравнивания из PFAM: файл mafft.ali. Из этого файла были удалены все строки, относящиеся к доменному выравниванию, после чего файл GeneDoc.aa стал использоваться для построения профиля.
5. С помощью программы pfmake, используя взвешенное выравнивание, был построен профиль: файл myprofile.prf, затем этот профиль нормировался с использованием малой базы: файл myprofile.scaled.prf и большой базы: файл myprofile.scaled1.prf. Полученные профили не отличаются между собой. Профиль содержит информацию о частоте встречаемости каждого аминокислотного остатка в каждой позиции выравнивания. Далее, при поиске по профилю последовательности приписывается некий вес, определяющий насколько данная последовательность удовлетворяет профилю.При нормировании профиля он проверяется на случайно сгенерированной базе данный, что позволяет сделать его более избирательным.
Поиск гомологов по профилю, выбор порогового значения веса
6. C помощью программы pfsearch пакета PFTOOLS был произведен поиск гомологичных белков в обеих заданных группах бактерий: альфа- и гаммапротеобактерии с использованием нормированного варианта профиля. Каждый поиск был повторен 3 раза с разными значениями порога (варианты - 5.0, 10.0, 30.0). Выходные файлы:
Альфапротеобактерии
Порог 5.0: файл alpha5.search;
Порог 10.0: файл alpha10.search;
Порог 30.0: файл alpha30.search;
Гаммапротеобактерии
Порог 5.0: файл gamma5.search;
Порог 10.0: файл gamma10.search;
Порог 30.0: файл gamma30.search;
7. На основании полученных выше результатов была составлена таблица:
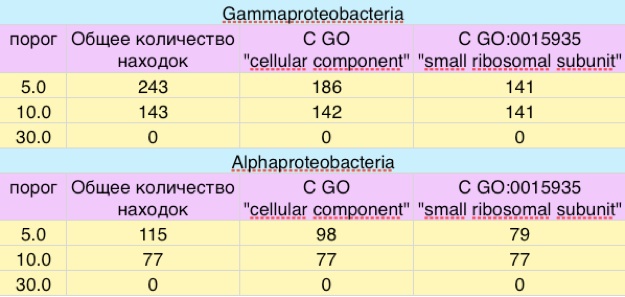
Анализ результатов
8. Значения весов находок по заданному профилю были импортированы в Excel: файл Gistogramm.xls. Также построины две гистограммы: распределение весов находок в альфа- и распределение весов находок в гаммапротеобактериях.
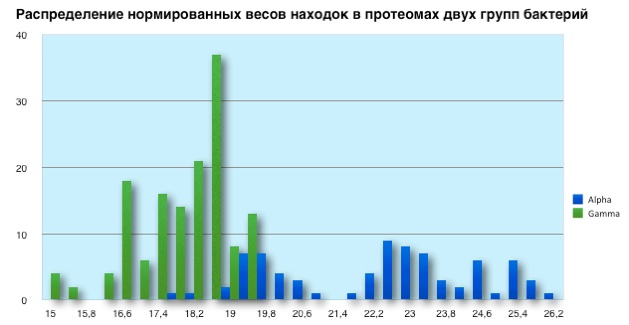
По полученной гистограмме видно, что каждый из двух классов протеобактерий разделяется предположительно на 3 кластера (по числу пиков). Медианы распределения: для альфапротеобактерий =22.39, для гамма =18.04.Это позволяет сделать промежуточный вывод о том, что последовательности из протеома альфапротеобактерий несколько ближе к исследуемому профилю.
9. Используя статистический пакет STADIA был проведен тест Вилкоксона:
КРИТЕРИИ СДВИГА (ПОЛОЖЕНИЯ). Файл:
Пропущн=66 66
Переменные: x1, x2
Вилкоксон=1,379E4, Z=-11,73, Значимость=0, степ.своб = 77,143
Гипотеза 1: <Есть различия между медианами выборок>
Исходя из того, что тест Вилкоксона показал существование различий между медианами,
для получения дополнительной информации была проведена описательная статистика:
ОПИСАТЕЛЬНАЯ СТАТИСТИКА. Файл:
Пропущн=66 66
Переменная Размер <---Диапазон---> Среднее---Ошибка Дисперс Ст.откл
альфапротеобактерии 77 17,63 26,18 22,16 0,2473 4,708 2,17
гаммапротеобактерии 143 14,4 19,39 17,7 0,09092 1,182 1,087
Переменная Медиана <--Квартили--> ДовИнтСр. <-ДовИнтДисп-> Ош.СтОткл
альфапротеобактерии 22,39 20,04 23,76 0,4861 3,936 4,708 0,6175
гаммапротеобактерии 18,04 17,04 18,54 0,1773 0,6912 0,6912 0,2647
Переменная Асимметр. Значим Эксцесс Значим
альфапротеобактерии -0,1383 0,3032 1,987 0,0323
гаммапротеобактерии -0,8135 0 3,356 0,1533
Таким образом, основываясь на результатах проведенного исследования, получили, что медиана альфапротеобактерий правее медианы гаммапротеобактерий. Это свидетельствует о том, что альфапротеобактерии ближе к рибосомальным белкам из митохондрий эукариот.
Филогенетический анализ
10. Была создана внешняя группа последовательностей рибосомальных белков из Firmicutes; для этого были использованы 6 последовательностей: файл out_group.txt. Последовательности белков из файла alpha10.search: файл alpha10_seqs.txt; последовательности белков из файла gamma10.search: файл gamma10_seqs.txt. При помощи программы muscle было построено множественное выравнивание последовательностей митохондриальных белков и белков из рибосом альфа-, гаммапротеобактерий и Firmicutes: файл all_seqs_align.html.
11. Было построено филогенетическое дерево методом максимального правдоподобия и получено его изображение с помощью TreeView:
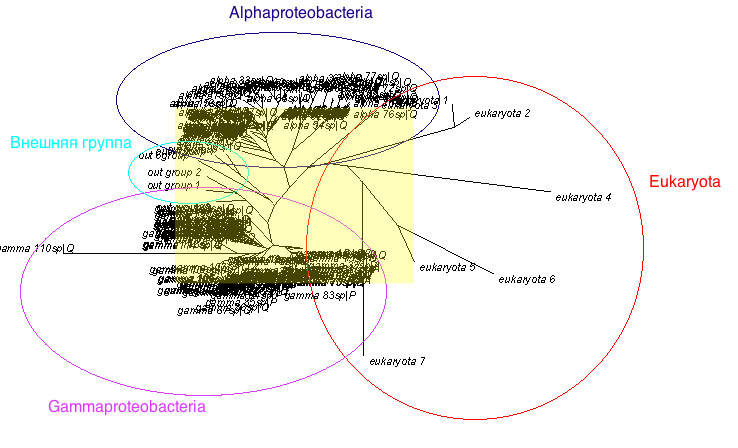
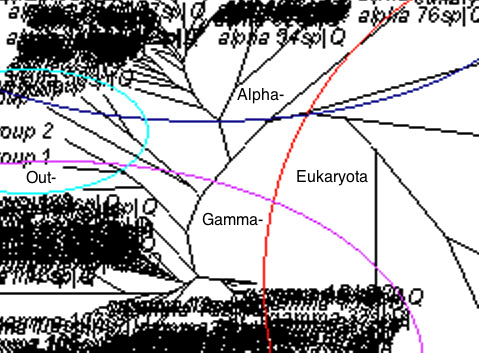
12. С помощью команды phylip protdist определелили попарные эволюционные расстояния по JTT и построили гистограмму распределения попарных расстояний между митохондриальными белками и белками из альфа- и гаммапротеобактерий: файл rasst_itog-1.xls,
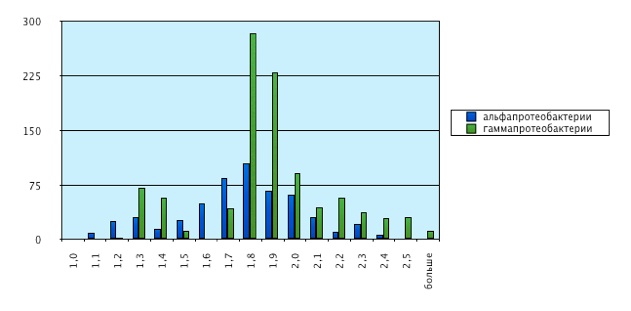
Медиана распределения количества последовательностей с определенным эволюционным расстоянием между ними по расстоянию для альфапротеобактерий лежит левее, чем для гаммапротеобактерий. То есть в среднем попарное эволюционное расстояние между митохондриями и гаммапротеобактериями больше, чем между митохондриями и альфапротеобактериями. А это, в свою очередь, говорит о том, что эволюционно митохондрии ближе альфапротеобактериям.
Резюме
В ходе работы по различным критериям сравнивались последовательности митохондриального рибосомального белка эукариот S04 с последовательностями рибосомальных белков альфа- и гаммапротеобактерий. Распределение нормированных весов находок в протеомах двух групп бактерий и тест Вилкоксона, а также филогенетический анализ, показали эволюционную близость белка S04 митохондрий белкам альфапротеобактерий. Поэтому можно сделать вывод о том, что происхождение митохондрий эукариот от альфапротеобактерий более вероятно, чем от гаммапротеобактерий.