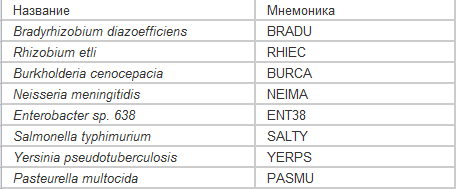
Практикум 4. Реконструкция филогении по нуклеотидным последовательностям. Паралоги.Задание 1.Построение дерева по нуклеотидным последовательностямВозьмем последовательности 16S рибосомальной РНК бактерий, которые я использовала ранее, из базы полных геномов NCBI. Нуклеотидные последовательности были обнаружены в файлах .frn формата.
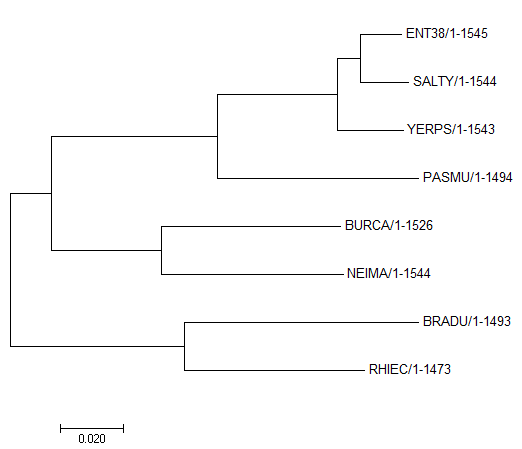
Рис1.Таблица с отобранными бактериямиПоследовательности были выравнены при помощи программы JalView Muscle. Выравнивание Дерево реконструировалось методом Neighbor-Joining.
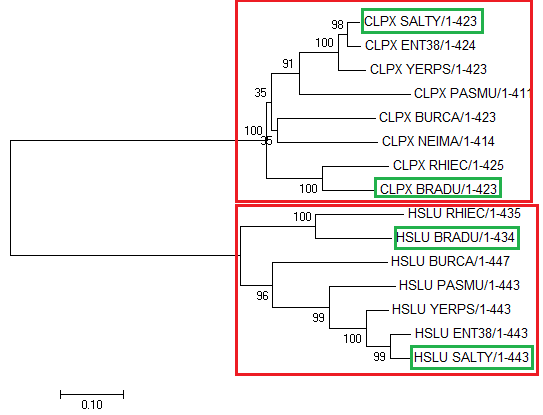
Дерево, реконструированное методом Neighbor-Joining в MEGA.Дерево полностью совпадает с правильным. Задание 2.Построение и анализ дерева, содержащего паралогиДля поиска гомологов белка CLPX_ECOLI был создан общий файл с протеомами 8 выбранных ранее бактерий, взятый из папки P:\y14\term4\Proteomes. Был произведен поиск при помощи программы blastp с выходной последовательностью CLPX_ECOLI по собранной базе протеомов. Порог по e-value 0.001. Выдача дала 27 находок.Из них были отобраны 15, содержащие группы CLPX и HSLU. Последовательности были выравнены при помощи программы JalView Muscle.
Дерево, реконструированное методом Neighbor-Joining в MEGA.Паралог - зеленый. Ортолог - красныйНеобходимо найти паралогов и ортологов.Паралоги - это гомологи из одного организма, где произошла дупликация генов, а ортологи - это гомологи из разных организмов, где разделение произошло из-за видообразования.Паралог отмечен на дереве зеленым цветом. Ортолог - красным |