Практикум 1. Построение и визуализация электронной плотности (ЭП). |
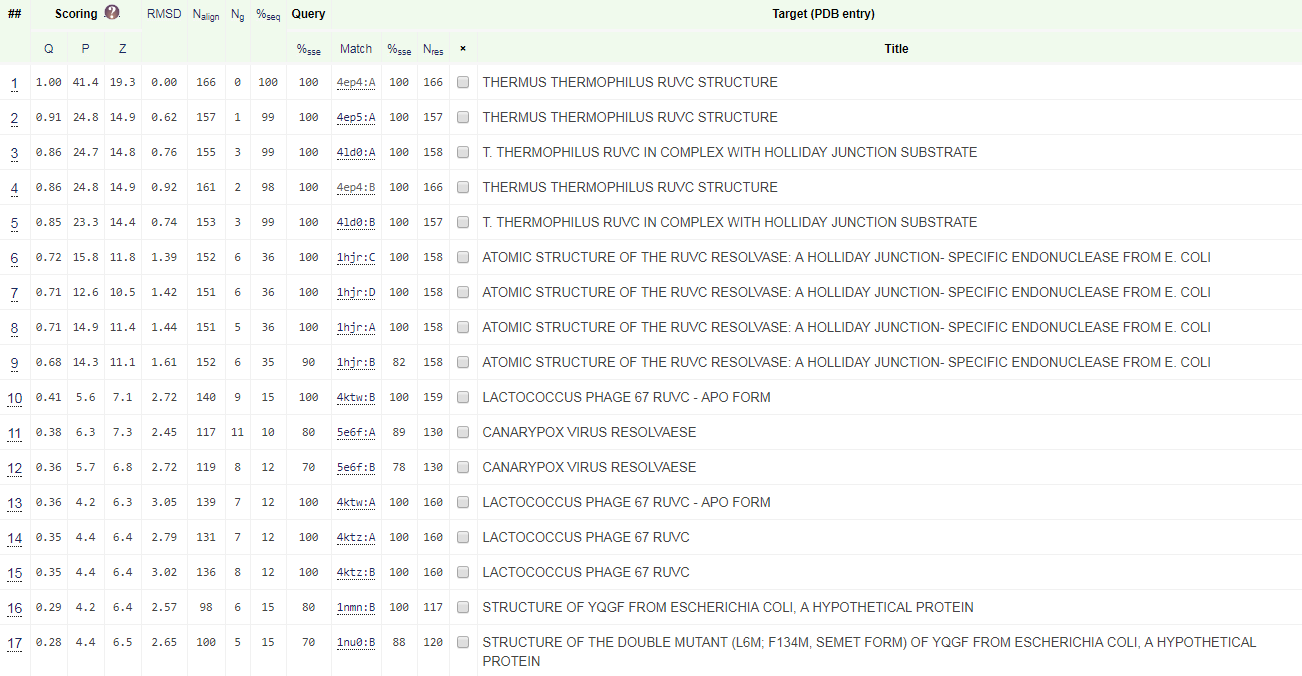
Задание 1. Постройте изображение электронной плотности вокруг остова полипептидной цепи.Для выполнения задания был выбрал белок из организма Alicycliphilus denitrificans K601 из первого семестра. В PDB банке код белка - 4EP4 (ссылка на белок на сайте PDB). С этого сайта я взяла EDS map (файл), структурные факторы (ссылка) и статью по результатам расшифровки (файл). Этот белок имеет много структурных гомологов и как видно на рисунке 1, есть находки с RMSD в пределе 0,8-3,0 ангстрем и N_align от 50 до 90% от числа аминокислотных остатков выбранного белка. Разрешение структуры - 1,28 ангстрем.
Рисунок 1. Результаты поиска гомологов к выбранному белку.Визуализация электронной плотности была реализована программой PyMOL и командой isomesh с уровенями подрезки 0-2.
Рисунок 2. Визуализация электронной плотности белка 4ep4. Уровень электронной плотности от 0 до 2. Остов полипептидной цепи - красный. |
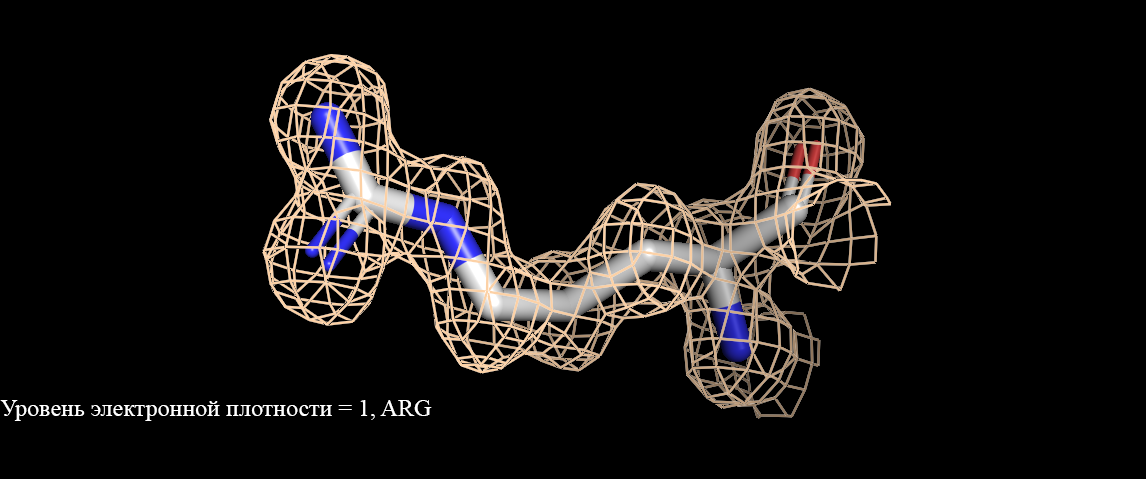
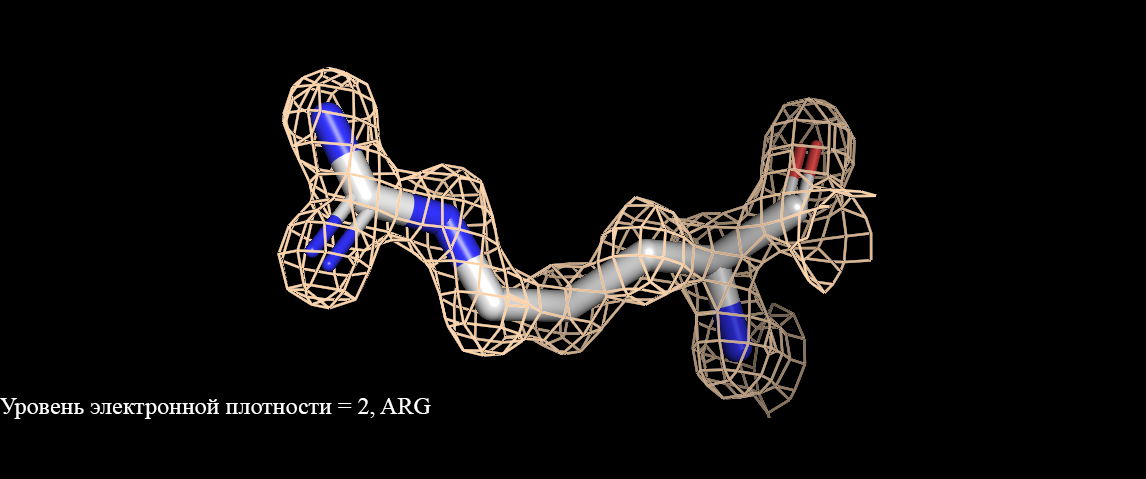
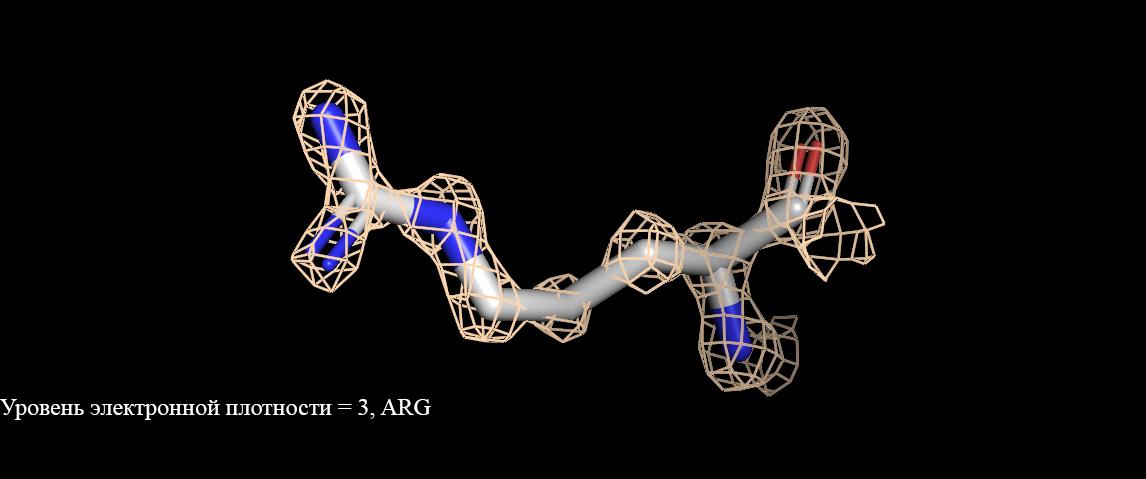
Задание 2. Построение и визуализация ЭП вокруг трех различных аминокислот.Мною были выбраны три аминокислоты: триптофан, гистидин и аргинин (рисунки 3-5).
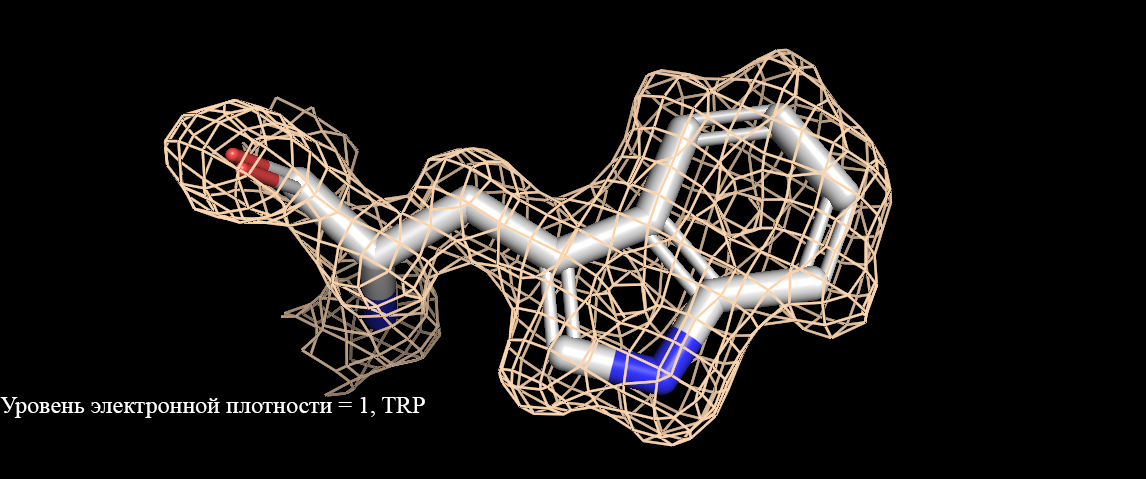
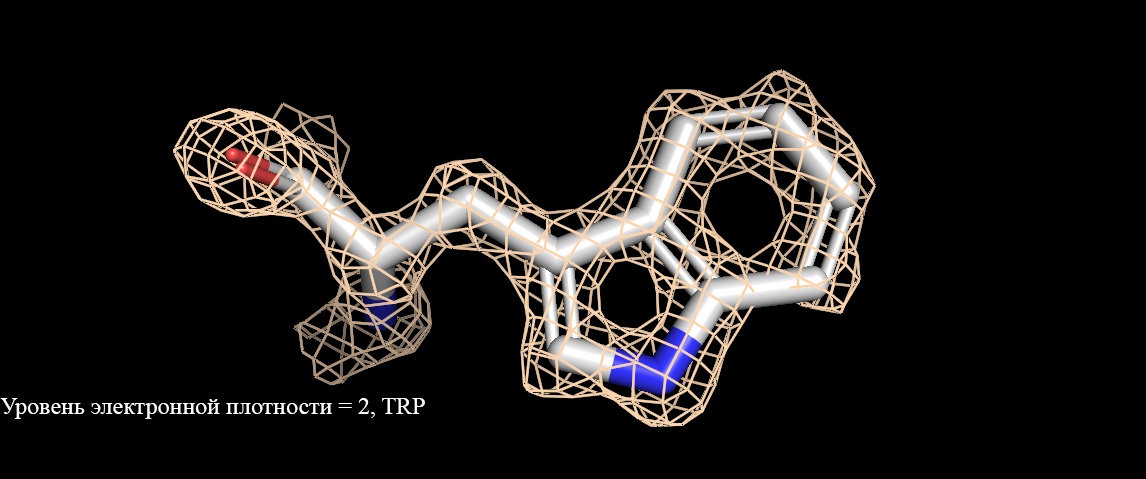
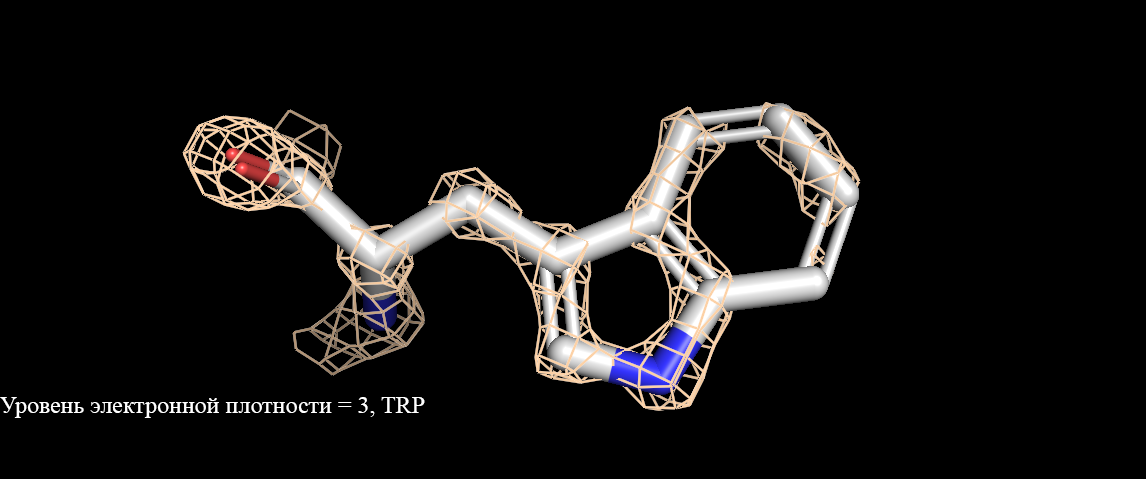
Рисунок 3. Визуализация электронной плотности триптофана(Trp). Уровень электронной плотности от 1 до 3.
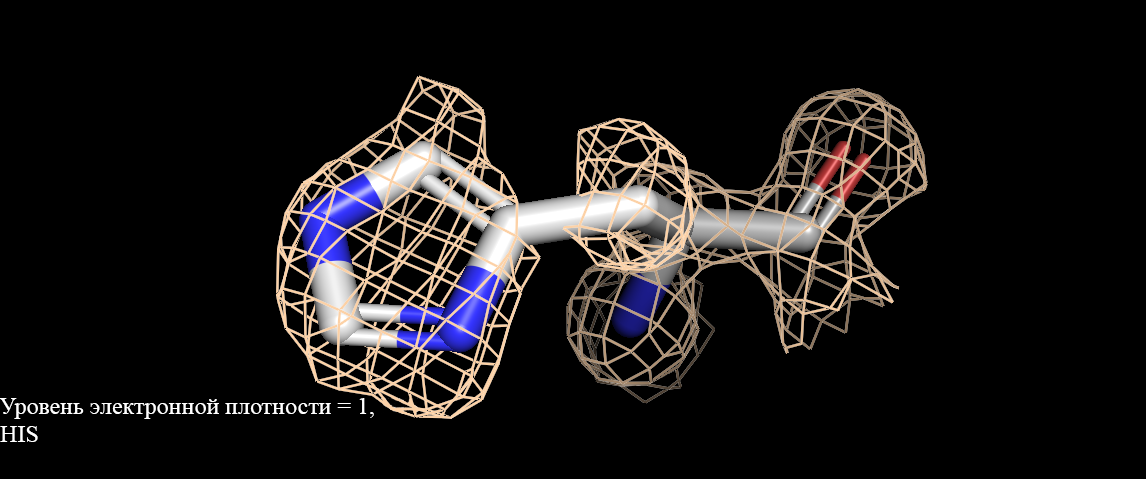
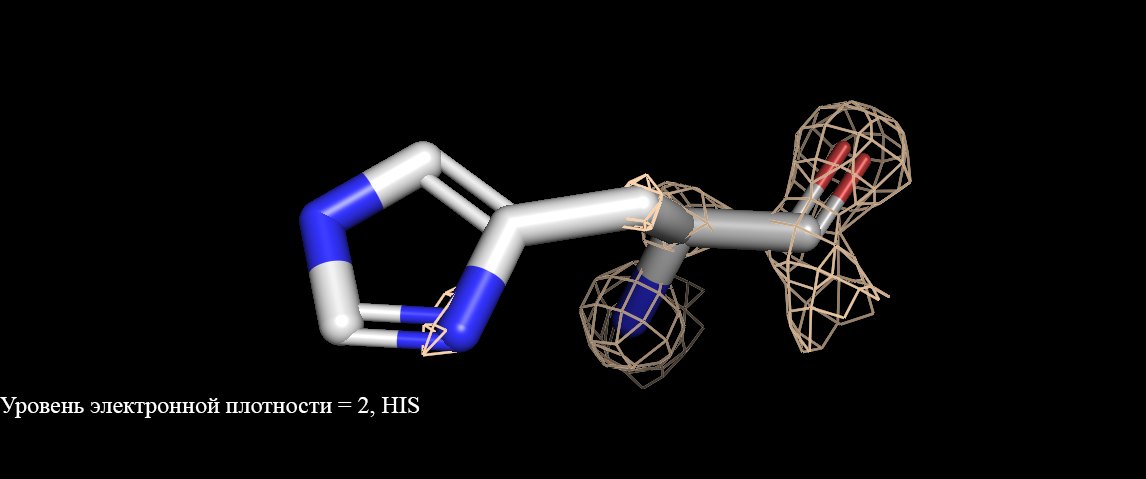
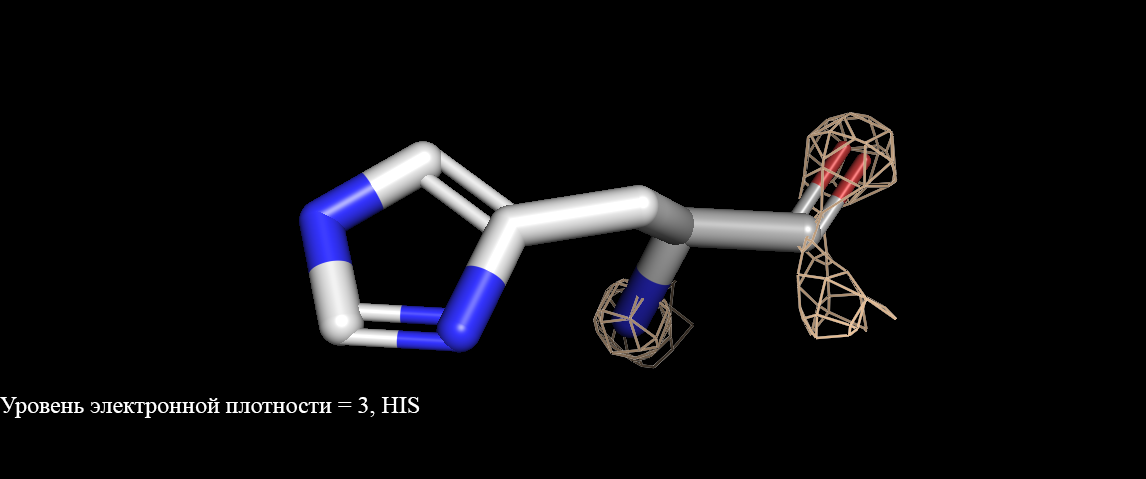
Рисунок 4. Визуализация электронной плотности гистидина(His). Уровень электронной плотности от 1 до 3.
Рисунок 5. Визуализация электронной плотности аргинина(Arg). Уровень электронной плотности от 1 до 3.Как видно, при уровне подрезки = 1 электронная плотность полностью окружает аминокислоты. При увеличении уровня подрезки уже не все атомы окружены (рисунок 3, уровень ЭП 3). Интересно, что при уровне подрезки 2 у имидазольного кольца гистидина практически полностью отсутствует сетка электронной плотности (рисунок 4), в сравнении с уровнем подрезки 1. У двух других аминокислот такого не наблюдается. Разрешение структуры довольно высокое (1,28 ангстрем), но не все аминокислоты точно описываются данной картой электронной плотности. |