Занятие 8. Структура тРНК
-
#1 Общее описание
Мне был предложена для рассмотрения структура тРНК с идентефикатором записи PDB 1tra.
Данная РНК принадлежит организму Saccharomyces cerevisae (пекарные дрожжи). Структура не содержит ничего кроме одной петляющей цепочки РНК. Идентефикатор цепи 1 (name: TRNAPHE).
#2 Последовательность
Последовательность тРНК, молекула единственная поэтому нумерацияс 1 по 76:
G C G G A U U U A 2MG C U C A G
H2U H2U G G G A G A G C M2G C C
A G A OMC U OMG A A YG A PSU 5MC
U G G A G 7MG U C 5MC U G U G 5MU
PSU C G 1MA U C C A C A G A A U U
C G C A C C A
Модифицированные основания:
2MG 2N-METHYLGUANOSINE-5'-MONOPHOSPHATE g
H2U 5,6-DIHYDROURIDINE-5'-MONOPHOSPHATE u
M2G N2-DIMETHYLGUANOSINE-5'-MONOPHOSPHATE g
OMC O2'-METHYLYCYTIDINE-5'-MONOPHOSPHATE c
OMG O2'-METHYLGUANOSINE-5'-MONOPHOSPHATE g
YG WYBUTOSINE n
PSU PSEUDOURIDINE-5'-MONOPHOSPHATE u
5MC 5-METHYLCYTIDINE-5'-MONOPHOSPHATE C
7MG 7N-METHYL-8-HYDROGUANOSINE-5'-MONOPHOSPHATE g
5MU 5-METHYLURIDINE 5'-MONOPHOSPHATE u
1MA 6-HYDRO-1-METHYLADENOSINE-5'-MONOPHOSPHATE a
MG MAGNESIUM ION
#3 Структура
Stand 1 Stand 2
Helix 1
1 | A:...1_:[..G]G-----C[..C]:..72_:A
2 | A:...2_:[..C]C-----G[..G]:..71_:A
3 | A:...3_:[..G]G-----C[..C]:..70_:A
4 | A:...4_:[..G]G-*---U[..U]:..69_:A
5 | A:...5_:[..A]A-----U[..U]:..68_:A
6 | A:...6_:[..U]U-----A[..A]:..67_:A
7 | A:...7_:[..U]U-----A[..A]:..66_:A
Helix2
8 | A:..49_:[5MC]c-----G[..G]:..65_:A
9 | A:..50_:[..U]U-----A[..A]:..64_:A
10 | A:..51_:[..G]G-----C[..C]:..63_:A
11 | A:..52_:[..U]U-----A[..A]:..62_:A
12 | A:..53_:[..G]G-----C[..C]:..61_:A
13 | A:..54_:[5MU]u-**--a[1MA]:..58_:A
Helix3
15 | A:..36_:[..A]A-*---U[..U]:..33_:A
16 | A:..38_:[..A]A-*---c[OMC]:..32_:A
17 | A:..39_:[PSU]P-*---A[..A]:..31_:A
18 | A:..40_:[5MC]c-----G[..G]:..30_:A
19 | A:..41_:[..U]U-----A[..A]:..29_:A
20 | A:..42_:[..G]G-----C[..C]:..28_:A
21 | A:..43_:[..G]G-----C[..C]:..27_:A
22 | A:..44_:[..A]A-*---g[M2G]:..26_:A
Helix4
23 | A:..10_:[2MG]g-----C[..C]:..25_:A
24 | A:..11_:[..C]C-----G[..G]:..24_:A
25 | A:..12_:[..U]U-----A[..A]:..23_:A
26 | A:..13_:[..C]C-----G[..G]:..22_:A
define helix1 1-7:a, 66-72:a |
На данной иллюстрации можно пронаблюдать внеспиральное взаимодествие цитозина и гуанина. |
|
Здесь представлено взаимодействие не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований: водородная связь между 6м азотом аденозина 36 и 6м кислородом вибутозина 37.> |
Очень сложно визуально оценвать такую сложную молекулу и я воспользовался возможностями программы find_pair и analyze. В итоге все цепи пердставляют собой А-формы, закрученные в право.
Программа einverted нашла два самых длинных участка, определенных find_pair, спиралей.
: Score 16: 8/10 ( 80%) matches, 0 gaps
22 gagcgccaga 31
|| | |||||
48 ctggaggtct 39
: Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
Остальные совпадения в основном единичны, за счет системы весов они не проходят в результаты einverted хотя они и имеют место быть. В итоге программа находит только самые простые для анализа (тоесть достаточно длинные) спиральные участки, и менее точна чем find_pair который анализирует третичную структуру.
Для получения результата в этой прорамме пришлось уменьшить парметр Р до 5ти. Но даже это не приблизило выдачу к действительности. Я считаю что структура слишком сложно — участки пересечений чередуются, много совпаденйи по единичным парам нуклеотидов, и поэтому программно предсказать ее структуру не удается.
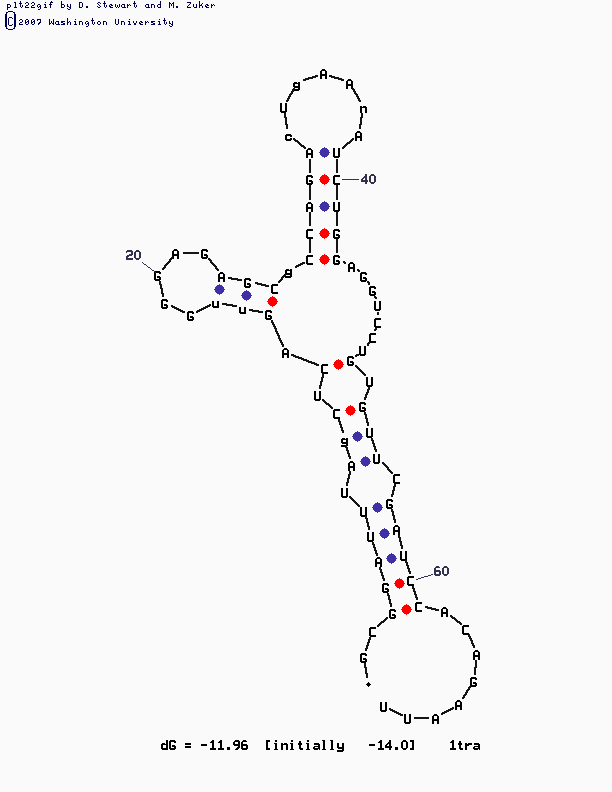
Результат не совсем соответвует реальному, но mfold тоже является программой с определеной степенью точности.