На страницу четвертого семестра
Классификация функций. Ферменты.
Код фермента: EC 3.6.1.1
- EC 3 hydrolases — гидролазы
- EC 3.6 Acting on acid anhydrides — гидролазы, функционирующие в кислотных ангидридах
- EC 3.6.1 In phosphorus-containing anhydrides — функционирующие в фосфо-содержащих ангидридах
- EC 3.6.1.1 inorganic diphosphatase — Неорганические дифосфатазы
Каждая следующая цифра все более конкретизирует функцию, проводя нас по дереву подклассов данного класса энзимов.
Использование средств Brenda для характеристики фермента 3.6.1.1
Всего существует с таким кодом: 486
Оптимум PH: 2.5 — 9.8
Данный фермент катализирует следующую реакцию:
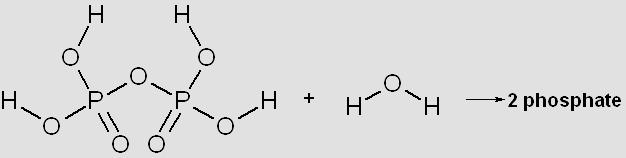
(diphosphate + H2O = 2 phosphate)
У данных ферментов бывают следующие ингибиторы:
- 1,1,1-azodimethyl diamide
- 2,3-Butanedione
- 2,4,6-Trinitrobenzenesulfonic acid

- 2-Phosphoglyceric acid
- 4-bromophenacyl bromide
- 4-Hydroxymercuribenzoate
- 7-Chloro-4-nitrobenzo-2-oxa-1,3-diazole
- ADP
- aminomethylenediphosphonate
- AMP
- Arsenite
- ATP
- Ba2+
- bisphosphonates
- Ca2+
- Ca2P2O7
- CaCl2
- Cd2+
- Co2+
- Cu2+
- Cyanate
- D-Glucose
- D-Glucose-6-phosphate
- Diazonium-1H-tetrazole
- Diethyldicarbonate
- Diethylpyrocarbonate
- Diphosphate
- EDTA
- F-
- Fe2+
- Fe3+
- Fluorescein 5'-isothiocyanate
- free divalent cations
- Fructose-1,6-bisphosphate
- Glutathione
- Guanidine HCl
- Guanidine nucleotides
- Hg2+
- hydroxymethylbisphosphonate
- I2
- Imidodiphosphate
- Iodoacetamide
- Iodoacetate
- KCN
- KF
- L-Malate
- Li+
- Maleic anhydride
- Mersalyl
- Mg2+
- MoO42-
- N,N'-diclohexylcarbodiimide
- N-Ethylmaleimide
- N-Phenylmaleimide
- Na+
- NaF
- NaN3
- O3PCHOHPO3
- O3PNHPO
- p-Chloromercuribenzenesulfonic acid
- p-Chloromercuribenzoate
- p-Chloromercuriphenyl sulfonate
- P2O74-
- Phenylglyoxal
- phenylmercuric acetate
- Phenylmethanesulfonyl fluoride
- Phosphonates
- risedronate
- Rose bengal
- Salts
- SDS
- Sodium cholate
- Succinic anhydride
- Triton X-100
- Zn2+
...и активаторы:
- Dithiothreitol
- K+
- KCl
- methylenebisphosphonate
По данным ресурса Brenda, с данным ферментом не ассоциированна ни одна запись о болезнях как человека, так и прочих живых существ.
Данные ферменты часто встречаются в олигомерной форме, у разных организмов могут быть и мономеры, и гексамеры:
Организмы с:
Мономерами:
- Dunaliella salina
- Sesamum indicum
- Sorghum vulgare
- Spinacia oleracea
Димерами:
- Dimer Bacillus subtilis
- Dimer Bos taurus
- Dimer Escherichia coli
- Dimer Geobacillus stearothermophilus
- Dimer Homo sapiens
- Dimer Oryctolagus cuniculus
- Dimer Rattus norvegicus
- Dimer Rhodopseudomonas palustris
- Dimer Saccharomyces cerevisiae
- Dimer Sus scrofa
- Dimer Vigna radiata
Тримерами:
- Bacillus subtilis
- Escherichia coli
- thermophilic bacterium PS-3
тетрамерами:
- Methanobacterium thermoautotrophicum
- Streptococcus faecalis
- Sulfolobus acidocaldarius
- Thiobacillus thiooxidans
гексамерами:
- Hexamer Escherichia coli
- Hexamer Helicobacter pylori
- Hexamer thermophilic bacterium PS-3
Вообще, у Escherichia coli показаны различные олигомеры, от мономера до гексамерной формы.
Сравнение ферментов с одинаковым кодом из эволюционно далеких организмов
Было найдено 4 белка из организмов Escherichia coli K-12, Methanococcus jannaschii, Homo sapiens. (Из человека — 3)
Запрос: "((([swissprot-ID:*_Ecoli*] | [swissprot-ID:*_Human*]) | [swissprot-ID:*_Metja*]) & ([swissprot-ECNumber:3.6.1.1*] ! ((((((((([swissprot-ECNumber:3.6.1.10*] | [swissprot-ECNumber:3.6.1.11*]) | [swissprot-ECNumber:3.6.1.12*]) | [swissprot-ECNumber:3.6.1.13*]) | [swissprot-ECNumber:3.6.1.14*]) | [swissprot-ECNumber:3.6.1.15*]) | [swissprot-ECNumber:3.6.1.16*]) | [swissprot-ECNumber:3.6.1.17*]) | [swissprot-ECNumber:3.6.1.18*]) | [swissprot-ECNumber:3.6.1.19*]))) "
Были получены данные о границах доменов:
| Организм | ID | AC | Начало домена | Конец домена |
| Homo sapiens | IPYR2_HUMAN | Q9H2U2 | 93 | 277 |
| Escherichia coli | IPYR_ECOLI | P0A7A9 | 16 | 174 |
| Homo sapiens | IPYR_HUMAN | Q15181 | 45 | 229 |
| Homo sapiens | Q6ZSW6_human | Q6ZSW6 | 2 | 65 |
Данные последовательности были получены с помощью машины SRS, далее с использованием программы needle
были получены значения Identity этих белков (скрипт 1), далее были получены выравнивания (скрипт 2).
Скрипт 1:
water IPYR2_HUMAN.fasta IPYR_ECOLI.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" > analyze.water
water IPYR2_HUMAN.fasta IPYR_HUMAN.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" >> analyze.water
water IPYR2_HUMAN.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" >> analyze.water
water IPYR_ECOLI.fasta IPYR_HUMAN.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" >> analyze.water
water IPYR_ECOLI.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" >> analyze.water
water IPYR_HUMAN.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 stdout | grep "# Identity:" >> analyze.water
Скрипт 2:
water IPYR2_HUMAN.fasta IPYR_ECOLI.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR2_HUMAN_IPYR_ECOLI.msf
water IPYR2_HUMAN.fasta IPYR_HUMAN.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR2_HUMAN_IPYR_HUMAN.msf
water IPYR2_HUMAN.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR2_HUMAN_Q6ZSW6_HUMAN.msf
water IPYR_ECOLI.fasta IPYR_HUMAN.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR_ECOLI_IPYR_HUMAN.msf
water IPYR_ECOLI.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR_ECOLI_Q6ZSW6_HUMAN.msf
water IPYR_HUMAN.fasta Q6ZSW6_HUMAN.fasta -gapopen 10 -gapextend 0.5 -aformat msf IPYR_HUMAN_Q6ZSW6_HUMAN.msf
Попарные значения Identity:
| Пара | значение Identity |
| IPYR2_HUMAN vs IPYR_ECOLI | 26.8% |
| IPYR2_HUMAN vs IPYR_HUMAN | 74.1% |
| IPYR2_HUMAN vs Q6ZSW6_HUMAN | 73.8% |
| IPYR_ECOLI vs IPYR_HUMAN | 29.7% |
| IPYR_ECOLI vs Q6ZSW6_HUMAN | 34.0% |
| IPYR_HUMAN vs Q6ZSW6_HUMAN | 100.0% |
Последняя цифра не удивляет, так как Q6ZSW6 — клон.
©
Галкин Иван, 2006
