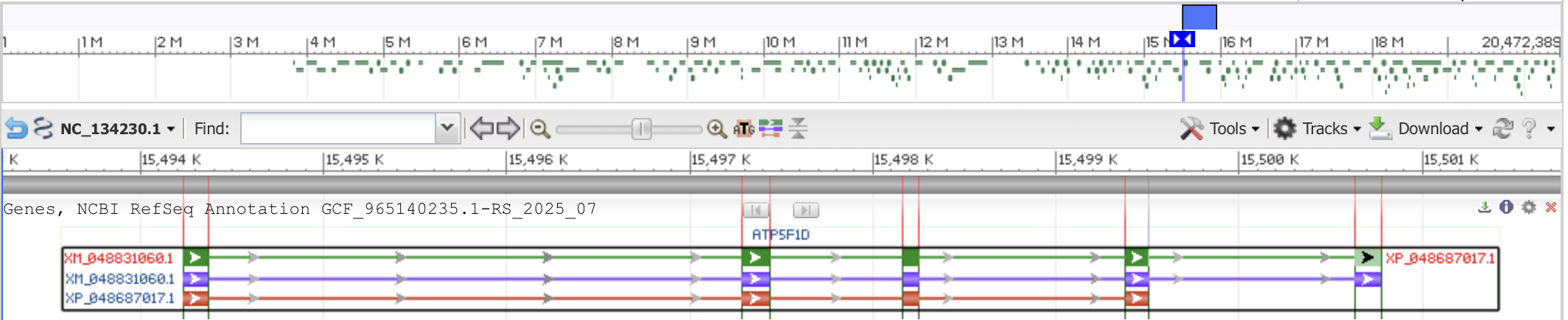
В таблице белковых последовательностей для сборки генома головастой морской черепахи (лат. Caretta caretta) была найдена одна запись, соответствующая δ-субъединице АТФ-синтазы (id:XP_048687017.1). Далее был найден идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок (NC_134230)
Файл с последовательностью кодирующей белок части.

Выбранный ранее организм вторичноротый, поэтому для этого задания возьмем кого-то из первичноротых. Было выбрано семейство пчёл (Apoidea).
Blastn более чувствителен для поиска дивергировавших нуклеотидных последовательностей по сравнению с megablast, поэтому будем использовать его. Со стандартными параметрами выдавало 0 находок,поэтому попробуем изменить параметры. Найдем все возможные совпадения, даже если они скорее всего случайные - просто посмотрим, есть ли хоть что-то. В итоге, даже при либеральных настройках (E-value=10, word size = 7) blastn нашел только 2 потенциальных гомолога с очень низкой идентичностью (40-50% и <40%).
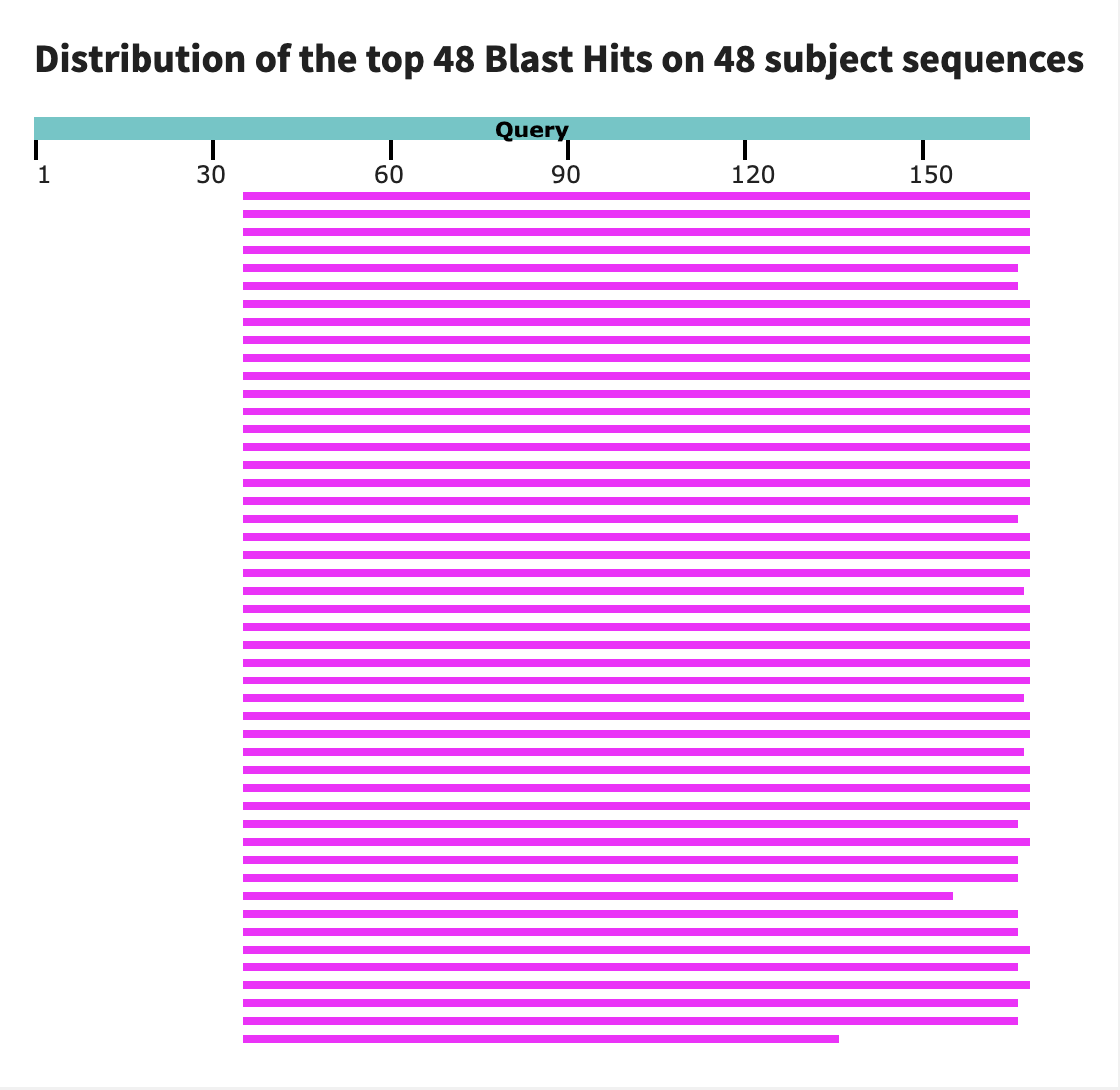
В то же время tblastn обнаружил 38 высоконадежных гомологов с типичной для консервативных белков идентичностью 60-80% и стандартными параметрами (word size = 5). Таким образом, для последовательности белок-кодирующей области δ-субъединицы АТФ-синтазы были найдены ей гомологичные во всех 38 сборках геномов пчёл из refseq_genomes. Из этого можно сделать вывод, что в последовательности δ-субъединицы АТФ синтазы есть продолжительные консервативные участки, сохранившиеся у представителей далёких друг от друга таксонов.
Индексируем геном
makeblastdb -in /Users/kseniagapon/Desktop/GCF_965140235.1_rCarCar1.hap1_genomic.fna -dbtype nucl -out carreta_genome
Программа makeblastdb принимает файл с последовательностями в fasta-формате (параметр "-in") и создаёт локальную базу данных. Параметр "-dbtype" указывает на тип последовательности – в случае нуклеотидной последовательности нужно указать "-dbtype nucl".
Для поиска гомологов бактериальных рРНК в геноме эукариота был выбран алгоритм blastn, поскольку алгоритмы, работающие на уровне аминокислот (tblastn/tblastx), неприменимы для поиска рибосомальных РНК, а megablast оптимизирован для поиска высокоподобных последовательностей. В файлы 16s_rrna.fasta и 23s_rrna.fasta были записаны последовательности, соответственно, 16S и 23S рРНК E. coli.
Команды для поиска:
blastn -task blastn -query /Users/kseniagapon/Desktop/23s_rrna.fasta -db carreta_genome -out /Users/kseniagapon/Desktop/blast_23s_results.txt -outfmt 7
blastn -task blastn -query /Users/kseniagapon/Desktop/16s_rrna.fasta -db carreta_genome -out /Users/kseniagapon/Desktop/blast_16s_results.txt -outfmt 7
-task: алгоритм, который используется при поиске.
-query: имя файла с последовательностью запроса
-db: имя базы данных
-outfmt: формат выходного файла. Значение по умолчанию — 0. Популярно также значение 7 — таблица с комментариями
Файл с результатами BLAST для 16s рРНК.
Файл с результатами BLAST для 23s рРНК.
При выравнивании 16S рРНК E. coli было обнаружено 20 совпадений. из которых можно выделить 11 потенциальных гомологов, однако обращая внимание на e-value находки можно выделить 8 гомологов.
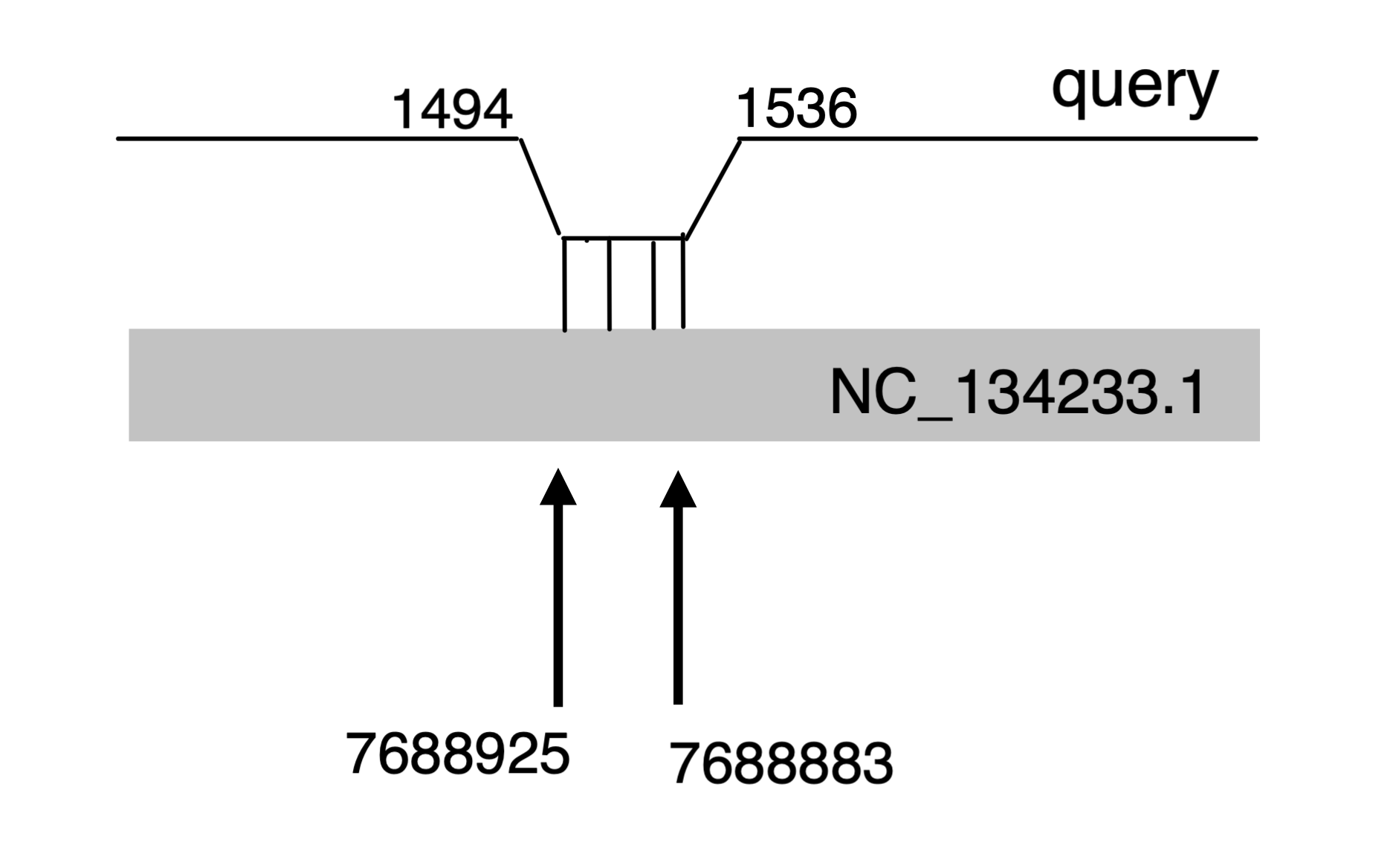
При выравнивании 23S рРНК E. coli было обнаружено 74 совпадения, из которых E-value менее 10^-2 имело 58. На NC_134233.1 среди сильных хитов видно 8 групп координат, но часть из них, как пара 7825850–7825759 и 7825558–7825459, сливаются в один локус. После такого слияния на NC_134233.1 получается порядка 4–5 гомологов, а с добавлением сильных хитов на других NC_... итоговое число гомологов 23S в геноме оказывается около 10–12 при пороге e‑value < 10^-2
Было выбрано две последовательности геномов Mycoplasma leachii и Mycoplasma mycoides
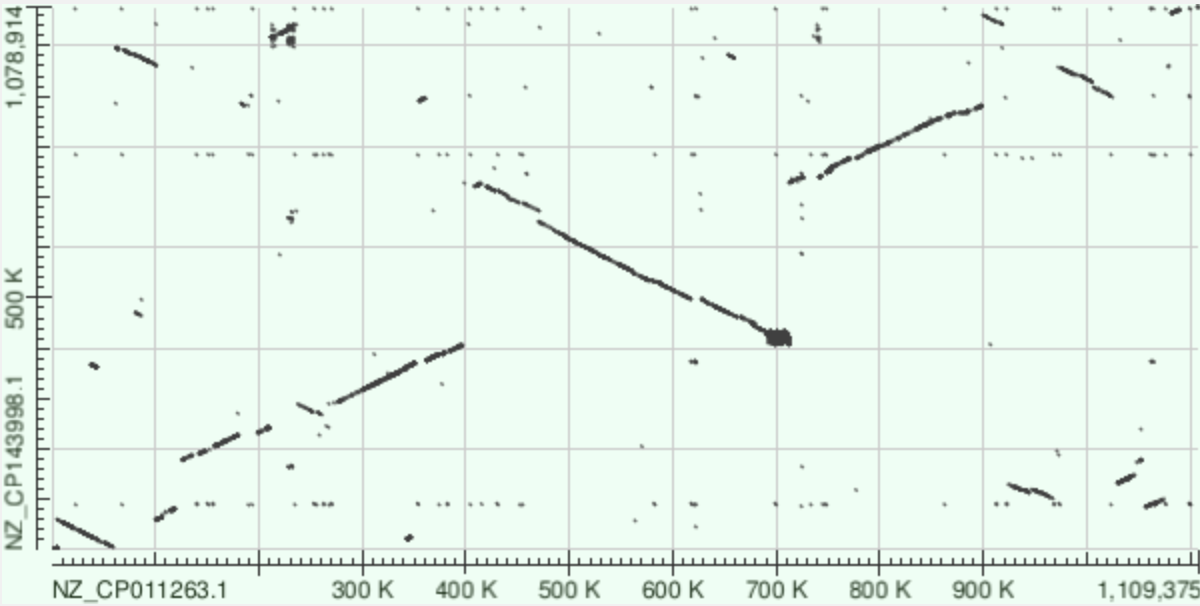
Карта выделяет крупные, высококонсервативные области нуклеотидного сходства между двумя геномами. Самая длинная диагональная линия указывает на основной синтенный (упорядоченно консервативный) регион. Разрыв и смещение основной диагонали указывают на инверсию.

Отображает больше точек и более коротких линий, чем megablast, что указывает на большее количество локальных сходств, возможно, включая регионы с более низкой общей идентичностью последовательностей.
Подобно megablast, подтверждает основные области синтенности и перестройки. Увеличенная плотность совпадений по сравнению с megablast предполагает, что стандартный BLASTN обнаруживает больше гомологичных областей, включая те, которые короче или немного менее консервативны, чем найденные megablast.
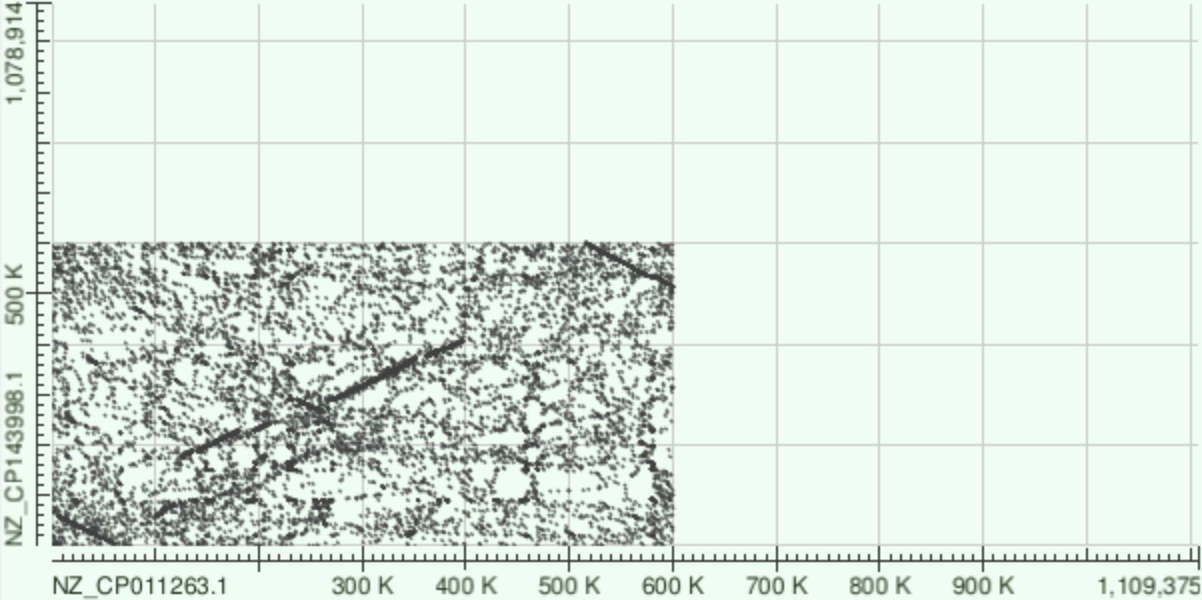
Для TBLASTX использовались фрагменты первых 600000 п.н. каждого генома
tblastx (сравнивает все шесть возможных трансляций нуклеотидной последовательности запроса со всеми шестью возможными трансляциями нуклеотидной последовательности субъекта). Этот инструмент обнаруживает сходство на уровне аминокислот, даже если лежащие в основе ДНК-последовательности разошлись из-за синонимичных мутаций.