Ферменты и метаболические пути
- ЕС код фермента белка KITH_ECOLI
Найдем EC код белка KITH_ECOLI, потом по этому белку определим функцию заданного фермента.
Для этого следует зайти на страницу International Union of Biochemistry and Molecular Biology и далее проследовать по ссылкам - в соответствии с кодом фермента. EC=2.7.1.21:
EC 2
Transferase(отдельный класс ферментов, катализирующих перенос функциональных групп и молекулярных остатков от одной молекулы к другой. Широко распространены в растительных и животных организмах, участвуют в превращениях углеводов, липидов, нуклеиновых и аминокислот.)
Трансферазы
EC 2.7
Transferring phosphorus-containing groups
Перенос групп, содержащих фосфор
EC 2.7.1
Phosphotransferases with an alcohol group as acceptor
Фосфотрансферазы, которые используют в качестве акцептора ОН-группу
EC 2.7.1.21
thymidine kinase
Тимидин киназа
К сожалению, мне не дано было уравнение реакции и графическое изображение. Соответственно, я использовал словесное описание реакции.Название: тимидин киназа Систематическое название: АТФ-тимидин 5`-фосфотрансфераза Реакция: ATP + thymidine = ADP + thymidine 5'-phosphate
- Метаболические пути фермента
В документе UNIPROT с описанием белка KITH_ECOLI найдём имя локуса гена.Это:b1238.
Далее открываем главную страничку БД KEGG и проверим поиск гена по названию данного локуса. В результате поиска гена по названию локуса в базе KEGG среди генов Escherichia coli K12 была найдена запись eco:b1238. В поле Pathways содержатся 2 идентификатора метаболических путей:eco00240 Pyrimidine metabolism Метаболизм пиримидина eco01100 Metabolic pathways Метаболические путиПосмотреть карту метаболизма пиримидина. Красным обозначен фермент тимидин киназа. - Структурные формулы химических соединений
Открываем страницу оглавления KEGG. Находим ссылку на
базу данных химических соединений (KEGG LIGAND).
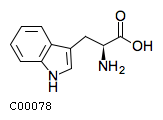
L-триптофан // L-Tryptophan
ID C00078
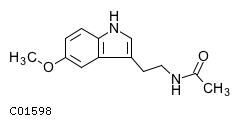
Мелатонин // Melatonin
ID C01598
- Метаболический путь от одного заданного соединения к другому Поиск осуществлялся в БД KEGG Pathway между веществами L-триптофан и Мелатонин. Были найдены две карты, в которых присутствовали оба соединения. Я выбрал карту для более специфических путей:
- Сравните метаболические пути у разных организмов Карта была переведена в режим разных организмов. В некоторых из этих организмов ферменты, необходимые в реакции, оказались незакодированы.
- Сравнение ферментов из далеких организмов
Выбранная цепочка ферментативных реакций: путь: метаболический путь трипотофана цепочка:Триптофан → МелатонинПуть от трипотофана до мелатонина можно посмотреть здесь.
** красным отмечено первое соединение цепочки, желтым - промежуточные, зеленым - последнее соединение цепочки
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | да посмотреть карту |
не все ферменты, необходимые в цепочке, присутствуют. |
| Archaeoglobus fulgidus | нет | ни одного фермента цепочки не закодировано |
| Arabidopsis thaliana | нет | Ни одного фермента цепочки не закодировано, найден только один ген |
| Homo sapiens | нет | Ни одного фермента цепочки не закодировано, найдены гены для 3 ферментов(EC=4.1.1.28;2.3.1.87;2.1.1.4) |
-
Был дан EC-код: 2.1.1.63
1. Найдем с помощью SRS ферменты с ЕС кодом 2.1.1.63 учеловека и археи Archaeoglobus fulgidus.
Опция использования маски (Use wildcards) была снята, чтобы находились только ферменты с кодом 2.1.1.63. Что бы нам жить было легче, воспользуемся тем, что для белков приняты короткие имена. ID белков человека заканчиваются на _HUMAN, белки археи Archaeoglobus fulgidus - на _ARCFU.
Запрос:
([uniprot-ECNumber:2.1.1.63] & ([uniprot-ID:*_human] | [uniprot-ID:*_ARCFU]))
Было найдено 2 находки:MGMT_HUMAN OGT_ARCFU2.В режиме SW_InterProMatches были сравнены доменные организации найденных белков.
| ID | Pfam AC | Изображение |
| MGMT_HUMAN | PF01035 PF02870 |
|
| OGT_ARCFU | PF01035 |
|
Для белков человека в Pfam описаны 2 домена: PF01035 и PF02870 являются метилтрансферазами
Для белка археи показан один домен, такой же, как у человека :PF01035(Methylated-DNA--protein-cysteine methyltransferase)
3.Последовательности фермента с ЕС кодом 2.1.1.63 из археи и человека были вырезаны. Затем для определения процента сходства между ними эти последовательности были выравнены при помощи программы needle. В результате был получен файл.
Identity: 43/246 (17.5%) Similarity: 68/246 (27.6%) Gaps: 138/246 (56.1%)4. Поиск ортологов:
Для белка MGMT_HUMAN название гена MGMT; для OGT_ARCFU - ogt. Соответсвующие id в KEGG hsa:4255 и afu:AF2314.
Ген ортолог для MGMT_HUMAN среди архей:
Ген: mac:MA4322 Организм: Methanosarcina acetivorans Процент идентичности: 0.353 Перекрытие последовательностей: 167
Ген ортолог для OGT_ARCFU среди эукариот:
Ген: ppp:PHYPADRAFT_93804 Организм: Physcomitrella patens subsp. patens Процент идентичности: 0.400 Перекрытие последовательностей: 85Белок в организме человека соответсвует белку в археи с низким процентом идентичности, но, не смотря на это, белки выполняют одинаковые функции, точнее они оба являются метилтрансферазами.
© Мкртчян 2010

