SCOP и CATH
-
Классификация доменов записи PDB 1LZW согласно
SCOP.
Согласно SCOP мой белок состоит из 2 доменов:
N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone from Escherichia coli
Adaptor protein ClpS (YljA) from Escherichia coli
Возьмём каждый домен по отдельности и проанализируем:
N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone
- Расположен в цепи B, занимает цепь полностью
- Класс - All alpha proteins; укладка - Double Clp-N motif; суперсемейство - Double Clp-N motif; семейство - Double Clp-N motif
- В данном суперсемействе содержится 2 семейства
- 2 суперсемейства имеют данную укладку
Adaptor protein ClpS (YljA)
- Расположен в цепи А, занимает всю цепь целиком
- Класс - Alpha and beta proteins (a+b); укладка - ClpS-like; суперсемейство - ClpS-like; семейство - Adaptor protein ClpS (YljA)
- В данном суперсемействе содержится 2 семейства
- 2 суперсемейства имеют данную укладку
Классификация доменов согласно
CATH.
Было найдено 2 домена, опять-таки проанализируем каждый домен по отдельности.
Domain 1lzwA00
- Расположен в цепи А, занимает позиции 1-91
- Классификация по CATH: 3.30.1390.10.3.1.1.2.1
- Класс - Alpha Beta, архитектура - 2-Layer Sandwich, топология - Ribosomal Protein L30; Chain: A
- 3 семейства содержится в данном суперсемействе
- 3 суперсемейства имеет данную топологию
Domain 1lzwB00
- Расположен в цепи B, занимает позиции 92-237
- Классификация по CATH: 1.10.1780.10.1.1.1.1.13
- Класс - Mainly Alpha, архитектура - Orthogonal Bundle, топология - Double Clp-N motif, суперсемейство - Double Clp-N motif
- 2 семейства содержится в данном суперсемействе
- 1 суперсемейство имеет данную топологию
Различия
между CATH и SCOP
Количество доменов в CATH и SCOP одинаковое, семейства сопадают (Double Clp-N motif). SCOP определил класс первого домена All alpha proteins, а CATH - Mainly Alpha. Это объясняется тем, что в CATH есть небольшое количество классов, а в SCOP их 11. Вторые домены по классификации совпадают.
Небольшая путаница возникла с координатами и расположением доменов. Вот, например, SCOP определил певый домен как полностью всю цепь, а CATH только с 1-91 и 92-237.
Все это может быть связано с разным количеством уровней (в СATH их 9, а в SCOP 6), это говорит о разной степени подробности каждого из уровней.
Выравнивание
Для примера возьмем цепь А 1xi3 и 2tps. Они имеют одинаковую укладку но разное суперсемейство. Выравнивание было проведено с помощью команды super (rmsd=3.542):
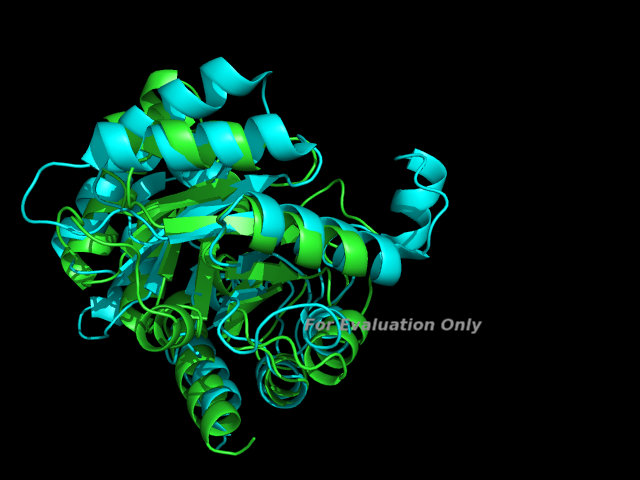
Видно, что структуры совпадают друг с другом по топологии, но я ещё использовал команду super, т.к с его помощью получается наилучшее выравнивание.
Да, видно, что структуры как-то наложились, но назвать это наложение удачным я бы не осмелился.
