Краткое описание структуры в файле 1f7v.pdb
В файле приведены координаты атомов следующих молекул:1) Аргининовая тРНК (идентификатор цепи В)
2) Аргинил -тРНК синтетаза (идентификатор цепи А)
Для исследования была выбрана цепь B, представляющая аргининовую тРНК со следующей последовательностью:
[901] 5' - PSU U C C U C G U 1MG 2MG C C C A A DHU G G DHU C A C G G C M2G PSU C U G G C U I C G A
A C C A G A A G A DHU U 5MC C A G G 5MU PSU C A 1MA G U C C U G G C G G G G A A G C C A - 3' [973], где 901 и 973 - номера первого и последнего нуклеотида.
Первый нуклеотид тРНК имеет номер 901, а последний - 973. Значит, длина молекулы равна 76 нуклеотидов.
Это нормально, ведь тРНК сама по себе очень маленькая нуклеиновая кислота. Обычно её размер не превышает 85 нуклеотидов.
Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (1F7V_NIC_old.out), была предсказана возможная вторичная структура. Рассмотрим следующую таблицу:
Strand I Strand II Helix
1 (0.043) B:.901_:[PSU]P-*---A[..A]:.972_:B (0.002) |
2 (0.002) B:.902_:[..U]U-----A[..A]:.971_:B (0.004) |
3 (0.004) B:.903_:[..C]C-----G[..G]:.970_:B (0.004) |
4 (0.005) B:.904_:[..C]C-----G[..G]:.969_:B (0.004) |
5 (0.003) B:.905_:[..U]U-*---G[..G]:.968_:B (0.008) |
6 (0.003) B:.906_:[..C]C-----G[..G]:.967_:B (0.011) |
7 (0.004) B:.907_:[..G]Gx----C[..C]:.966_:B (0.004) |
8 (0.013) B:.949_:[5MC]c-----G[..G]:.965_:B (0.005) |
9 (0.004) B:.950_:[..C]C-----G[..G]:.964_:B (0.007) |
10 (0.006) B:.951_:[..A]A-----U[..U]:.963_:B (0.005) |
11 (0.010) B:.952_:[..G]G-----C[..C]:.962_:B (0.006) |
12 (0.010) B:.953_:[..G]G----xC[..C]:.961_:B (0.004) |
13 (0.009) B:.954_:[5MU]u-**-xa[1MA]:.958_:B (0.009) |
14 (0.044) B:.955_:[PSU]Px**+xG[..G]:.917_:B (0.009) x
15 (0.008) B:.939_:[..C]C-----G[..G]:.931_:B (0.010) |
16 (0.003) B:.940_:[..C]C-----G[..G]:.930_:B (0.007) |
17 (0.006) B:.941_:[..A]A-----U[..U]:.929_:B (0.005) |
18 (0.010) B:.942_:[..G]G-----C[..C]:.928_:B (0.004) |
19 (0.003) B:.943_:[..A]A-*---P[PSU]:.927_:B (0.044) |
20 (0.008) B:.944_:[..A]Ax*---g[M2G]:.926_:B (0.013) |
21 (0.007) B:.910_:[2MG]g-----C[..C]:.925_:B (0.008) |
22 (0.003) B:.911_:[..C]C-----G[..G]:.924_:B (0.010) |
23 (0.004) B:.912_:[..C]C-----G[..G]:.923_:B (0.012) |
24 (0.010) B:.913_:[..C]C-*--xC[..C]:.922_:B (0.005) |
25 (0.003) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.004) |
26 (0.005) B:.915_:[..A]Ax**+xU[..U]:.948_:B (0.006) x
27 (0.010) B:.918_:[..G]G-----C[..C]:.956_:B (0.003) +
Для того, чтобы разобраться где какой стебель, нужно посмотреть на картинку:
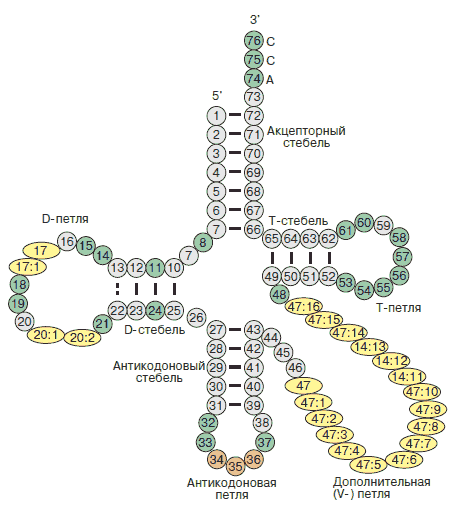 (Картинка была взята из статьи О.О.Фапоровой)
(Картинка была взята из статьи О.О.Фапоровой)
Акцепторный стебель : 901-907, 966-972.
D-стебель : 910-912, 923-925.
Антикодоновый стебель: 927-931, 939-943.
Т-стебель: 949-953, 961-965.
В структуре есть и вариабельная петля. Это 44-48.
Создаём скрипт для получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым.
| Рис.1. Вторичная структура тРНК из SACCHAROMYCES CEREVISIAE | Скрипт для получения изображения |
background white define steb_ac 901-907, 966-972 define steb_t 949-953, 961-965 define steb_d 910-912, 923-925 define strb_an 927-931, 939-943 select all cpk off wireframe off backbone 100 select steb_ac color red select steb_t color green select steb_d color blue select steb_an color orange select not (steb_ac, steb_t, steb_d, steb_an) color grey Данный скрипт служит для получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. Теперь разберёмся с антикодоном. Для кодона аргининовой кислоты антикодоном являются 6 триплетов:ACG, GCG, UCG, CCG, UCU и UCC. Найдём UCG. Это атомы 931, 932, 933. |
| Рис.2. Антикодон на антикодоновой петле | Скрипт для получения изображения |
background white define steb_ac 901-907, 966-972 define steb_t 949-953, 961-965 define steb_d 910-912, 923-925 define strb_an 927-931, 939-943 select all cpk off wireframe off backbone 100 select steb_ac color red select steb_t color green select steb_d color blue select steb_an color orange select 931, 932, 933 color cpk select not (steb_ac, steb_t, steb_d, steb_an) color grey |
| Рис.3. Неканонические взаимодействия | Скрипт для получения изображения |
| background white restrict 908, 914 |
1.G 945-2mG 910 (2N-метилгуанозин)
2.G 917-PSU 955 (псевдоуридин)
3.5mU 954-1mA 958 (6-Гидро-1-метиладенозин)
Исследование третичной структуры
В РНК спирали один из элементов структуры, и основания, не входящие в них, могут образовывать стекинг-связи с отдаленными основаниями, сворачивая цепь. C помощью программы analyze, мы смогли обнаружить 26 стекинг-взаимодействий. Вы можете сами в этом убедиться :
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 PU/AA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.74( 2.50) 2.74( 2.50)
2 UC/GA 0.12( 0.00) 0.00( 0.00) 0.00( 0.00) 4.08( 2.87) 4.20( 2.87)
3 CC/GG 0.00( 0.00) 0.00( 0.00) 0.51( 0.00) 3.94( 2.54) 4.44( 2.54)
4 CU/GG 2.28( 0.61) 0.00( 0.00) 0.05( 0.00) 4.64( 3.14) 6.97( 3.76)
5 UC/GG 0.38( 0.00) 0.00( 0.00) 0.23( 0.00) 1.84( 0.57) 2.46( 0.57)
6 CG/CG 0.20( 0.00) 0.00( 0.00) 4.02( 1.35) 0.26( 0.00) 4.48( 1.35)
7 Gc/GC 5.97( 1.41) 0.00( 0.00) 0.00( 0.00) 7.08( 4.30) 13.05( 5.71)
8 cC/GG 0.00( 0.00) 0.00( 0.00) 0.97( 0.00) 2.98( 1.45) 3.95( 1.45)
9 CA/UG 0.00( 0.00) 0.00( 0.00) 3.10( 1.07) 0.49( 0.06) 3.58( 1.13)
10 AG/CU 5.03( 2.85) 0.00( 0.00) 0.03( 0.00) 0.08( 0.00) 5.13( 2.85)
11 GG/CC 3.98( 2.42) 0.00( 0.00) 0.81( 0.00) 0.00( 0.00) 4.79( 2.42)
12 Gu/aC 7.08( 1.99) 0.00( 0.00) 0.00( 0.00) 4.95( 1.78) 12.03( 3.77)
13 uP/Ga 5.41( 1.91) 0.00( 0.00) 0.00( 0.00) 7.84( 2.58) 13.25( 4.49)
14 PC/GG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 CC/GG 0.00( 0.00) 0.00( 0.00) 0.99( 0.00) 3.13( 1.59) 4.13( 1.59)
16 CA/UG 0.00( 0.00) 0.00( 0.00) 3.98( 1.80) 0.05( 0.00) 4.03( 1.80)
17 AG/CU 1.98( 1.88) 0.00( 0.00) 0.83( 0.00) 0.00( 0.00) 2.81( 1.88)
18 GA/PC 3.12( 1.62) 0.00( 0.00) 0.08( 0.01) 0.00( 0.00) 3.20( 1.63)
19 AA/gP 3.79( 2.38) 0.00( 0.00) 0.00( 0.00) 3.93( 1.35) 7.72( 3.74)
20 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.23( 0.05) 0.28( 0.08) 1.52( 0.13)
21 gC/GC 3.31( 1.06) 0.00( 0.00) 0.00( 0.00) 5.48( 2.49) 8.79( 3.55)
22 CC/GG 0.00( 0.00) 0.00( 0.00) 0.92( 0.00) 3.51( 1.97) 4.42( 1.97)
23 CC/CG 2.84( 1.12) 0.00( 0.00) 0.00( 0.00) 1.73( 0.88) 4.58( 2.00)
24 CA/UC 0.00( 0.00) 2.49( 0.10) 4.49( 3.16) 0.00( 0.00) 6.98( 3.26)
25 AA/UU 6.31( 4.27) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 6.31( 4.27)
26 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
С помощью этой таблицы можно найти данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований.
Стекинг-взаимодествие между основаниями акцепторного (G907-C966) и T-стебля (C65-G49):
7 (0.004) B:.907_:[..G]Gx----C[..C]:.966_:B (0.004) | 8 (0.013) B:.949_:[5MC]c-----G[..G]:.965_:B (0.005) |Смотрим на табличку и ищим это взаимодействие :
7 Gc/GC 5.97( 1.41) 0.00( 0.00) 0.00( 0.00) 7.08( 4.30) 13.05( 5.71)
Воспользуемся командой stack2img: 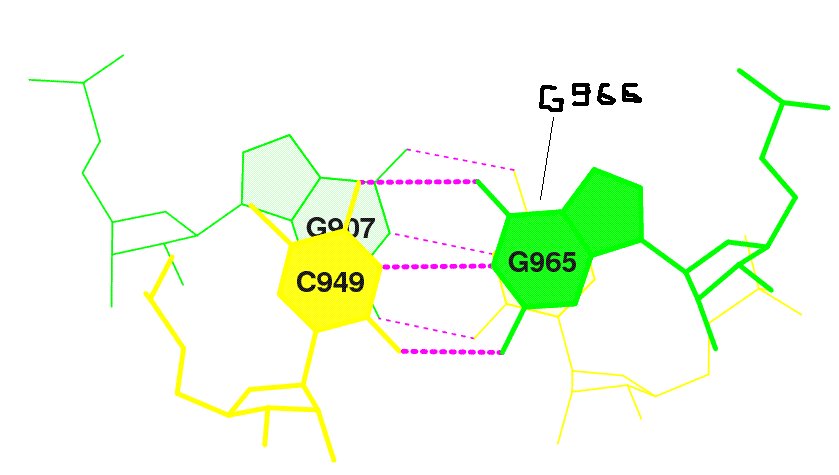
Дополнительные водородные связи между основаниями D- и Т-петель
Есть 2 водородные связи, стабилизирующие структуру :14 (0.044) B:.955_:[PSU]Px**+xG[..G]:.917_:B (0.009) x 27 (0.010) B:.918_:[..G]G-----C[..C]:.956_:B (0.003) +
Предсказание вторичной структуры тРНК
Алгоритм Зукера используется для предсказания вторичной структуры тРНК. Данный алгоритм осуществляется программой mfold. В первую очередь сохраняем последовательность тРНК в файл.
| Рис.1. Предсказанная вторичная структура тРНК из SACCHAROMYCES CEREVISIAE | Текст |
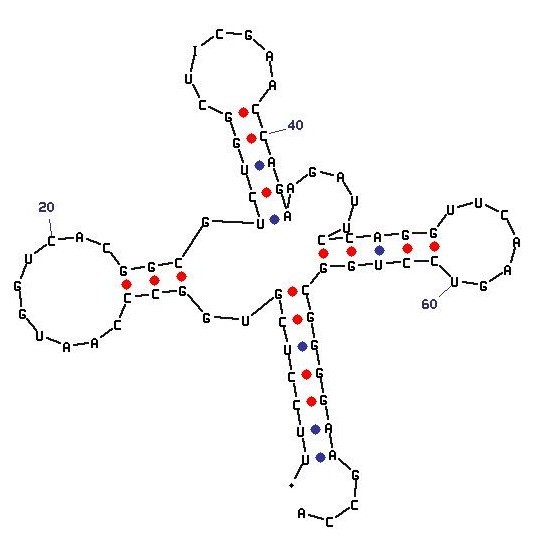 |
В файле 1f7v.fasta нужно было поменять все модифицированные основания на нормальные, чтобы программа более точно предсказала структуру.
Но у меня ничего не получалось. Всё из-за инозина (I), близкого родственника гуанину. Его трогать не надо было. Тогда при P=10, у меня получилась картинка слева. Эта картинка полностью соответствует построенной тРНК.Можно разлечить каждую петлю, даже вариабельную.
|
После использования программы analyze, можно сказать что спирали тРНК похожи на А-форму ДНК. Для быстрого поиска инвертированных последовательностей была использована программа einverted. Рекомендуемые параметры для einverted:
Gap penalty = 3
Minimum score threshold = 5
Match score = 3
Mismatch score = -3
Описание программы einverted можно прочитать, выполнив tfm einverted. Список параметров получаем, выполнив einverted -help
tRNA: Score 15: 5/5 (100%) matches, 0 gaps
27 tctgg 31
|||||
43 agacc 39
tRNA: Score 15: 5/5 (100%) matches, 0 gaps
49 ccagg 53
|||||
65 ggtcc 61| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 901-907 3' 5' 966-972 3' Всего 7 пар |
Предсказано 7 пар из 7 реальных |
| D-стебель | 5' 910-913 3' 5' 922-925 3' Всего 4 пары |
Предсказано 4 пары из 4 реальных |
| T-стебель | 5' 949-953 3' 5' 961-965 3' Всего 5 пар |
Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5' 939-943 3' 5' 927-931 3' Всего 5 пар |
Предсказано 5 пар из 5 реальных |
| Общее число канонических пар нуклеотидов | 21 | 21 |