На данной странице представлен практикум по поиску и визуализации последовательности Шайна-Дальгарно в геноме бактерии Bacillus paralicheniformis штамма Bac84 с помощью сервисов MEME и FIMO. Скрипты были написаны в соавторстве с Литвиновой Анастасией.
1) Описание SD и выбор бактерии:
Последовательность Шайна-Дальгарно (SD) – сайт связывания рибосомы на молекулах мРНК прокариот. Обычно она распологается на расстоянии ~10 нуклеотидов от стартового кодона ATG и комплементарна 3'-концевому фрагменту 16s рРНК. Консенсусом для большинства прокариот является последовательность AGGAGG. В данном практикуме проверяется соответствие данного консенсуса для выбранной бактерии.
Была выбрана бактерия Bacillus paralicheniformis штамма Bac84 (GCF_002993925.1), для которой мной в первом семестре был написан обзор. Данная бактерия как и другие представители данного рода – вид грамположительных факультативно анаэробных бактерий, способных образовывать эндоспоры. Она впервые была выделена в 2015 году из твенджана.
2) Получение последовательностей:
Для получения последовательностей, в которых будет искаться SD, со страницы NCBI, посвященной геному B. paralicheniformis, были скачены файл с геномом и файл с таблицей локальных особенностей данной бактерии. Данная таблица была преобразована в более удобный формат при помощи скрипта. Далее с помощью скрипта случайным образом из полученной таблицы были выбраны гены, длина которых больше 500 нуклеотидов и их продукт не являются hypothetical protein. С помощью скрипта по координатам полученных "хороших" генов были вырезаны последовательности длиной 28 нуклеотидов, включающих стартовый ATG и 25 upstream нуклеотидов, в которых и будет искаться SD. Далее полученный fasta файл был вручную отредактирован, чтобы избавиться от плохо-аннотированных генов, а также в него был добавлена 3'-концевая последовательность 16s рРНК. Суммарно получилось 283 последовательности (файл), которые будут использоваться как обучающая выборка. Для получения тестовой выборки был выполнен скрипт по всей таблице локальных особенностей, который также вырезает последовательности длины 28 нуклеотидов (3 ATG и 25 upstream нуклеотидов). Далее при помощи скрипта полученные последовательности были отфильтрованы по наличию трех последних ATG (понимаю, что это слишком суровая фильтрация, но вручную 4000 генов отсмотреть не получилось бы, поэтому была выбрана именно такая метрика качества). Cуммарно получилось 1624 последовательности (файл), они будут использоваться в качестве тестовой выборки.
3) MEME и FIMO:
Полученная учебная выборка была загружена в веб-сервис MEME с параметрами (Рис. 1):
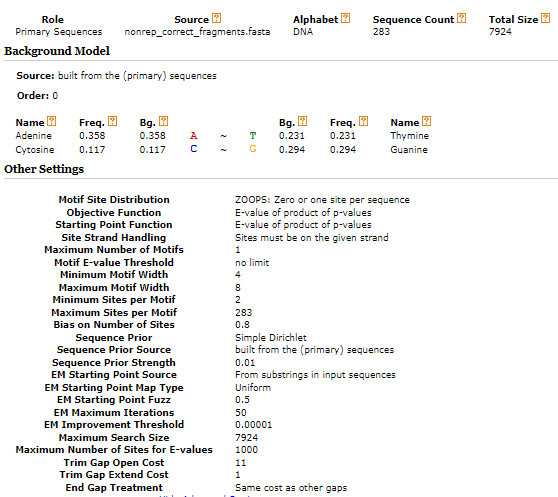
Результаты работы МЕМЕ:
При выборе параметров при запуске МЕМЕ были выставлены границы длины мотива – 6-8 нуклеотидов, поэтому найденный мотив имеет длину 8 нуклеотидов. Возможно два аденина перед консенсуным AGGAGGG также важны при взаимодействии с рРНК. Это подтверждается 3'-концевой последовательностью 16s rРНК – 3'-TTTCCTCC-5' (записано с 3'-конца для удобства прослеживания сходства). Logo найденного мотива на рисунке 2.
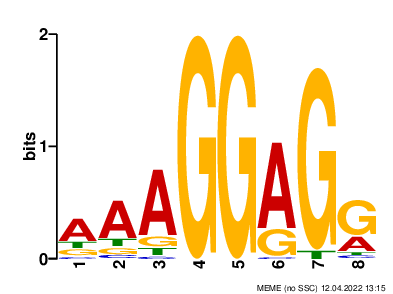
Найденный мотив в формате meme и тестовая выборка были загружены в веб-сервис FIMO с рекомендованными настройками и ограничением p-value 0,001. В результате в тестовой выборке было найдено 926 мотивов в изучаемых генах с p-value меньше чем 0,001. Полная выдача FIMO по ссылке.
По итогам проделанной работы можно сделать вывод о соответствии мотива SD B. paralicheniformis консенсу AGGAGG, свойственному большенству прокариот. Кроме этого найденный мотив был расширен до 8 нуклеотидов – AAAGGAGG, что соотвествует 3'-концевой последовательности 16s рРНК. Также с порогом p-value в 0,001 были найдены 926 генов, соответствующих данному мотиву.