На данной странице представлен практикум по изучению представленности сайтов эндонуклеаз рестрикции в бактериальном геноме. Скрипты были написаны в соавторстве с Литвиновой Анастасией.
1) Анализ представленности сайтов рестриктаз:
Для анализа была выбрана бактерия Bacillus subtilis 168 (AL009126), которая является одним из бактериальных модельных организмов. Наличие эндонуклеаз рестрикции II типа было проверено в базе данных REBASE.
Со страницы NCBI был скачен геном данной бактерии. Также со страницы данной бактерии в базе данных REBASE был получен сайт рестрикции, по которому разрезают рестриктазы (всего их 3, но у всех один сайт) данной бактерии – CTCGAG. С помощью скрипта из таблицы был сформирован список всех известных сайтов рестрикци длины большей, чем 2 нуклеотида. Далее на сервере кодомо с помощью команды:
была оценена представленность полученных сайтов рестрикции в геноме B. subtilis. После этого с помощью скрипта из полученной tsv таблицы были отфильтрованы сайты со значением представленности меньшим 80% (рекомендованный порог). Таких сайтов получилось 7 штук, файл c их последовательностями. Как и ожидалось сайт CTCGAG, характерный для рестриктаз B. subtilis оказался недопредставлен.
Далее производился поиск в геноме B. subtilis гомологов рестриктаз из других бактерий, которые режут по недопредставленным сайтам. С помощью скрипта из файла были получены идентификаторы рестриктаз, которые разрезают по недопредставленным сайтам. Всего таких рестриктаз оказалось 2169. Файл с их идентификаторами. При помощи скрипта были получены последовательности данных рестриктаз. С помощью команд на сервере кодомо:
tblastn -query forBlast.fasta -db bacsu_genome.fasta -evalue 0.001 -out result.txt
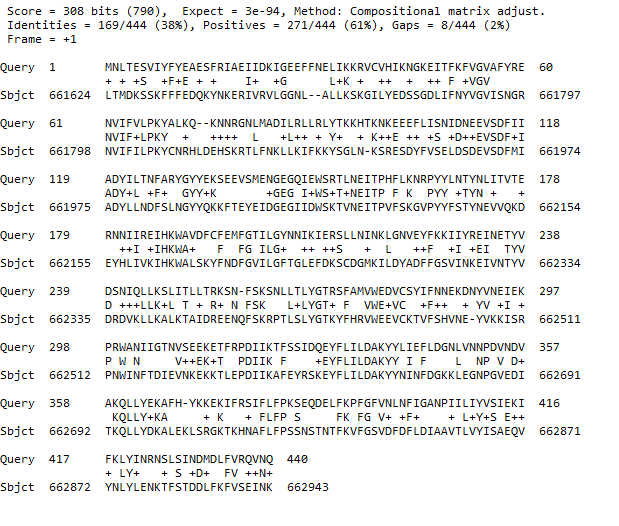
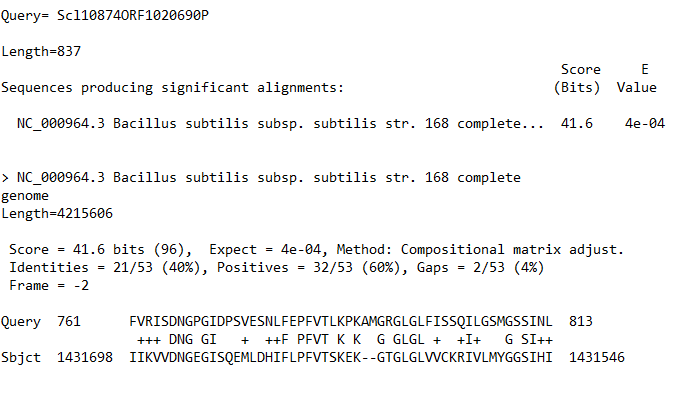
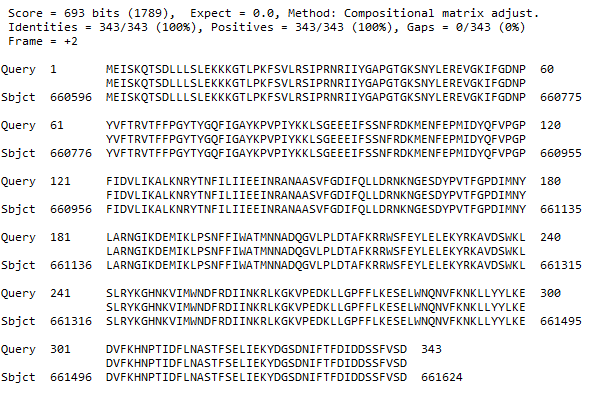
был произведен BLAST+ поиск гомологов полученных рестриктаз алгоритмом tblastn в геноме B. subtilis c порогом e-value 0.001. Файл выдачи. Проанализировать полученный файл адекватным образом мне не видится возможным, т.к. текстовая выдача составляет 48000 строк, отмечу лишь, что возможные гомологи в геноме есть (Рис. 1,2). Также были найдены последовательности в близкородственных бактериях, полностью идентичные запросам (Рис.3)
2) PSI-BLAST:
Мной был выбран белок Ribosome-associated inhibitor A (RaiA) (P0AD49), участвющий в регуляции трансляции, препятствуя диссоциации рибосомы. Организм – Esherihia coli.
Таблица с результатом. Порог E-value был выбран 0,005 по умолчанию. После первой итерации было найдено 22 белка с E-value ниже 0,005 и один белок с E-value слегка выше выбранного порога. После построения PSSM по найденным последовательностям и выполнения следующей итерации, алгоритмом были найдены 27 белков. Среди 5 добавленных была и та, что в первой итерации была с E-value слегка выше порогового. Последующие итерации не привели к изменению списка находок, что говорит о возможном факте находки всех белков из семейства, к которому относится белок RaiA, и высоком родстве белков внутри данного семейства.